http://www.biocloudservice.com/366/366.php
1、软件内容及流程:
共包含1个R语言脚本,脚本介绍如下:
singcell.R
程序名称:
singcell.R
功能描述:
该软件是云生信团队专为单细胞测序分析设计打造,可实现一站式单细胞测序数据的下游分析,包括:
- 数据质控过滤,
- 数据标准化,
- PCA主成分分析,
- tSNE降维聚类分析,
- 查找细胞群maker基因,
- 自动化注释细胞类型,
- 细胞轨迹分析
等等单细胞数据分析的科学方法。并且会自动生成分析的结果图。对用户操作十分友好,无需任何编码,即可快速拿到高质量的分析结果。
使用方法:
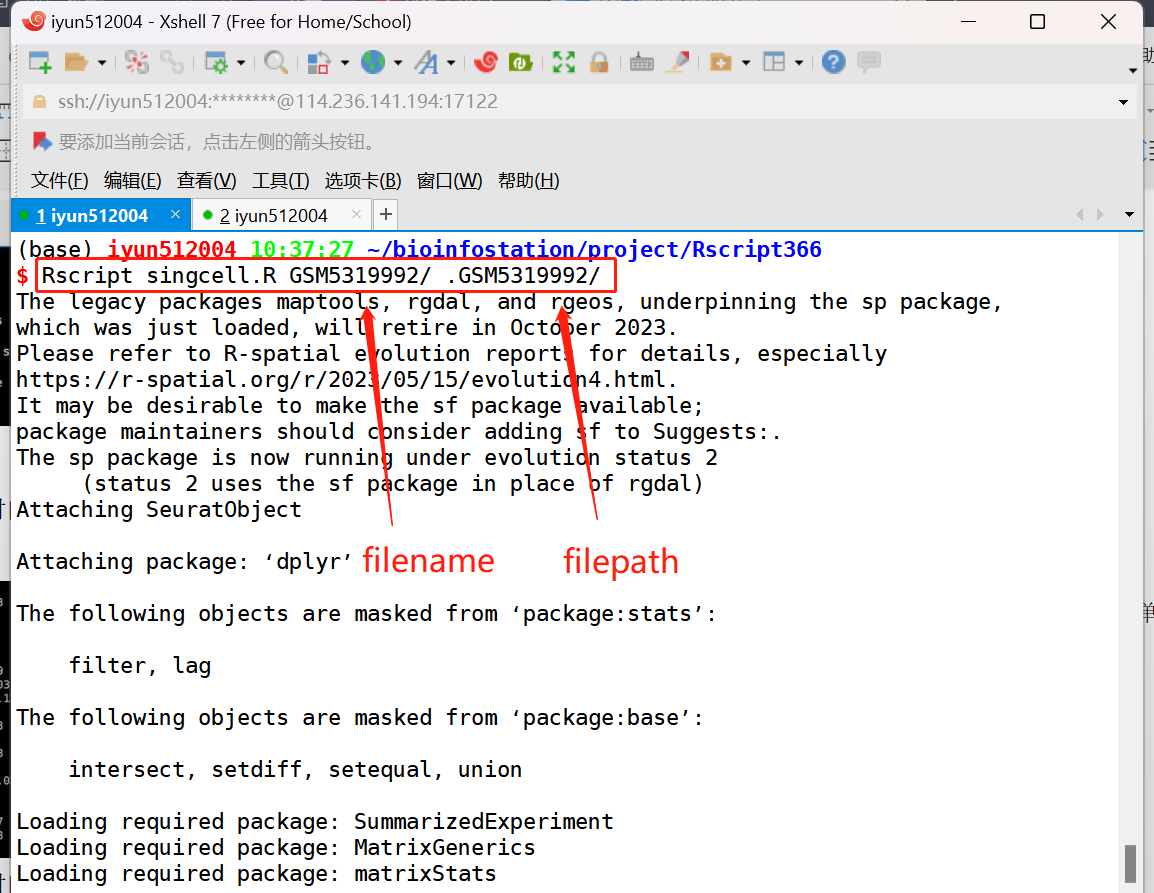
Rscript singcell.R -filename -filepath
参数说明:
USAGE:
singcell.R -filename=<file> -filepath=<path>
PARAMETERS:
-filename The name of the single-cell data folder that needs to be analyzed. Subfolders under the folder may include multiple samples. Under the subfolders should include barcodes,feature, feature_barcode_matrix three single-cell sequencing upstream processed files.
-filepath The path to the single-cell data folder that needs to be analyzed.
- 打开命令行界面,输入“Rscript singcell.R filename filepath”这里,必须输入参数是-filename,-filepath;filename表示需要进行分析的单细胞数据文件夹名称。文件夹下可以包括多个样本的子文件夹。子文件夹下应包括barcodes,feature,feature_barcode_matrix三个单细胞测序上游处理后的文件;filepath表示需要进行分析的单细胞数据文件夹的路径。
3、完成参数提交后,按下回车键,整个程序即正式开始进入执行。程序执行完毕后,界面会显示“Program execution is completed”结束语。
操作界面截图:
- 程序运行命令
- 输入文件界面展示截图示例
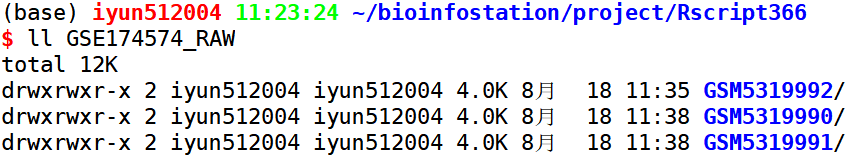
示例输入的文件GSE174574_RAW/ 下有三个样本
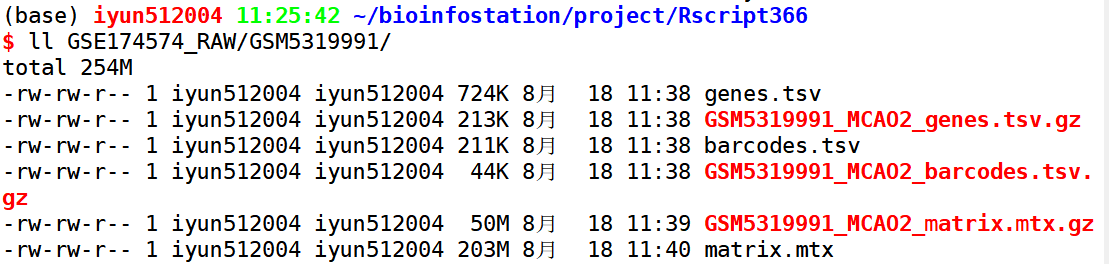
样本文件夹下,应包括barcodes, feature, feature_barcode_matrix三个单细胞测序上游处理后的文件。
- 程序运行界面截图
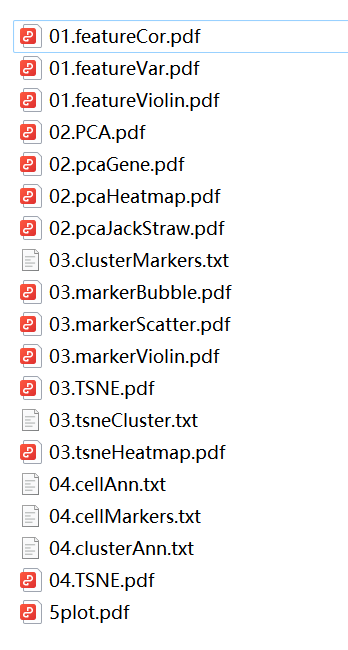
- 程序运行结果文件展示

输出文件:
结果文件分为五个部分,分别是:
- 基因特征
- PCA分析
- Maker基因识别
- tSNE聚类
- 细胞轨迹图
输出的图片将以pdf格式保存。分析数据以txt格式保存。
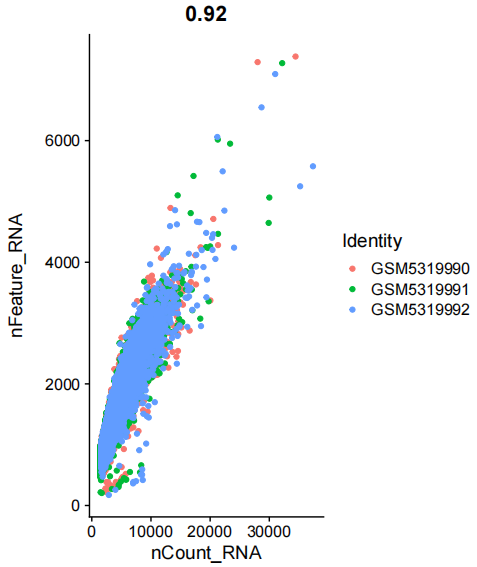
- 测序深度相关性图
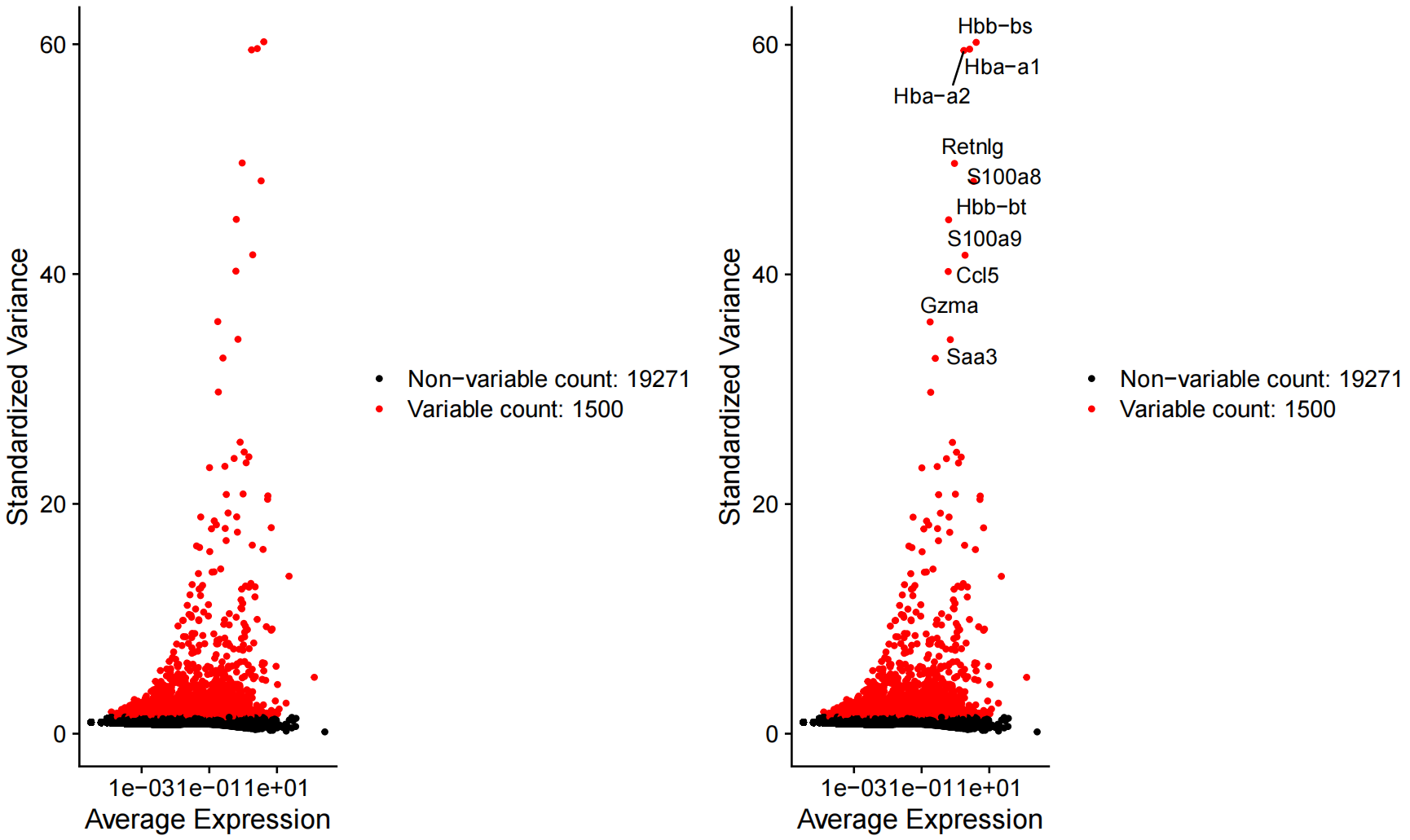
- 特征方差图
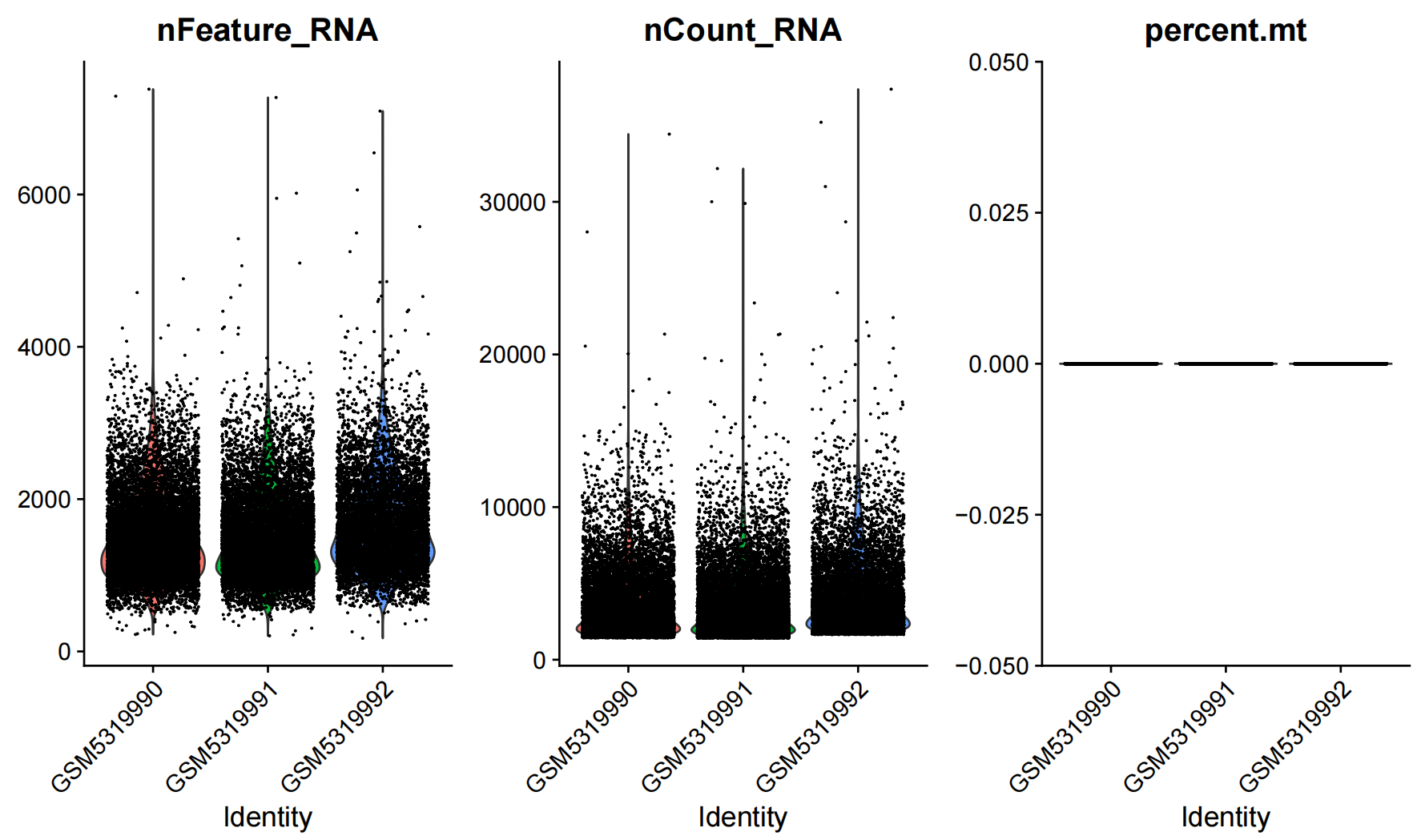
- 单细胞分布小提琴图
- PCA分析热图
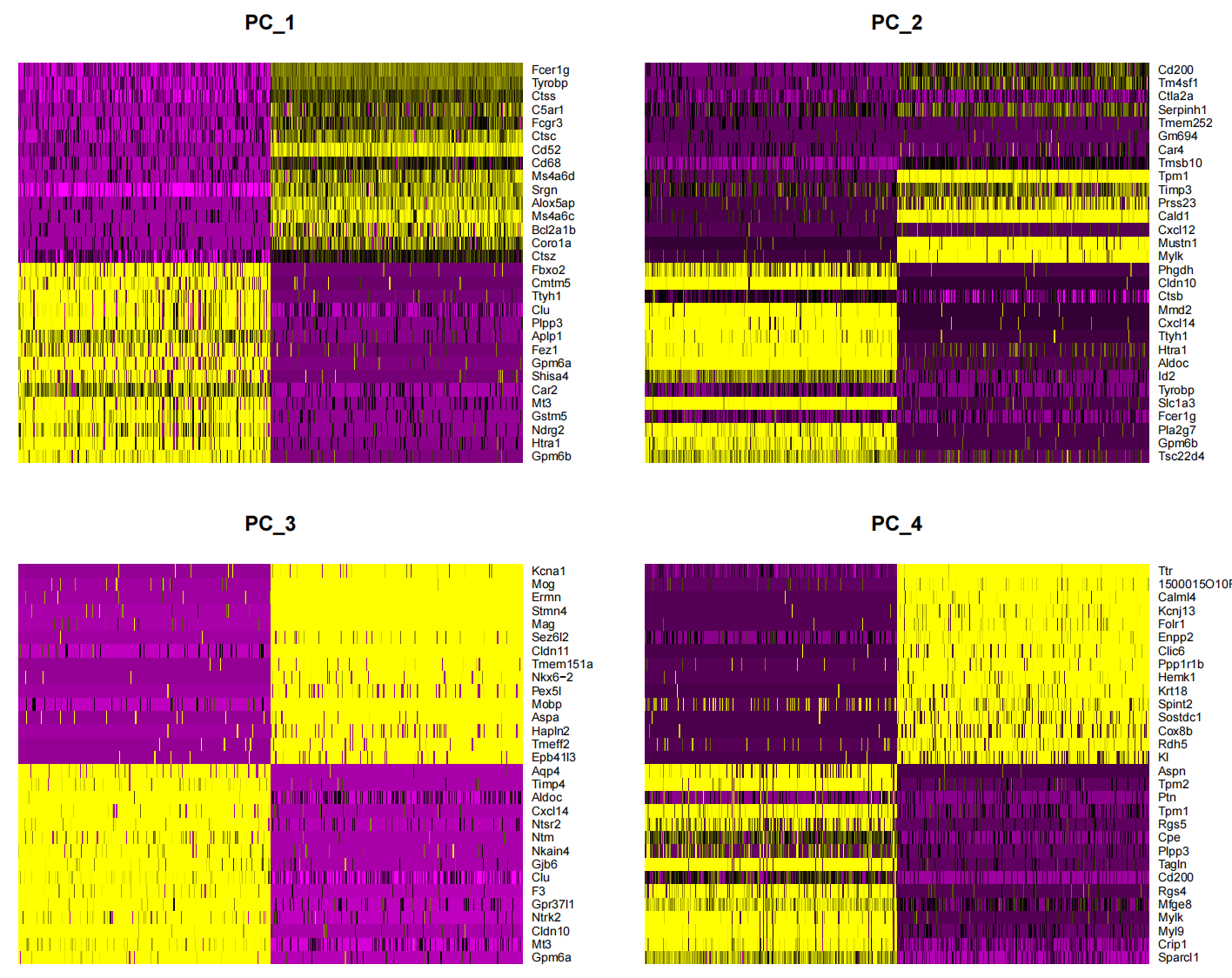
- FeaturePlot

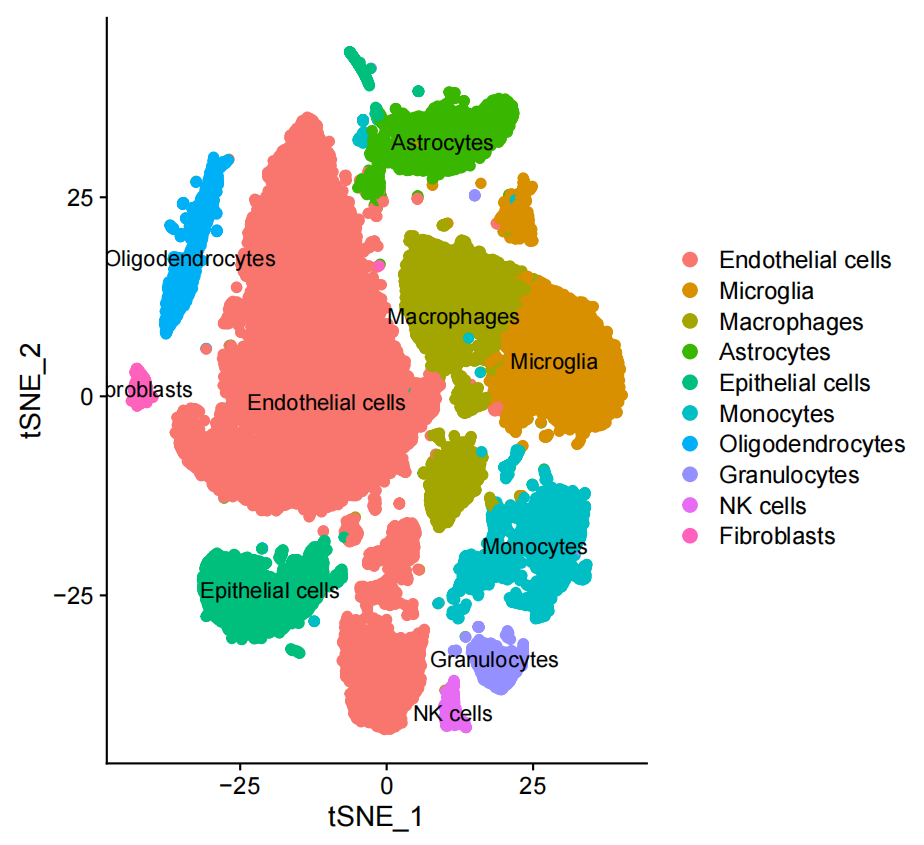
- tSNE投影图

- 识别细胞类型
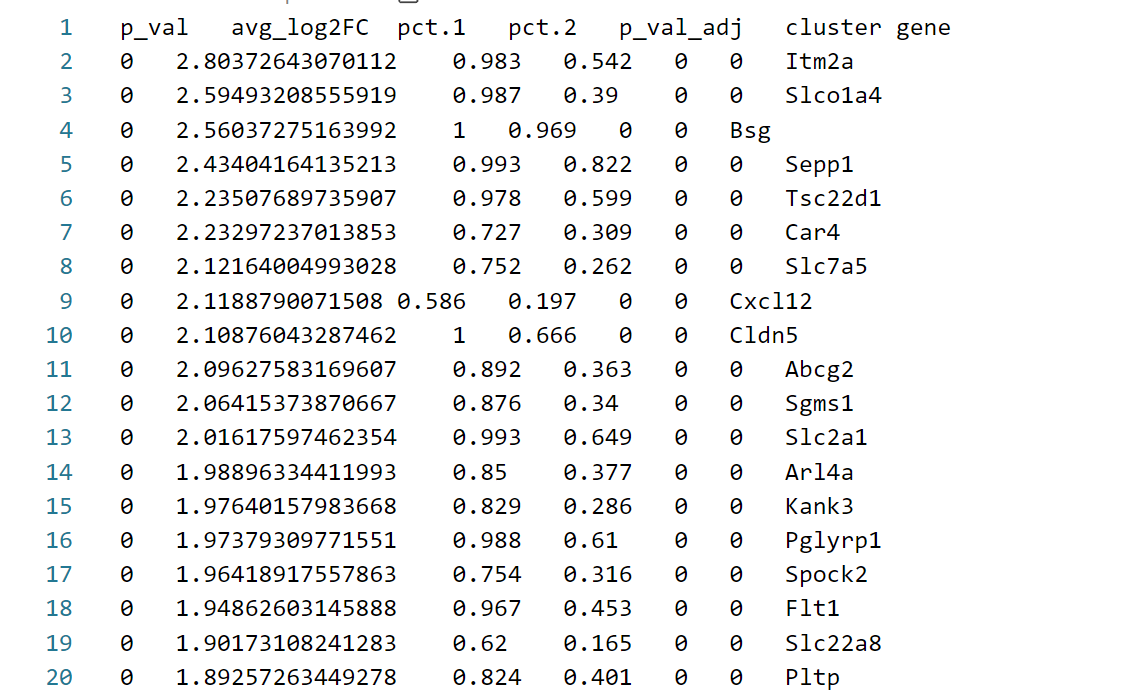
- 03.clusterMarkers.txt
- 04.cellAnn.txt

等文件
云平台下操作截图:
(1)运行示例数据
可得到示例数据的结果

点击下载全部文件,即可下载全部的分析结果文件。
- 上传数据

点击上传数据
即可进入文件上传的页面。选择我们要分析的数据
上传好后,点击运行自有数据,即可开始分析。
程序运行完成后,将会输出以下结果。