上一节,小果向大家介绍了ClusterProfiler进行GO富集分析的方法,ClusterProfiler包还可以进行基因通路的富集。KEGG(Kyoto Encyclopedia of Genes and Genomes)是一个广泛使用的基因通路数据库,提供了关于生物系统功能和调控的丰富信息。在这一节中,我们将介绍如何使用R语言中的clusterProfiler包进行KEGG富集分析,以揭示给定基因集合中富集的KEGG通路。使用clusterProfiler进行KEGG(Kyoto Encyclopedia of Genes and Genomes)分析的步骤与GO分析类似,下面接着跟小果一起来学习吧!
- 安装加载包
在开始之前,确保已在R环境中安装了clusterProfiler包。可以使用以下代码进行安装和加载:
代码:
if (!require(“BiocManager”, quietly = TRUE))
install.packages(“BiocManager”)
BiocManager::install(“clusterProfiler”)
library(clusterProfiler)
- 准备输入数据
为了进行KEGG富集分析,我们需要准备一个基因列表,该列表包含我们感兴趣的基因。基因列表可以是基因符号、Ensembl ID或其他常见的基因识别符。例如,我们准备了一个名为”gene.txt”的文本文件,其中包含Ensembl符号列表,每行一个基因。
图1 基因列表文件内容示例
代码:
genes <- read.delim(‘gene.txt’, header = TRUE, stringsAsFactors = FALSE)[[1]]
- 进行基因ID转换
在进行KEGG富集分析之前,我们需要对基因列表进行基因注释,将基因识别符映射到ENTREZID信息。在clusterProfiler中,可以使用bitr函数进行基因注释。
代码:
# 将ENSEMBL ID映射到ENTREZID(以人类为例)
gene2kegg <- bitr(genes, fromType = “ENSEMBL”, toType = “ENTREZID”, OrgDb = org.Hs.eg.db)
- 进行KEGG富集分析
我们可以使用enrichKEGG函数对注释后的基因进行KEGG富集分析。该函数根据基因在特定KEGG通路中的富集程度计算富集分数和调整的p值。您可以选择不同的统计方法和参数设置。相比上述GO富集,clusterProfiler的KEGG富集分析可以直接链接到KEGG的在线数据库获取已有物种的注释。
代码:
getOption(“clusterProfiler.download.method”)
R.utils::setOption(“clusterProfiler.download.method”,”auto”)
#KEGG 富集分析
#每次打开 R 计算时,它会自动连接 KEGG 官网获得最近的物种注释信息
kegg <- enrichKEGG(
gene = gene2kegg$ENTREZID, #基因列表文件中的基因名称
keyType = ‘kegg’, #KEGG 富集
organism = “hsa”, #例如,hsa 代表人类,其它物种更改这行即可
pAdjustMethod = “BH”,
pvalueCutoff = 0.1, #指定 p 值阈值(可指定 1 以输出全部)
qvalueCutoff = 0.2) #指定 q 值阈值(可指定 1 以输出全部)
#输出结果
write.table(kegg, ‘kegg.txt’, sep = ‘\t’, quote = FALSE, row.names = FALSE)
在上述代码中,我们指定了注释后的基因(gene = gene2kegg$ENTREZID),选择了人类(organism = “hsa”)作为研究对象,并设置了p值和q值的阈值(pvalueCutoff = 0.05, qvalueCutoff = 0.2)。可以通过KEGG官网(KEGG Organisms: Complete Genomes)查看KEGG数据库中提供的物种注释列表。
- 结果解释和可视化
经过KEGG富集分析后,我们可以对结果进行解释和可视化,以更好地理解基因集合中富集的KEGG通路。可以使用summary函数和barplot、dotplot函数对结果进行摘要和可视化。
summary函数:summary(kegg)

图2 查看富集分析结果摘要
通过summary函数,我们可以查看KEGG富集分析的结果摘要信息,包括富集的KEGG通路、调整的p值、基因数等。这些信息可以帮助我们理解基因集合在不同通路中的富集情况。
barplot函数:barplot(kegg)
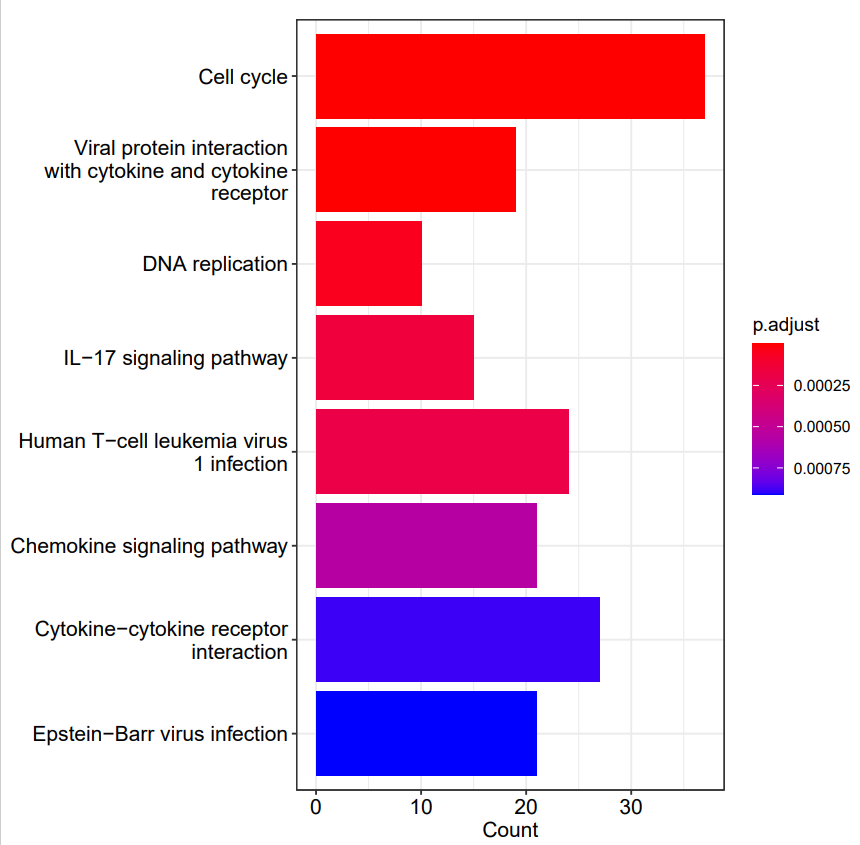
图3 富集柱形图
dotplot函数:dotplot(kegg)
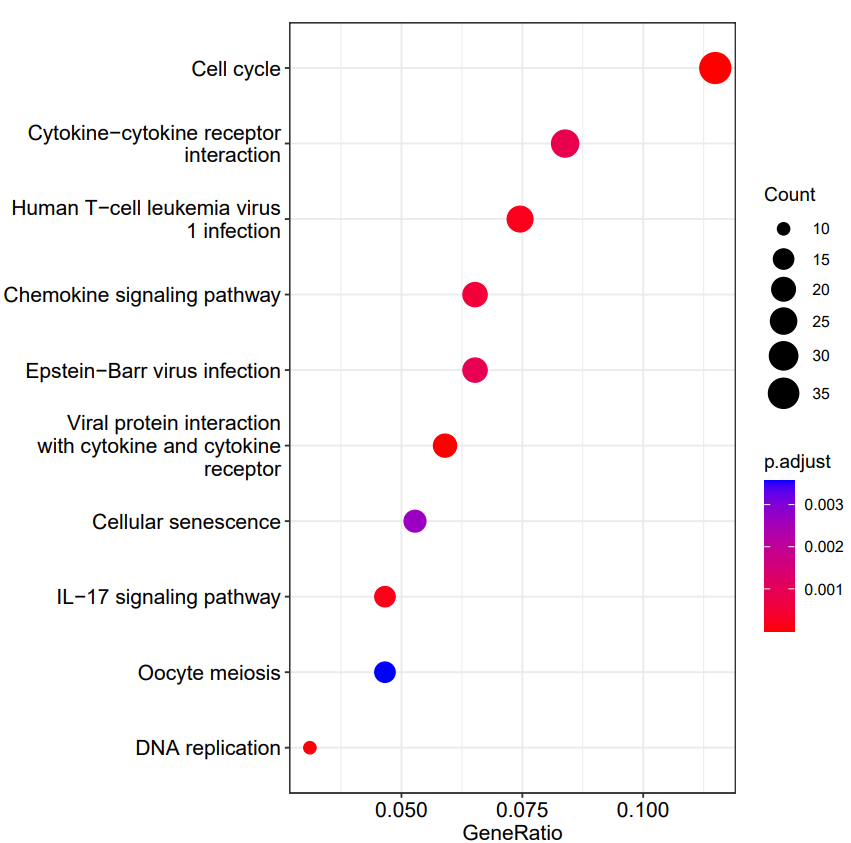
图4 富集气泡图
使用barplot函数和dotplot函数,我们可以生成富集结果的可视化图表,以更直观地展示基因集合的KEGG富集情况。该函数可以生成柱状图或气泡图,其中每个柱子或气泡代表一个富集的KEGG通路,其高度或大小表示富集程度的指标(如p值、基因数等)。
pathview包可以可视化KEGG 通路,我们可以用它查看特定通路图。
代码:
BiocManager::install(“pathview”)
library(pathview)
hsa04750 <- pathview(gene.data = geneList,
pathway.id = “hsa04750”, #上述结果中的hsa04750通路
species = “hsa”,
limit = list(gene=max(abs(geneList)), cpd=1))
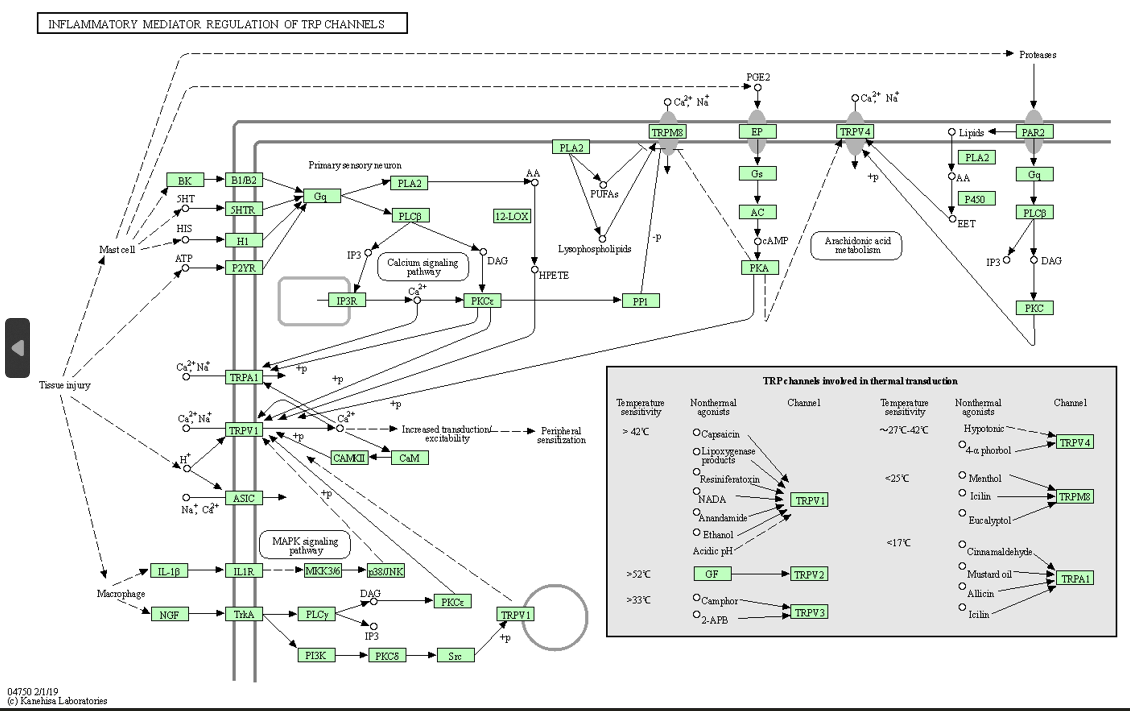
图5 特定通路(hsa04750)图
除了上述基本的KEGG富集分析外,clusterProfiler还提供了其他功能和工具,以进一步深入分析和解释基因集合的生物学意义。例如:
- 通过设置不同的p值和q值阈值,可以调整富集结果的严格程度。
- 可以使用compareCluster函数进行基因集合之间的KEGG通路比较分析,找出不同集合之间的差异和相似性。
- 可以使用geneSets函数生成基因集合的富集结果表格,以便进一步进行统计分析或导出结果。
- 可以使用geneSetPlot函数生成单个KEGG通路的富集结果图表,以详细展示该通路中的基因和其注释信息。
通过使用clusterProfiler包进行KEGG富集分析,我们可以轻松地探索给定基因集合在KEGG通路中的富集情况,进一步理解基因的生物学功能和调控机制。以上教程小果提供了基本的步骤和示例代码,供新手快速入门并进行KEGG富集分析。随着进一步学习和实践,您可以利用clusterProfiler的更多功能和工具,深入挖掘基因集合的生物学意义。更多学习资源请大家移步小果专属云生信平台(云生信 – 学生物信息学 (biocloudservice.com))搜索更多资源哦!