小伙伴们好啊!不知道大家有没有分析拓扑相关域(Topologically Associated Domains,TAD)的需求呢。今天大海哥带来了TADCompare,它功能极其强大。接下来,跟着大海哥认识和学会使用TADCompare吧!
1 介绍
近期的研究利用染色质构象捕获技术,如Hi-C,已经证明了拓扑关联域(TADs)和较小的染色质环,在此统称为“交互域”的重要性。许多这样的域在发育或疾病过程中发生变化,并表现出细胞和条件特异性差异。对交互域动态行为进行量化将有助于更好地理解基因组调控。比较不同细胞和条件下的交互域的方法非常有限。
据我们所知,目前只有三种方法可以适应检测相互作用域边界的变化;其中大多数是为检测特定于TAD的边界变化而开发的。在这三种方法中,localtadsim、HiCDB和DiffTAD,没有一个提供直观、易于使用的方式来调用差异边界。localtadsim和DiffTAD都是两步程序,需要分别定义TADs并通过命令行实用程序进行比较。HiCDB有一个内置的TAD调用器,但不允许对染色体特异性接触矩阵进行比较。所有这三种方法都需要高度特定的数据类型和文件名才能运行。缺乏可用性的问题与诸如缺乏维护、运行缓慢和缺乏统计严谨性等问题相结合。
作者开发了TADCompare,这是一个R包,旨在提供一个快速、准确、用户友好且记录完善的TAD和染色质环路边界差异分析方法。他们引入了一种基于边界得分统计量的方法,并用它来识别五种类型的边界变化。该方法被扩展以允许调用共识边界并在Hi-C重复组之间进行比较。他们还展示了如何使用边界得分统计量来分析相互作用域边界随时间的变化动态。对于差异边界检测和时间序列分析,他们提供了用于分类边界变化的新颖术语。他们使用具有预定义交互作用域的模拟数据演示了TADCompare的鲁棒性以及其揭示不同边界变化的不同生物学角色的能力。总之,TADCompare提供了一个从共识边界调用到差异边界检测的一站式管道,包括时间序列分析。输出以常用的BED格式格式化,允许灵活的下游分析和可视化。
2 功能
TimeCompare是一个R语言包,主要用于比较不同时间序列数据之间的相似性。这个包提供了多种方法来度量和比较时间序列数据的相似性,包括动态时间规整(DTW)、最长公共子序列(LCSS)等。其主要功能包括:
- 计算两个时间序列之间的相似度:通过使用不同的相似度度量方法,可以计算出两个时间序列之间的相似度。这些方法包括DTW、LCSS、欧氏距离等。
- 可视化时间序列数据:TimeCompare包提供了多种可视化工具,可以帮助用户更好地理解和分析时间序列数据。这些工具包括时序图、散点图等。
- 提供多种相似度度量方法:除了DTW、LCSS、欧氏距离等常见的相似度度量方法外,TimeCompare包还提供了其他一些特殊的相似度度量方法,如动态时间规整加权(DTW-Weighted)、动态时间规整加权平均(DTW-Weighted Average)等。
- 支持多种数据类型:TimeCompare包不仅可以处理连续的时间序列数据,还可以处理离散的时间序列数据。此外,它还可以处理混合类型的时间序列数据,即包含连续和离散两种类型的时间序列数据。
3 安装
需要安装的包有些多,小伙伴们可以直接使用大海哥写好的代码:
4 TADCompare
4.1 介绍
TADCompare是一个函数,它允许用户自动识别两个数据集之间的差异性TAD边界。对于每个差异性边界,该包提供了独特的分类(复杂、合并、分割、移动和强度变化),定义了TAD边界在数据集之间的变化方式。
4.2 运行
唯一需要输入的是两个接触矩阵,它们需要符合在“输入数据”小节(https://bioconductor.org/packages/release/bioc/vignettes/TADCompare/inst/doc/Input_Data.html)中指定的允许格式之一。TADCompare函数将自动确定矩阵的类型并将其转换为适当的格式,只要它是支持的格式之一即可。唯一的要求是所有矩阵都采用相同的格式。为了获得最快的结果,大海哥建议使用n×n的矩阵。此外,建议小伙伴们提供自己数据的分辨率。如果未提供分辨率,该包将使用接触矩阵的数字列名进行估计。大海哥将使用来自GM12878细胞系、染色体22、50kb分辨率的数据来演示运行TADCompare的过程。
| #获取包内构建的RAO接触矩阵。
data(“rao_chr22_prim”) data(“rao_chr22_rep”) #我们可以看到,这些都是n x n的矩阵。 dim(rao_chr22_prim) ## [1] 704 704 dim(rao_chr22_rep) ## [1] 704 704 #简单查看数据 rao_chr22_prim[100:105, 100:105] ## 2.1e+07 21050000 21100000 21150000 21200000 21250000 ## 2.1e+07 6215 1047 1073 714 458 383 ## 21050000 1047 4542 2640 1236 619 489 ## 21100000 1073 2640 13468 4680 1893 1266 ## 21150000 714 1236 4680 13600 4430 2124 ## 21200000 458 619 1893 4430 11313 4465 ## 21250000 383 489 1266 2124 4465 11824 #运行指定分辨率的算法 results = TADCompare(rao_chr22_prim, rao_chr22_rep, resolution = 50000) #在没有指定分辨率的情况下重复 no_res = TADCompare(rao_chr22_prim, rao_chr22_rep) ## Estimating resolution |
4.3 TAD的类型比较输出
TADCompare函数返回一个列表,其中包含两个数据框和一个图。第一个数据框包含在至少一个接触矩阵中包含TAD边界的基因组的所有区域。结果如下所示:
| head(results$TAD_Frame)
## Boundary Gap_Score TAD_Score1 TAD_Score2 Differential Enriched_In ## 1 16300000 -18.6589859 0.7698503 7.704411 Differential Matrix 2 ## 2 16850000 0.2361100 7.8431724 7.580056 Non-Differential Matrix 1 ## 3 17350000 0.9683905 3.6628411 3.220253 Non-Differential Matrix 1 ## 4 18000000 -0.1033894 2.3616107 2.347392 Non-Differential Matrix 2 ## 5 18750000 3.0426660 4.7997622 3.558975 Differential Matrix 1 ## 6 18850000 2.6911660 3.7674844 2.680707 Differential Matrix 1 ## Type ## 1 <NA> ## 2 Non-Differential ## 3 Non-Differential ## 4 Non-Differential ## 5 Strength Change ## 6 Strength Change |
“Boundary”列包含给定边界的基因组坐标。“Gap_Score”对应于差异边界得分(边界得分之间的差值的Z分数)。“TAD_Score1”和“TAD_Score2”对应于两个接触矩阵的边界得分。“Differential”简单地表示边界是否具有差异性。“Enriched_In”指示哪个矩阵包含TAD边界。“Type”标识TAD边界变化的类型。注意:由于没有左右参考边界,第一个边界始终被归类为“NA”。
第二个数据框包含与第一个数据框相同的信息,但包括基因组的每一个区域。我们在下面展示它:
| head(results$Boundary_Scores)
## Boundary TAD_Score1 TAD_Score2 Gap_Score Differential Enriched_In ## 1 16300000 0.7698503 7.7044114 -18.658985912 Differential Matrix 2 ## 2 16850000 7.8431724 7.5800558 0.236110024 Non-Differential Matrix 1 ## 3 16900000 -0.4402121 -0.4337948 0.009161239 Non-Differential Matrix 1 ## 4 16950000 -0.7460531 -0.5551327 -0.467725582 Non-Differential Matrix 2 ## 5 17000000 -0.6803509 -0.6512025 -0.037456910 Non-Differential Matrix 2 ## 6 17050000 -0.6626352 -0.5562997 -0.245694062 Non-Differential Matrix 2 ## Type ## 1 <NA> ## 2 Non-Differential ## 3 Non-Differential ## 4 Non-Differential ## 5 Non-Differential ## 6 Non-Differential |
最后,还有一个包含堆叠条形图的图,该图显示了每种类型的TAD边界的流行程度。条形图是一个ggplot2对象,因此完全可定制。我们在下面展示了这个图:
| p <- results$Count_Plot
class(p) ## [1] “gg” “ggplot” plot(p) ## Warning: Removed 1 rows containing missing values (`position_stack()`). |

4.4 预先规定的转录调控区域
我们认识到,用户可能希望在边界比较之前使用自己的TAD调用器来识别TAD边界。因此,我们提供了一种预先规定TAD的选项,以两个数据框的形式,包含两种接触矩阵的TAD边界的染色体、起始和结束坐标。通过这个选项,只有提供的TAD边界将被测试。大海哥在这提供一个使用SpectralTAD TAD调用器的示例:
| #调用SpectralTAD进行拓扑关联域分析
bed_coords1 = bind_rows(SpectralTAD::SpectralTAD(rao_chr22_prim, chr = “chr22”, levels = 3)) ## Estimating resolution bed_coords2 = bind_rows(SpectralTAD(rao_chr22_rep, chr = “chr22”, levels = 3)) ## Estimating resolution #将接触矩阵的TAD边界进行组合 Combined_Bed = list(bed_coords1, bed_coords2) #打印组合的TAD边界列表 Combined_Bed[1] ##[[1]] ## A tibble: 261 × 4 ## chr start end Level ## <chr> <dbl> <dbl> <dbl> ## 1 chr2 8000000 8480000 1 ## 2 chr2 8480000 8880000 1 ## 3 chr2 8880000 9600000 1 ## 4 chr2 9600000 10240000 1 ## 5 chr2 10240000 10880000 1 ## 6 chr2 10880000 11320000 1 ## 7 chr2 11320000 12000000 1 ## 8 chr2 12000000 12320000 1 ## 9 chr2 12320000 12920000 1 ##10 chr2 12920000 13320000 1 ## ℹ 251 more rows ## ℹ Use `print(n = …)` to see more rows |
在上面,我们可以看到SpectralTAD输出的数据集包含名为“start”和“end”的列,分别表示每个TAD的起始和结束坐标。这是必需的,尽管任何TAD调用器的输出都可以有效地与一些数据操作一起使用。 “Level”列表示TAD在层次结构中的级别。
| #用预先规定的TADs运行TADCompare
TD_Compare = TADCompare(rao_chr22_prim, rao_chr22_rep, resolution = 50000, pre_tads = Combined_Bed) #返回边界 head(TD_Compare$TAD_Frame) ## Boundary Gap_Score TAD_Score1 TAD_Score2 Differential Enriched_In ## 1 16850000 0.23611002 7.8431724 7.5800558 Non-Differential Matrix 1 ## 2 17250000 -0.08875306 0.1103987 0.1409999 Non-Differential Matrix 2 ## 3 17300000 -0.12694247 -0.6160007 -0.5549497 Non-Differential Matrix 2 ## 4 17350000 0.96839048 3.6628411 3.2202526 Non-Differential Matrix 1 ## 5 17600000 -0.06733403 -0.4153272 -0.3809658 Non-Differential Matrix 2 ## 6 18000000 -0.10338944 2.3616107 2.3473922 Non-Differential Matrix 2 ## Type ## 1 Non-Overlap ## 2 Non-Overlap ## 3 Non-Overlap ## 4 Non-Overlap ## 5 Non-Overlap ## 6 Non-Differential #进行测试来显示所有预先规定的边界都被返回了 table(TD_Compare$TAD_Frame$Boundary %in% bind_rows(Combined_Bed)$end) ## ## TRUE ## 96 |
在这里,我们可以看到返回的边界仅限于我们预先规定的那些。
4.5 TADCompare结果的可视化
我们提供了使用DiffPlot函数可视化差异性TAD边界的方法。作为输入,DiffPlot接受来自TADCompare函数和原始接触矩阵的输出。作为输出,它返回一个可以进一步定制的ggplot2对象。我们演示了在染色体2的子集上,以40kb分辨率处理的数据中,GM12878和IMR90细胞系之间的差异可视化。
| data(“GM12878.40kb.raw.chr2”)
data(“IMR90.40kb.raw.chr2”) mtx1 <- GM12878.40kb.raw.chr2 mtx2 <- IMR90.40kb.raw.chr2 res <- 40000 # Set resolution #全局标准化矩阵,以便于进行更直观的比较(不会影响TAD调用) mtx1_total <- sum(mtx1) mtx2_total <- sum(mtx2) (scaling_factor <- mtx1_total / mtx2_total) ## [1] 0.3837505 #根据哪个矩阵较小来重新调整矩阵的大小。 if (mtx1_total > mtx2_total) { mtx2 <- mtx2 * scaling_factor } else { mtx1 <- mtx1 * (1 / scaling_factor) } #有趣的区域的坐标 start_coord <- 8000000 end_coord <- 16000000 #另一个有趣的坐标 # start_coord <- 40000000 # end_coord <- 48000000 |
4.5.1 比较TAD边界分数
| #运行TADCompare的当前版本
TD_Compare <- TADCompare(mtx1, mtx2, resolution = res) #运行预先规定的TADs绘图算法 p <- DiffPlot(tad_diff = TD_Compare, cont_mat1 = mtx1, cont_mat2 = mtx2, resolution = res, start_coord = start_coord, end_coord = end_coord, show_types = TRUE, point_size = 5, max_height = 5, rel_heights = c(1, 2), palette = “RdYlBu”) ## Warning: The `<scale>` argument of `guides()` cannot be `FALSE`. Use “none” instead as ## of ggplot2 3.3.4. ## ℹ The deprecated feature was likely used in the TADCompare package. ## Please report the issue at ## <https://github.com/dozmorovlab/TADCompare/issues>. ## This warning is displayed once every 8 hours. ## Call `lifecycle::last_lifecycle_warnings()` to see where this warning was ## generated. plot(p) |
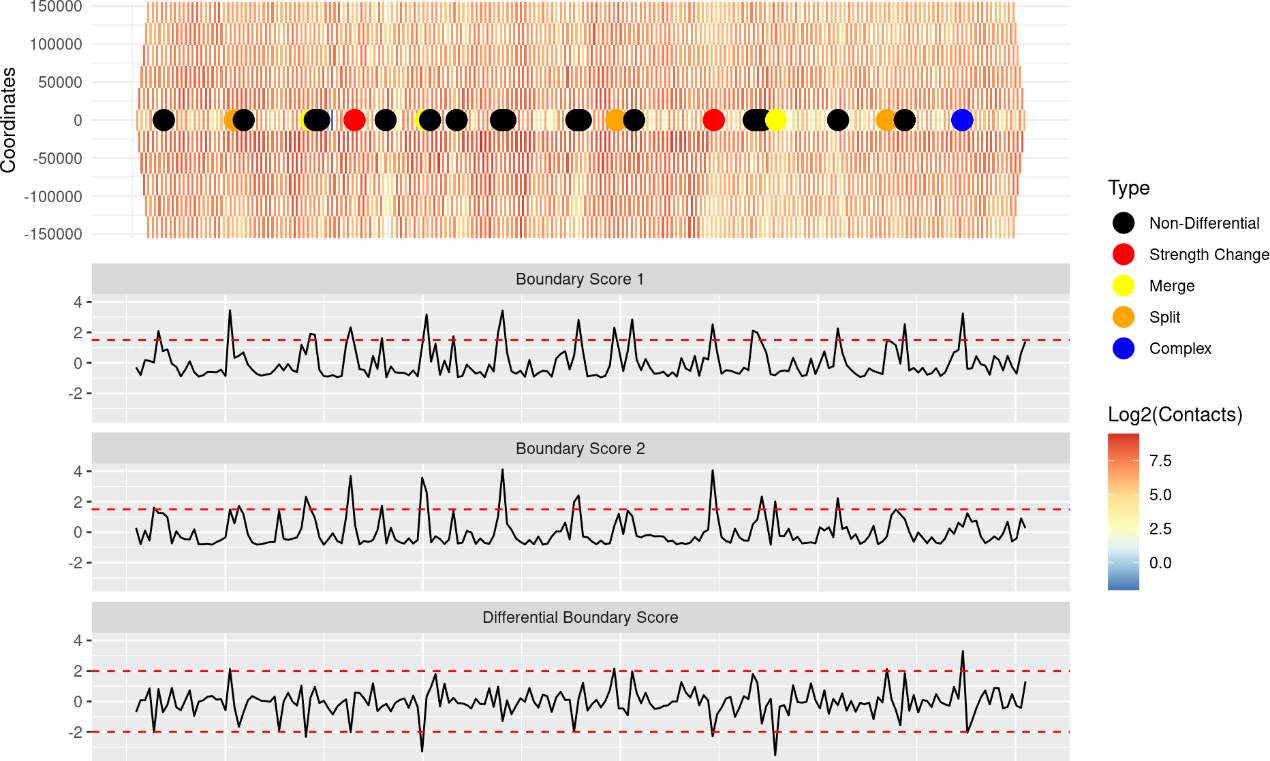
从这些结果中,我们可以看到两个矩阵(“Boundary score 1”和“Boundary score 2”)中的边界得分经常被检测为显著边界(边界得分高于1.5的阈值),但它们是非差异性的(黑色圆点)。差异性边界对应于“Differential boundary score”轨迹,其中检测到绝对边界得分差异高于2.0的阈值。不同类型的差异性边界根据TADCompare手稿中描述的模式进行定义。请注意,通过将“show_types”设置为FALSE,可以禁用按类型着色;只会显示差异性和非差异性标签。
4.5.2 比较预定义的TAD边界
我们还可以提供包含每个接触矩阵的TAD边界的TAD坐标列表来使用预指定的TAD。该列表的长度应为2。下面我们将展示如何使用SpectralTAD进行预指定:
| #调用SpectralTAD进行拓扑关联域分析
bed_coords1 = bind_rows(SpectralTAD(mtx1, chr = “chr2”, levels = 3)) ## Estimating resolution bed_coords2 = bind_rows(SpectralTAD::SpectralTAD(mtx2, chr = “chr2”, levels = 3)) ## Estimating resolution #将数据放入列表以进行绘图过程 Combined_Bed = list(bed_coords1, bed_coords2) #运行带有预先规定的 TADs 的 TADCompare TD_Compare <- TADCompare(mtx1, mtx2, resolution = res, pre_tads = Combined_Bed) #运行预先规定的TADs绘图算法 p <- DiffPlot(tad_diff = TD_Compare, cont_mat1 = mtx1, cont_mat2 = mtx2, resolution = res, start_coord = start_coord, end_coord = end_coord, pre_tad = Combined_Bed, show_types = FALSE, point_size = 5, max_height = 10, rel_heights = c(1.5, 2), palette = “RdYlBu”) plot(p) |
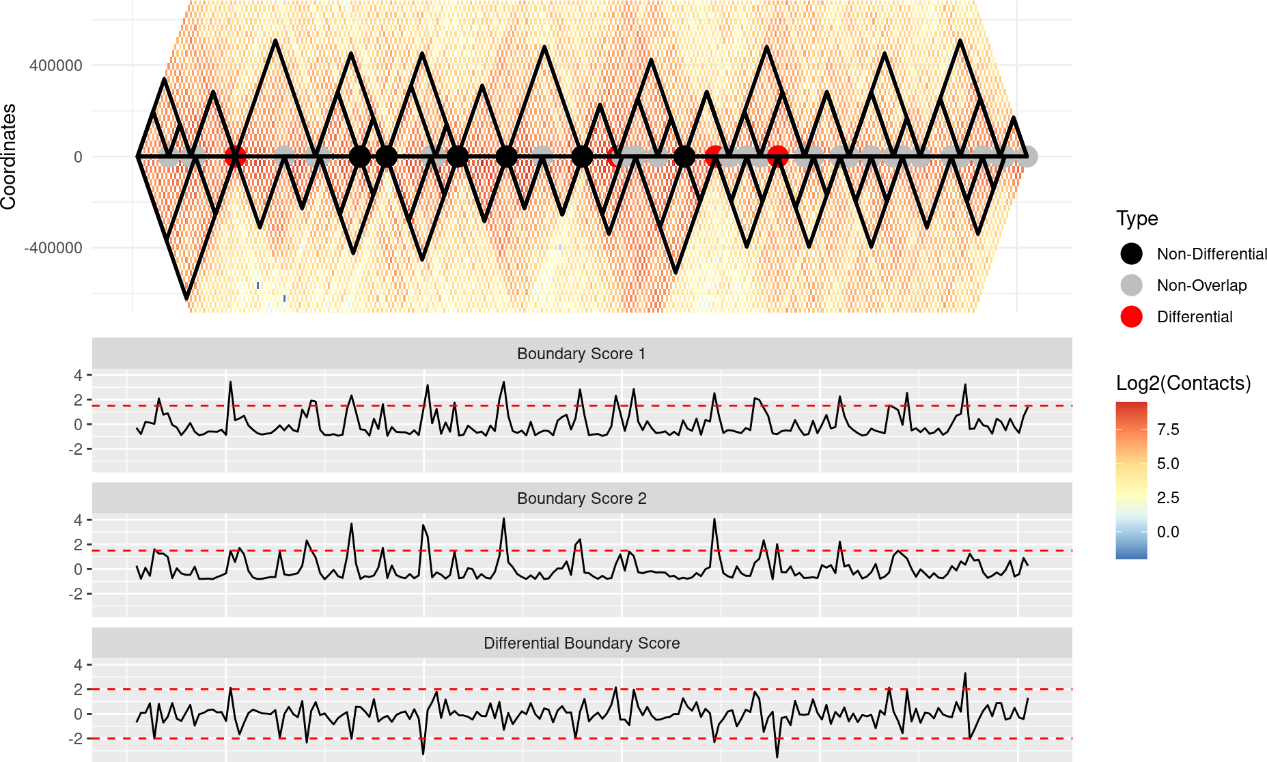
正如我们所看到的,预先规定的TADs允许我们勾勒出TAD边界,从而增强可视化效果。此外,现在呈现的差异边界与那些由TAD调用者调用的边界相对应,而不是那些由TADCompare使用边界分数检测到的边界。请注意,预先规定的TAD边界并不一定对应于边界分数;因此,分类模式是根据预定义的TAD边界应用的。然而,使用预先规定的TADs使得视觉上更容易解释差异的差异。因此,我们建议将“show_types”设置为FALSE。此外,对于预先规定的TADs,引入了一个名为“Non-Overlap”的新类别。非重叠对应于被TADCompare确定为非差异的边界,但在被TAD调用者调用时不重叠。
5 TimeCompare
5.1 介绍
TimeCompare是一个用于时间序列数据分析的函数。简单来说,用户输入一个代表至少四个时间点的接触矩阵列表。TimeCompare函数可以处理较少的时间点,但时间变化的分类可能会出错。输出是一个包含在至少一个时间点检测到的所有具有TAD边界的区域的数据框。这些区域根据TAD边界随时间如何演变进一步分为六种不同的时间变化类型(高度常见、动态、早期/晚期出现和早期/晚期消失)。该函数还返回一个总结每个TAD边界出现的图,以及另一个包含每个区域的更改摘要的数据框,无论是否检测到边界。
5.2 运行
TimeCompare函数接收一组格式类似于TADCompare函数的矩阵。与TADCompare一样,TimeCompare函数将估计分辨率并将矩阵转换为适当的格式。在这个例子中,我们使用时间变化下的稀疏3列接触矩阵,表示HCT-116细胞系。这些矩阵代表用TAD边界破坏的植物生长素处理的一个单独的染色体22细胞样本。数据然后在植物生长素撤出后四个时间点(20、40、60和180分钟)进行测序。一旦植物生长素被撤出,TAD边界会慢慢恢复。使用TimeCompare函数,我们可以追踪植物生长素撤出后的TADs的恢复情况。
| #获取联系矩阵的列表
data(“time_mats”) #检查格式 head(time_mats[[1]]) ## # A tibble: 6 × 3 ## X1 X2 X3 ## <dbl> <dbl> <dbl> ## 1 16050000 16050000 14 ## 2 16100000 16100000 3 ## 3 16200000 16200000 1 ## 4 16250000 16250000 1 ## 5 16300000 16300000 10 ## 6 16350000 16350000 2 #这些是稀疏的三列矩阵 #运行TimeCompare time_var <- TimeCompare(time_mats, resolution = 50000) ## Converting to n x n matrix ## Matrix dimensions: 704×704 ## Matrix dimensions: 704×704 ## Matrix dimensions: 704×704 ## Matrix dimensions: 704×704 |
TimeCompare函数返回的第一个项目是TAD_Bounds,这是一个数据框,包含了在至少一个时间点检测到的TAD边界的所有区域。
| head(time_var$TAD_Bounds)
## Coordinate Sample 1 Sample 2 Sample 3 Sample 4 Consensus_Score ## 1 16900000 -0.6733709 -0.7751516 -0.7653696 15.1272252 -0.71937026 ## 2 17350000 3.6406563 2.3436229 3.0253018 0.7840556 2.68446232 ## 3 18850000 0.6372268 6.3662244 -0.7876844 6.9255446 3.50172562 ## 4 20700000 1.5667926 3.0968633 2.9130479 2.8300136 2.87153075 ## 5 22000000 -1.0079676 -0.7982571 0.6007264 3.1909178 -0.09876534 ## 6 22050000 -1.0405532 -0.9892242 -0.2675822 4.2737511 -0.62840320 ## Category ## 1 Late Appearing TAD ## 2 Early Disappearing TAD ## 3 Early Appearing TAD ## 4 Dynamic TAD ## 5 Late Appearing TAD ## 6 Late Appearing TAD |
第一列对应于基因组坐标。以“Sample”为前缀的列对应于每个样本中给定坐标处的边界得分。共识得分是所有样本中得分的中位数,类别对应于变化的类型。
All_Bounds是第二个列表条目,其结构与TAD_Bounds数据框相同,但它包括了基因组中的每个区域,无论它是否是转录调控区域。
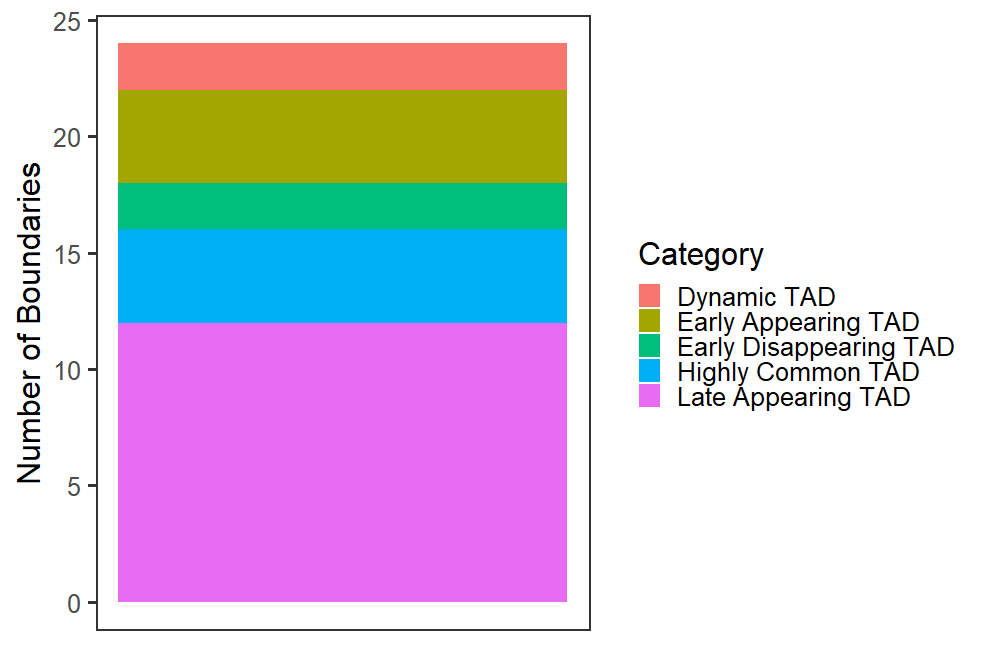
还有一个堆叠条形图,该图显示了数据中每种类型的时间边界出现的次数。这个图是在ggplot2中创建的,并且是完全可定制的。
| time_var$Count_Plot |
6 ConsensusTAD
6.1 介绍
ConsensusTADs函数实现了一种在多个数据集上调用TADs的方法。它使用n个重复或细胞系的中位数边界得分来计算一致的TAD边界共识,即在所有重复中一致检测到的。这有效地过滤掉了只在一两个重复或细胞系中出现的噪声TAD边界。
6.2 运行
ConsensusTADs函数与TimeCompare函数具有基本相同的输入(一个接触矩阵列表)。它为每个区域提供共识TAD分数,并在基因组的每个接触矩阵上进行汇总。它还提供了一组具有显著TAD分数的区域。这些区域可以被视为共识TAD边界。通过使用这些边界,我们可以在一组重复、条件或时间点上获得一组单一的TAD汇总。在本例中,我们使用两个重复样本。
我们演示了如何通过调用共识TADs来运行ConsensusTADs,该函数对单个体样品进行处理后创建的时间变化接触矩阵进行操作,其中处理破坏了其TAD边界,然后跟踪它们在四个时间点(20、40、60和180分钟)的返回。共识边界得分旨在提供所有时间点的TAD边界的总结。
| #获取包内构建的RAO接触矩阵。
data(“time_mats”) #运行ConsensusTADs con_tads <- ConsensusTADs(time_mats, resolution = 50000) ## Converting to n x n matrix ## Matrix dimensions: 704×704 ## Matrix dimensions: 704×704 ## Matrix dimensions: 704×704 ## Matrix dimensions: 704×704 |
ConsensusTADs 函数返回两个数据框。第一个数据框,Consensus,包含了基于共识得分的所有包含共识 TADs 的区域。
| head(con_tads$Consensus)
## Coordinate Sample 1 Sample 2 Sample 3 Sample 4 Consensus_Score ## 1 16900000 -0.6733709 -0.7751516 -0.7653696 15.1272252 -0.71937026 ## 2 17350000 3.6406563 2.3436229 3.0253018 0.7840556 2.68446232 ## 3 18850000 0.6372268 6.3662244 -0.7876844 6.9255446 3.50172562 ## 4 20700000 1.5667926 3.0968633 2.9130479 2.8300136 2.87153075 ## 5 22000000 -1.0079676 -0.7982571 0.6007264 3.1909178 -0.09876534 ## 6 22050000 -1.0405532 -0.9892242 -0.2675822 4.2737511 -0.62840320 |
列对应于具有显著边界得分的区域的坐标,每个区域的个体边界得分以及共识得分。
第二个数据框All_Regions与Consensus数据框相同,但它包括基因组中出现在两个矩阵中的每个区域。
| head(con_tads$All_Regions)
## Coordinate Sample 1 Sample 2 Sample 3 Sample 4 Consensus_Score ## 1 16900000 -0.6733709 -0.7751516 -0.7653696 15.1272252 -0.7193703 ## 2 16950000 -0.6343671 -0.6624276 -0.4107173 -0.6656620 -0.6483973 ## 3 17000000 -1.0409280 -0.7618272 0.7910678 -1.0008876 -0.8813574 ## 4 17050000 -0.9199917 -0.7123180 0.6712399 -0.8254422 -0.7688801 ## 5 17100000 -0.6097913 -0.4785485 -0.6232367 -0.2095729 -0.5441699 ## 6 17150000 -0.7215028 -0.5669957 0.2413230 -0.6332710 -0.6001333 |
7 结语
看了这么多,小伙伴可能有些晕乎乎的,大海哥来总结一下。TADCompare有三个主要功能:TADCompare用于差异性TAD分析,TimeCompare用于时间序列分析,ConsensusTADs用于共识边界识别。
TimeCompare是一个功能强大、操作简便的时间序列数据分析工具。大海哥相信无论是对于学术研究还是实际应用,都有很大的帮助。
如果小伙伴有其他数据分析需求,可以尝试使用本公司新开发的生信分析小工具云平台,零代码完成分析,非常方便奥,云平台网址为:(http://www.biocloudservice.com/home.html),其中也包括了通路表达分析(http://www.biocloudservice.com/313/313.php),单细胞的基因共表达分析(http://www.biocloudservice.com/906/906.php)等各种小工具哦,有兴趣的小伙伴可以登录网站进行了解 (http://www.biocloudservice.com/home.html) 。
参考文献
Cresswell, K. G., & Dozmorov, M. G. (2020). TADCompare: An R Package for Differential and Temporal Analysis of Topologically Associated Domains. Frontiers in genetics, 11, 158. https://doi.org/10.3389/fgene.2020.00158