今天小果给大家介绍一个简单好用的生信分析工具箱— —oncoEnrichR(2023年8月 发表 于International Journal of Cancer (IJC))功能非常强大!
oncoEnrichR 是一个 R 软件包,用于在癌症背景下对人类基因列表进行功能检测。该软件 包利用大量的先验分子知识,从系统层面和单个基因层面进行多维度的生物学解释。主要用 于对来自高通量癌症生物学实验(如 CRISPR 筛选、蛋白质近似标记或基因表达谱分析)的 基因列表(此处称为查询集)进行探索性分析和优先排序。该工具可查询大量高质量的数据 资源,以便在交互式报告中汇总癌症相关注释和分析。
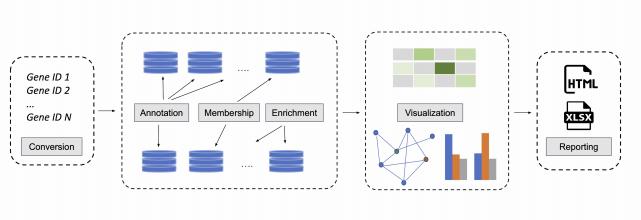
值得注意oncoEnrichR 还有在线工具: https://oncotools.elixir.no
一 、安装
下面小果来教你怎么进行安装
要求: oncoEnrichR requires R (>= version 4.1)
安装命令
install.packages(‘remotes’)
remotes::install_github(‘sigven/oncoEnrichR’, ref = “v1.4.2”) library(oncoEnrichR)]()
二 、运行
oncoEnrichR 通过以下三个函数执行其操作:
1、oncoEnrichR::load_db()
加载 oncoEnrichR 的底层注释数据存储库,并将其保存到本地缓存目录。使用 googledrive R 软件包
2、oncoEnrichR::oncoo_enrich():
包括两个主要处理步骤
(1)将人类基因/蛋白质标识符(如 UniProt 编号、 RefSeq/Ensembl 转录本标识符等)的 输入/查询列表作为输入,并进行统一的标识符转换;
(2)根据人类基因的癌症相关属性及其相互关系的基础数据源,对查询集进行广泛的注 释、富集和成员分析。
从技术上讲,该方法返回一个包含所有分析内容的列表对象。具体参数/选项和默认值概述 如下:
onco_enrich(
query = NULL,
oeDB = NULL,
query_id_type = “symbol”,
ignore_id_err = TRUE,
html_floating_toc = T,
html_report_theme = “default”,
project_title = “_Project title_”,
project_owner = “_Project owner_”,
project_description = “_Project description_”,
bgset = NULL,
bgset_id_type = “symbol”,
bgset_description = “All protein-coding genes”,
enrichment p value_cutoff = 0.05,
enrichment p value_adj = “BH”,
enrichment q value_cutoff = 0.2,
enrichment_min_geneset_size = 10,
enrichment_max_geneset_size = 500,
enrichment_plot_num_terms = 20,
enrichment_simplify_go = TRUE,
subcellcomp_min_confidence = 3,
subcellcomp_min_channels = 1,
subcellcomp_show_cytosol = FALSE,
regulatory_min_confidence = “D”,
fitness_max_score = -2,
ppi_add_nodes = 30,
ppi_string_min_score = 0.9,
ppi_string_network_type = “functional”,
ppi_biogrid_min_evidence = 3,
ppi_node_shadow = TRUE,
ppi_show_drugs = TRUE,
ppi_show_isolated_nodes = FALSE,
show_ppi = TRUE,
show_disease = TRUE,
show_top_diseases_only = TRUE,
show_cancer_hallmarks = TRUE,
show_drug = TRUE,
show_enrichment = TRUE,
show_aberration = TRUE,
show_coexpression = TRUE,
show_cell_tissue = FALSE,
show_ligand_receptor = TRUE,
show_regulatory = TRUE,
show_unknown_function = TRUE,
show_prognostic = TRUE,
show_subcell_comp = TRUE,
show_synleth = TRUE,
show_fitness = TRUE,
show_complex = TRUE,
show_domain = TRUE)
3、oncoEnrichR::write()
包括两个主要处理步骤:
(1)将 oncoEnrichR::oncoo_enrich() 返回的原始分析结果转换为各种可视化和交互式表 格
(2)通过以下方式组装和生成最终分析报告
A) 结构化交互式 oncoEnrichR HTML 报告
B) 多表 Excel 工作簿
三 、输出说明
使用 oncoEnrichR 进行分析的输出结果以两种不同的格式提供:
(1)交互式 HTML 报告
(2)多表 Excel 工作簿
1、HTML 报告
在这里,我们展示了来自 OncoEnricHR 交互式 HTML 报告的结果,下面是可以使用该工具 回答的各种类型的问题。
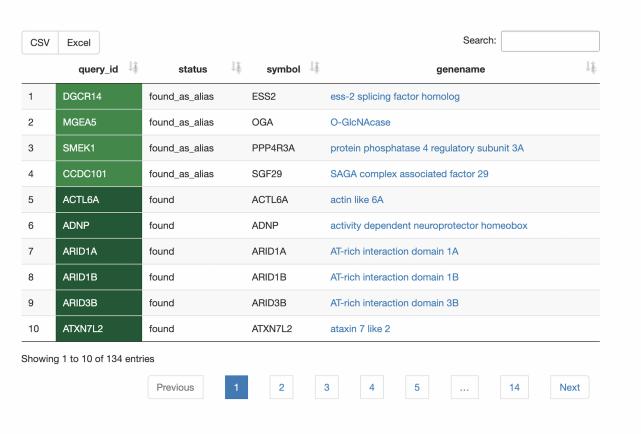
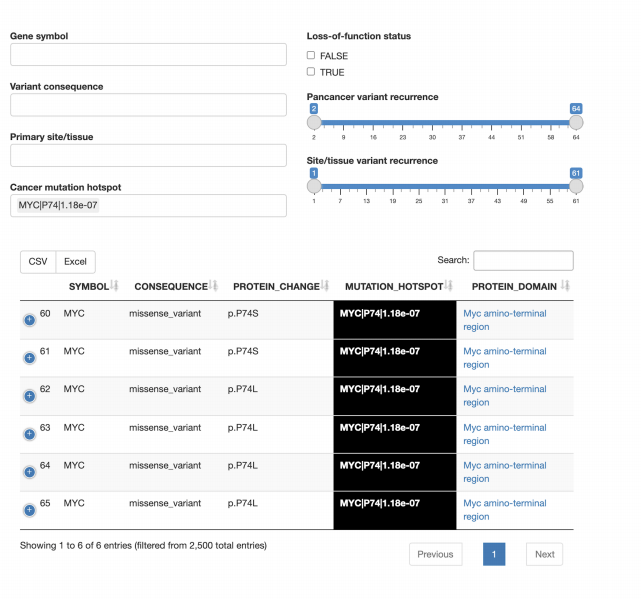
查询验证
![]() 所有的基因/查询标识符都被正确识别了吗?某些查询标识符是否已过时, 并且不 再被视为主标识符?
所有的基因/查询标识符都被正确识别了吗?某些查询标识符是否已过时, 并且不 再被视为主标识符?
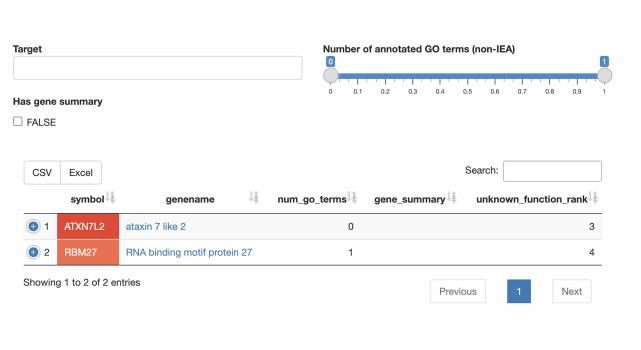
特征未知基因
· 查询集的哪些成员具有特征不明显/未知的功能?
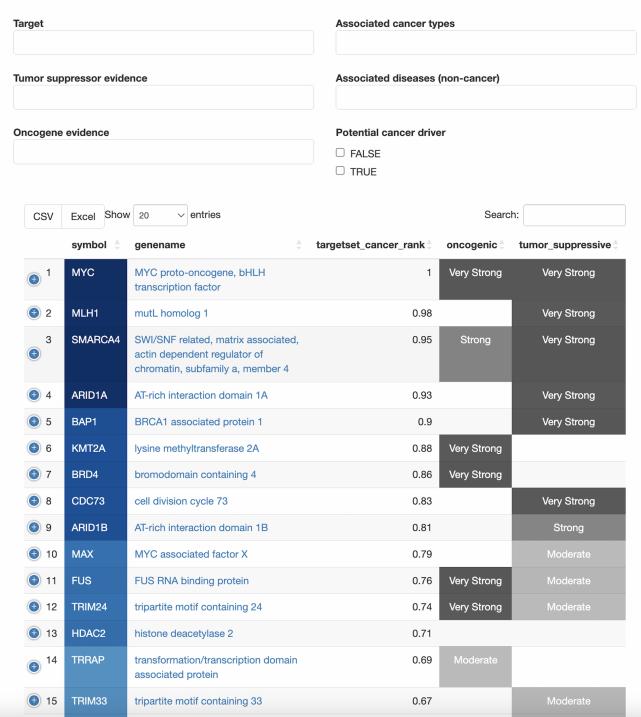
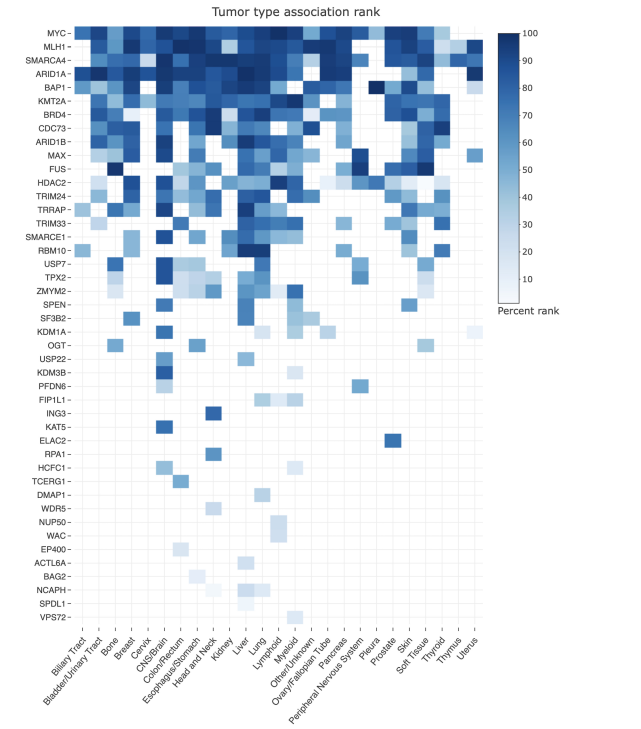
癌症联系
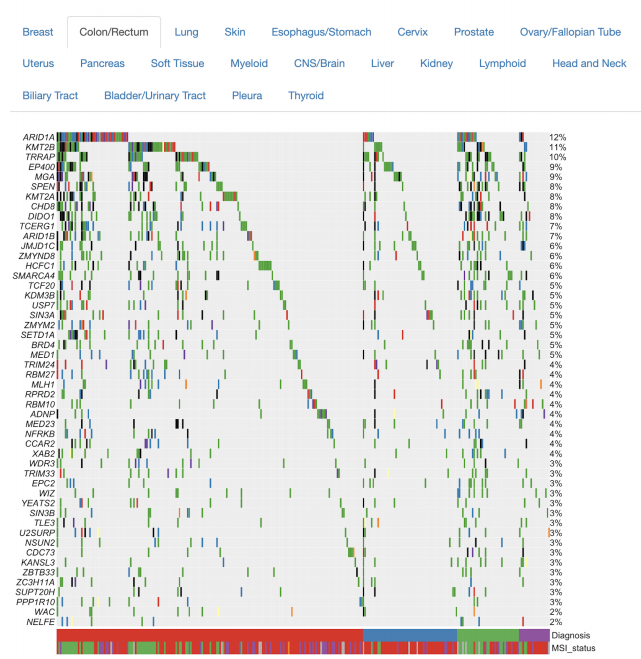
· 哪些肿瘤类型与 QuerySet 中的基因相关?到什么程度?哪些基因被归类为肿瘤抑 制基因、原癌基因或潜在的癌症驱动基因?
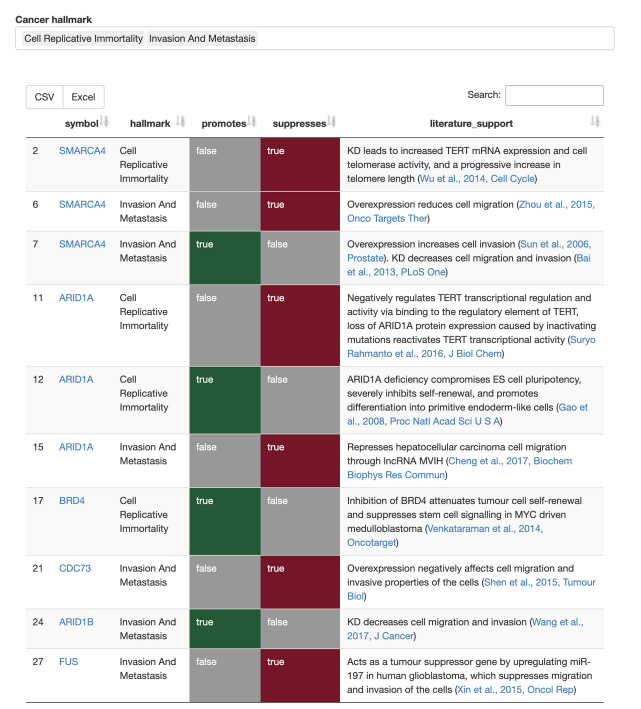
癌症标志性证据
· 哪些基因被认为是癌症的特征?
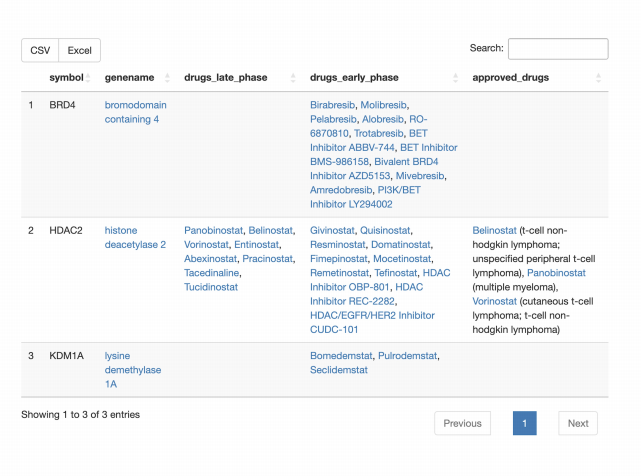
药物联系
· 是否有适用于查询集成员的靶向癌症药物?对于哪些癌症适应症,有针对查询集成 员的已批准药物?所有查询集成员的目标可跟踪性是什么?
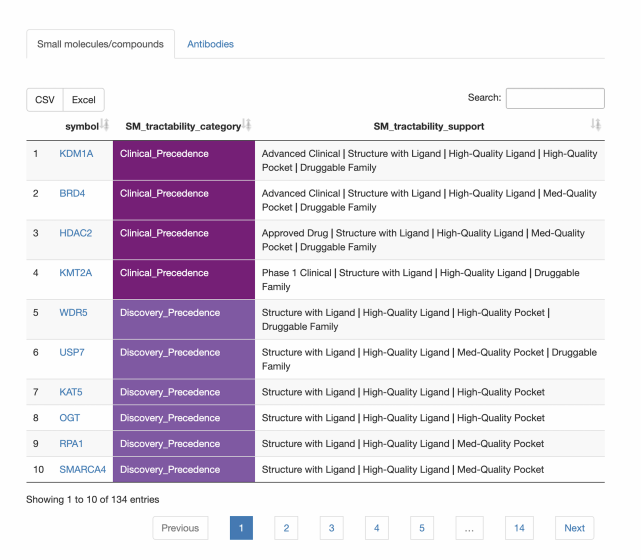
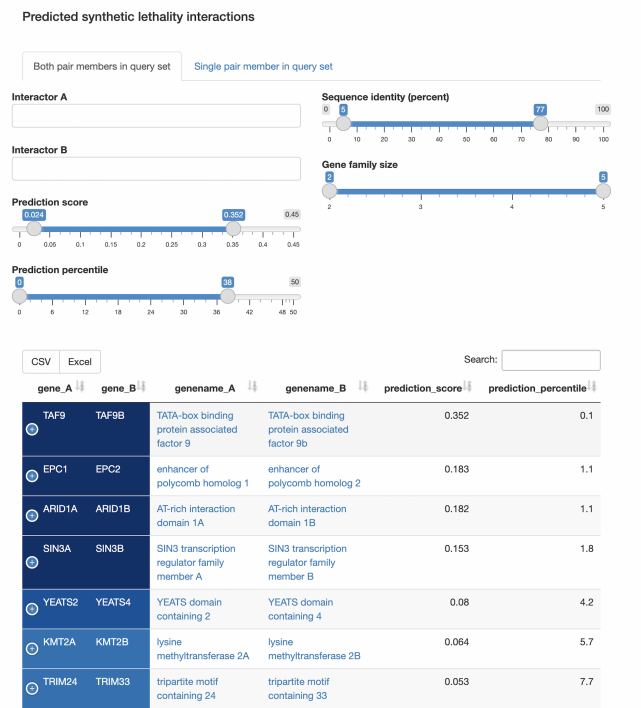
合成致死率
· 预测哪些基因参与合成致死性相互作用?
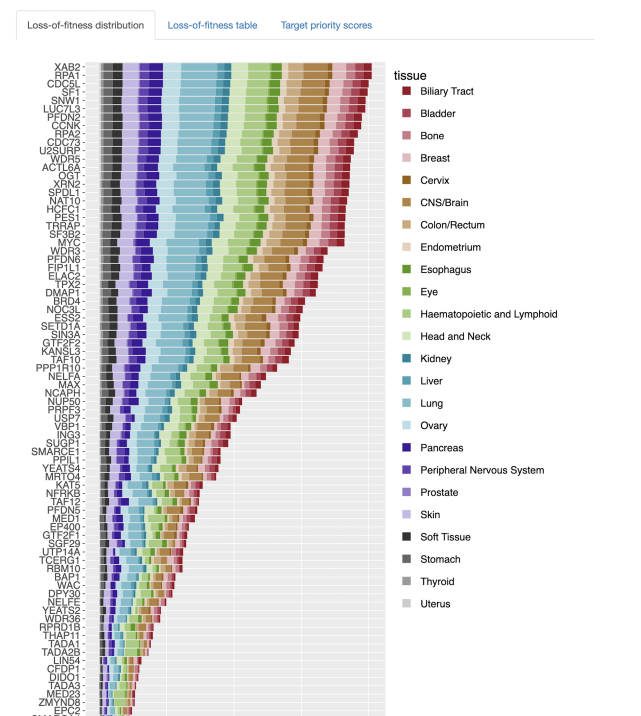
基因适合度得分
· 考虑到癌细胞系的大规模全基因组 CRISPR–Cas9 脱落筛选数据,查询集中的哪些 成员对细胞适合度有显著影响?
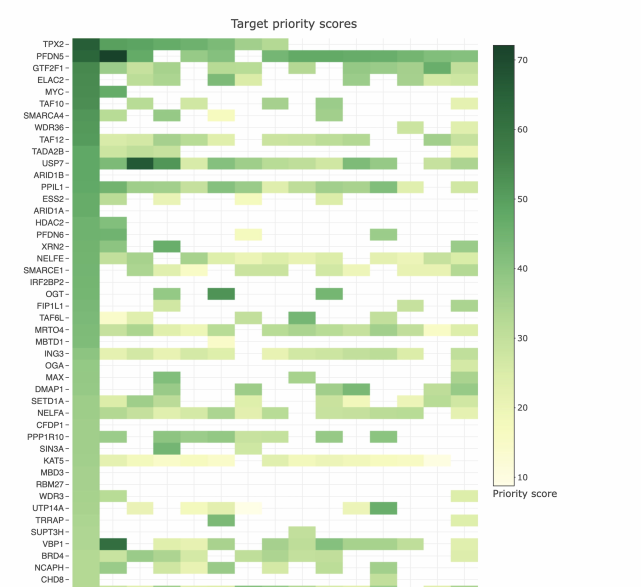
蛋白质复合物
· 查询集的成员是否与癌症相关的蛋白质复合物有关?
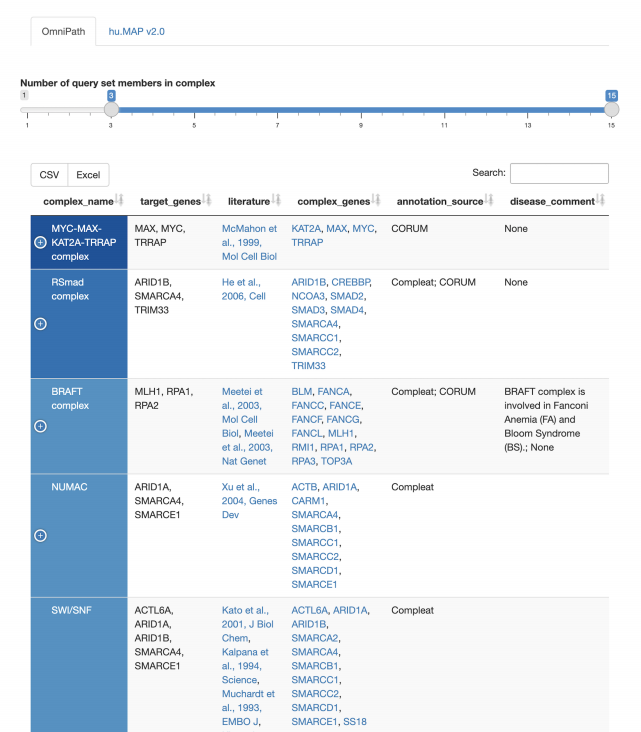
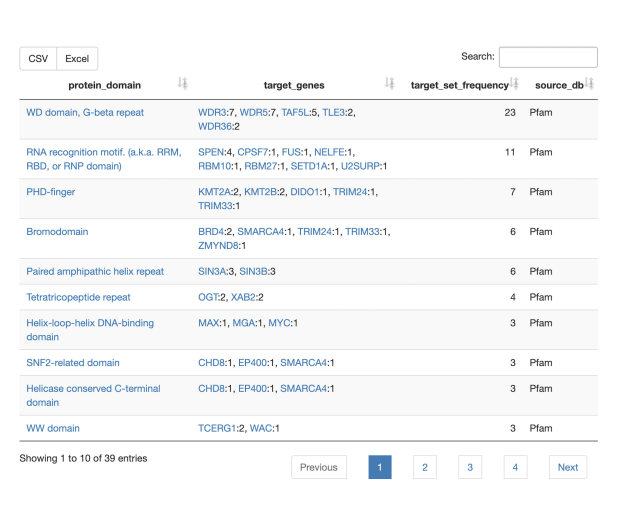
蛋白质结构域
· 哪些蛋白质结构域在查询集的成员中最常见?
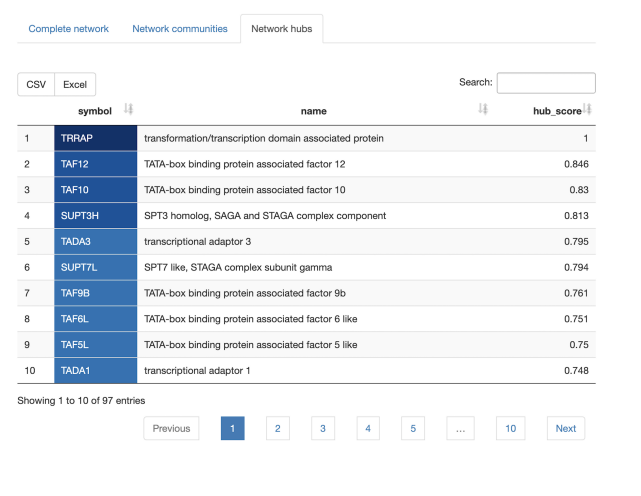
蛋白质相互作用网络
· 如何在查询集中表示蛋白质-蛋白质相互作用?哪些蛋白质是相互作用网络中的枢 纽?查询集的哪些成员形成网络中的社区结构?

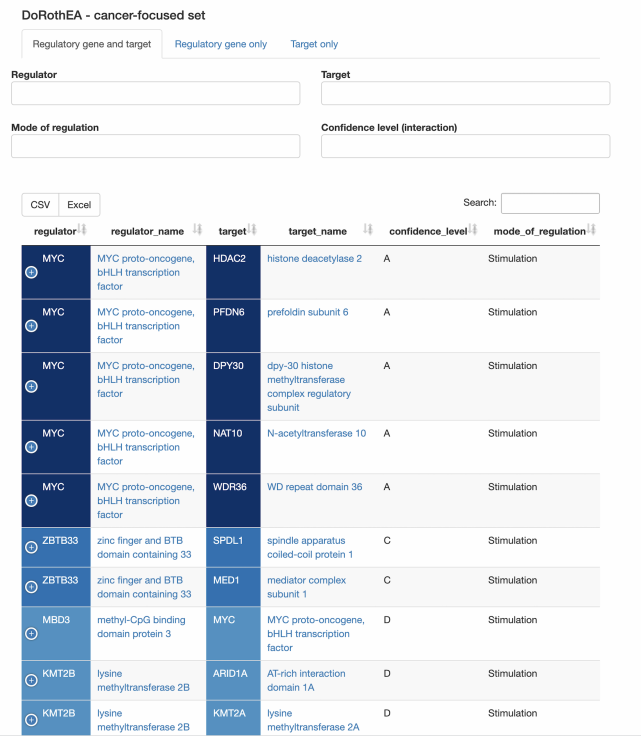
调节相互作用
· 查询集中是否存在转录调控相互作用?是否同时存在 TF/调控因子和靶标? 存在哪 些其他 TF 的调控靶标?其他目标的调控因子是什么?
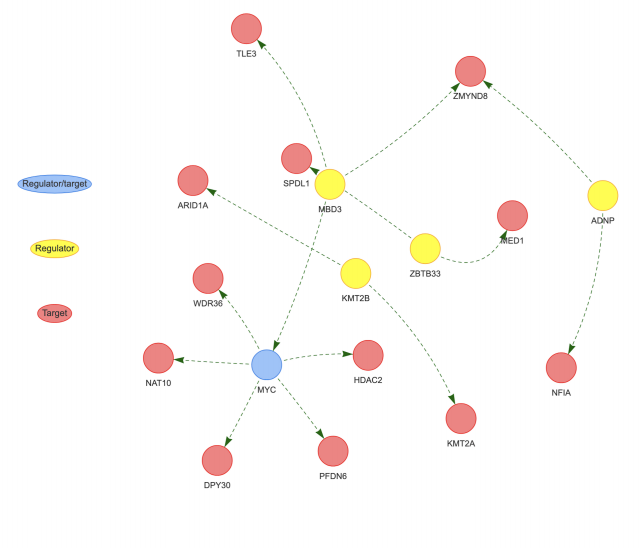
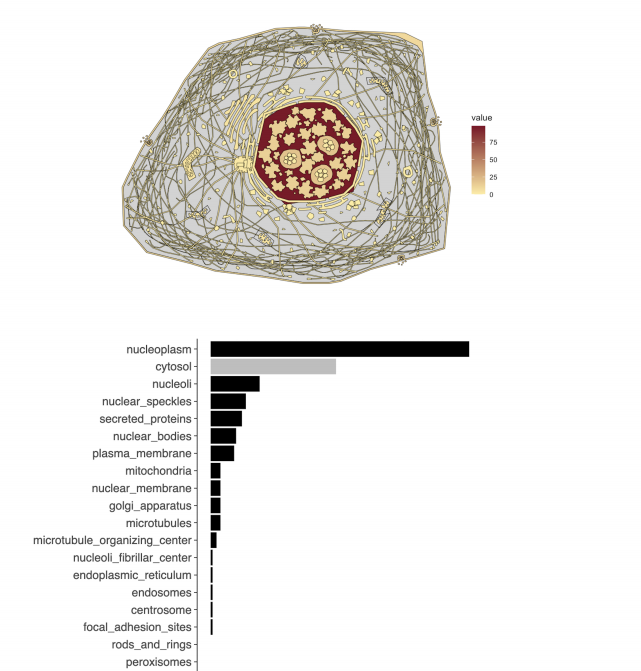
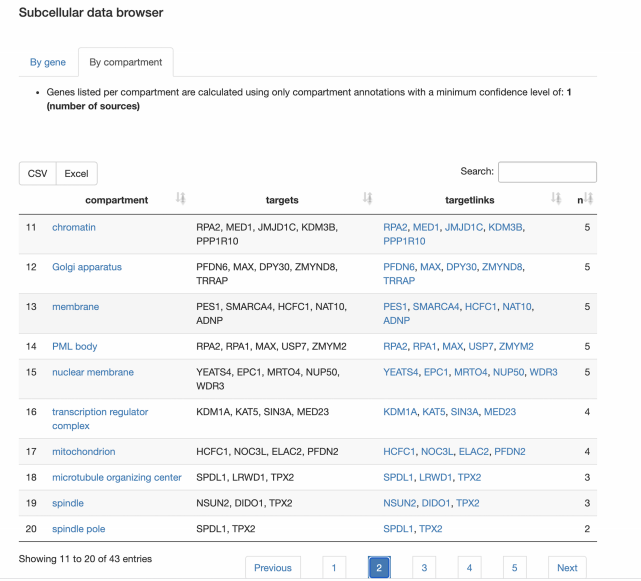
亚细胞区室
· 如何在查询集中表示亚细胞隔室?
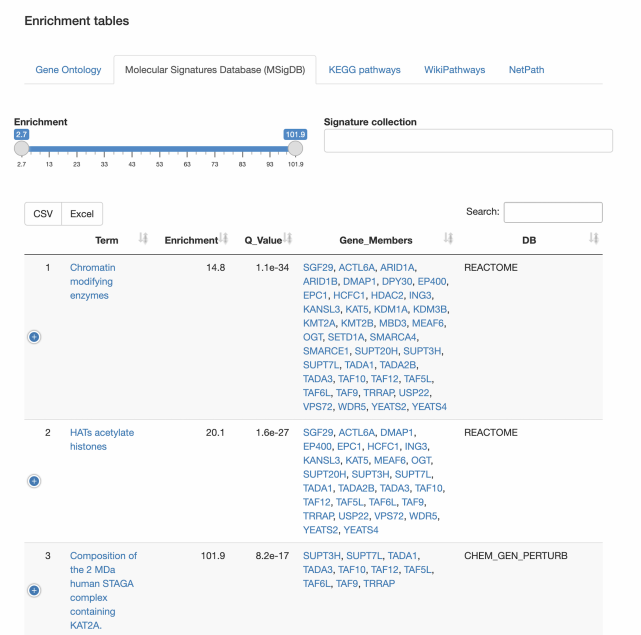
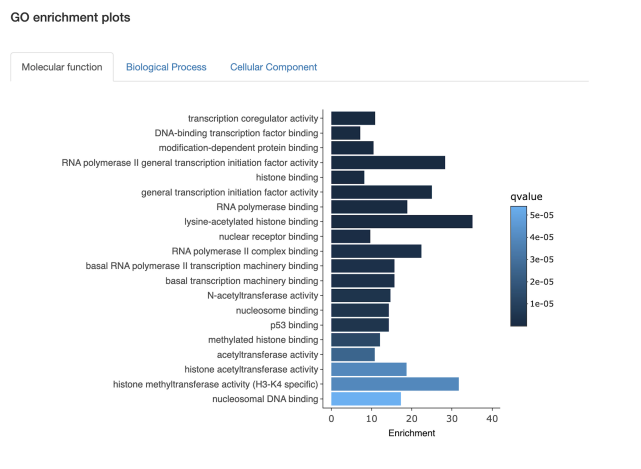
功能和途径富集
· 查询集中富集了哪些生物途径/过程、分子功能或癌症基因标记?
组织和细胞类型富集
· 正常/健康组织中查询基因的组织和细胞类型特异性表达模式是什么?
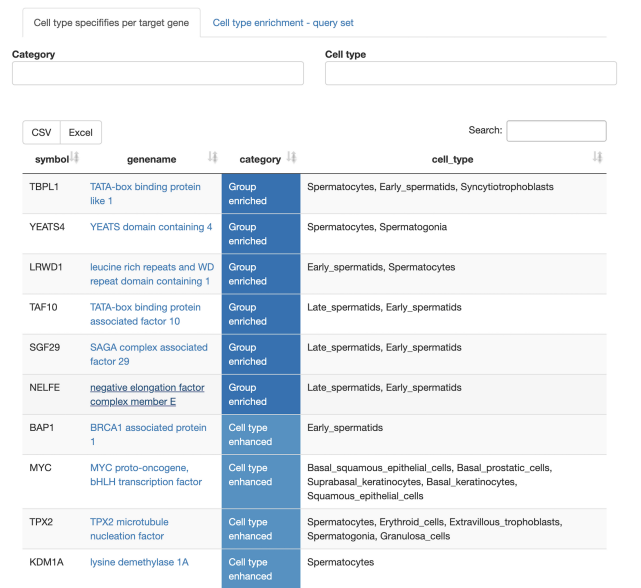
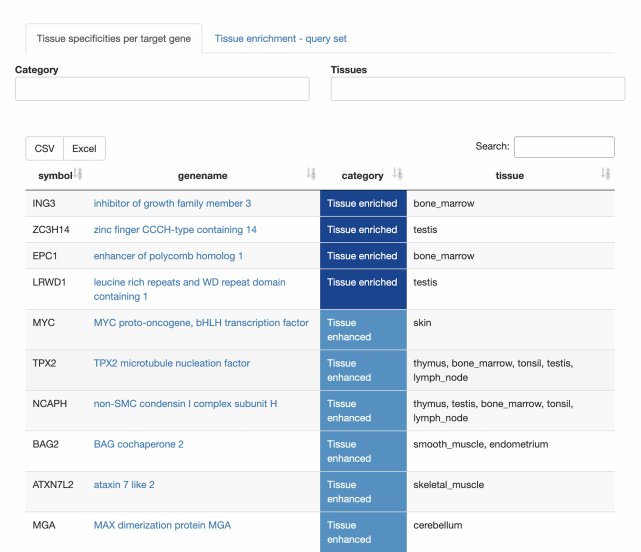
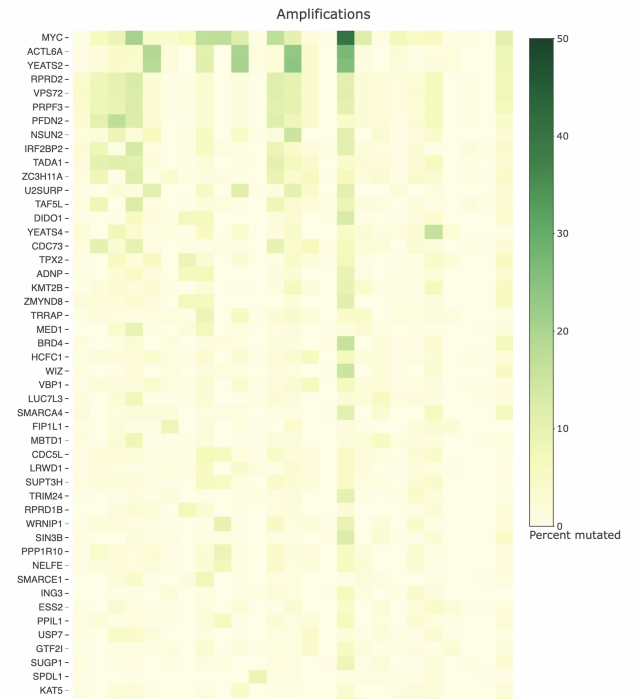
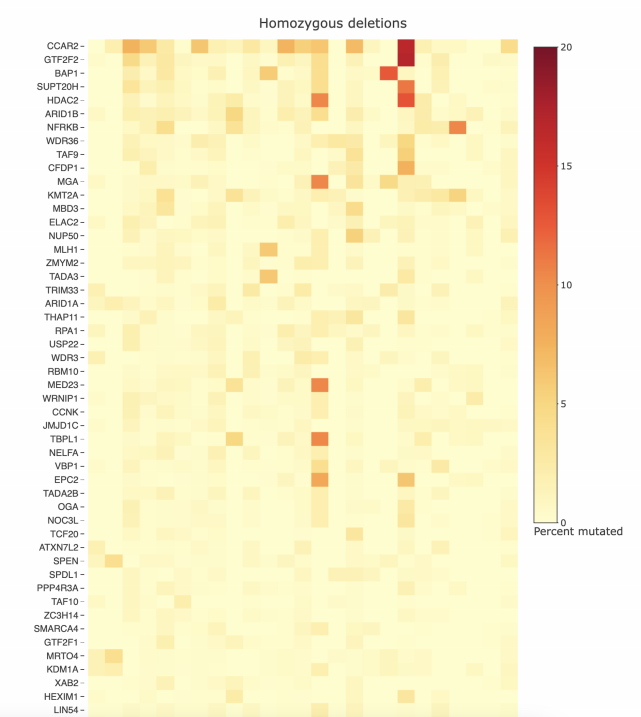
肿瘤畸变频率
· 通过拷贝数改变或点突变/INDEL,查询集中的基因在肿瘤样本中的突变程度如 何?
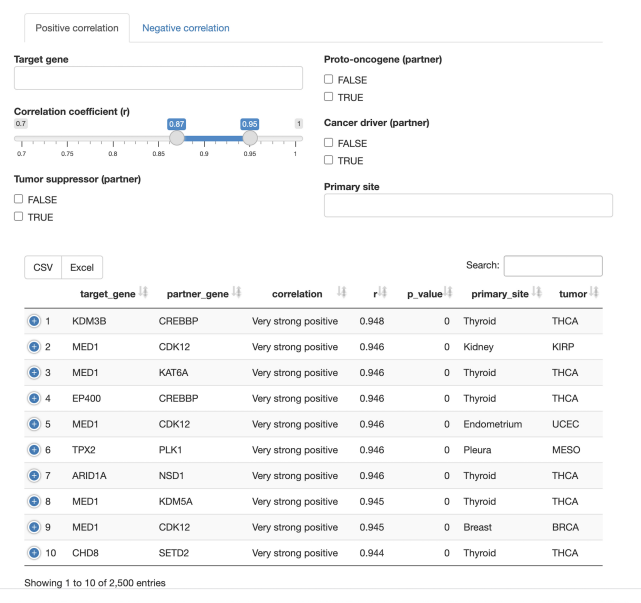
肿瘤共表达
· 在肿瘤样本中,查询集的成员是否与其他癌症基因共表达?
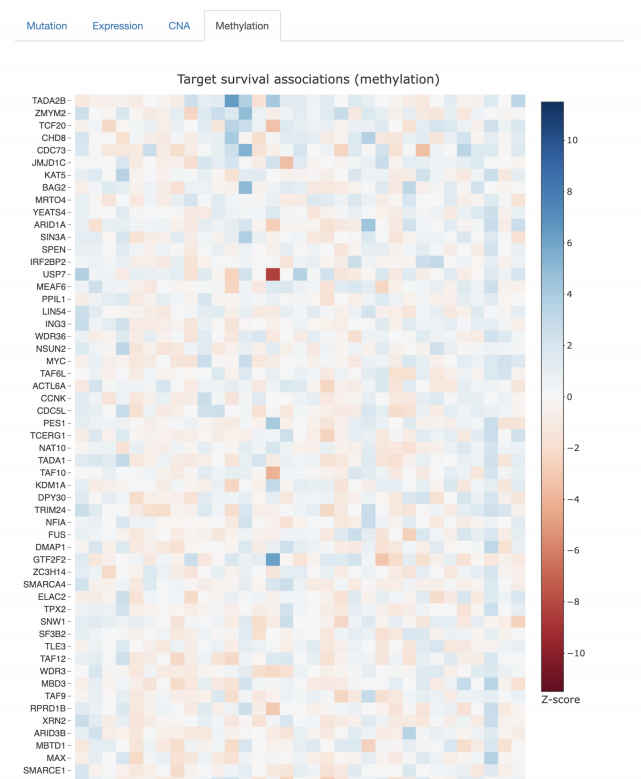
预后关联
· 考虑到肿瘤样本中查询基因的表达或甲基化水平或突变状态,哪些基因与癌症患者 的生存有关?
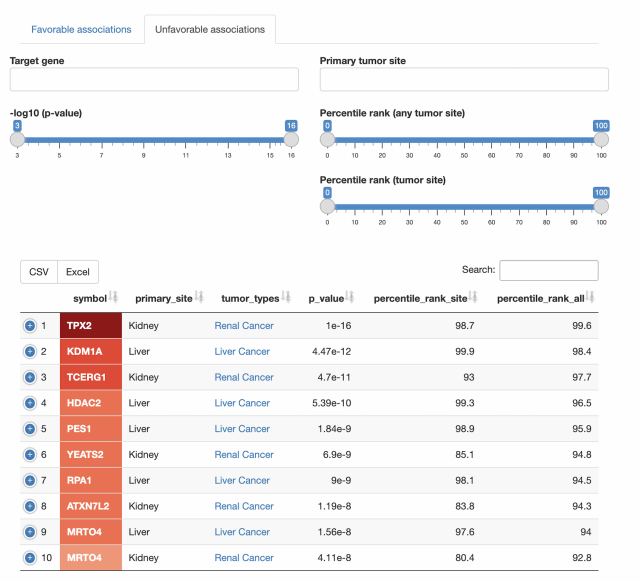
Excel 工作簿
· Excel 工作簿包含所进行的各种分析的所有结果,这些结果被组织到多个工作表中 (如下面屏幕截图中的红色圆圈所示)

四 、示例代码
library(oncoEnrichR)
myc_interact_targets <-
read.csv(system.file(“extdata”,”myc_data.csv”, package = “oncoEnrichR”), stringsAsFactors = F)
oeDB <- oncoEnrichR::load_db(cache_dir = “LOCAL_FOLDER”) myc_report <- oncoEnrichR::onco_enrich(query =
myc_interact_targets$symbol, oeDB = oeDB, show_cell_tissue = T, project_title = “cMYC_BioID_screen”, project_owner = “Raught et al.”)
oncoEnrichR::write(report = myc_report, oeDB = oeDB, file =
“LOCAL_FOLDER/myc_report_oncoenrichr.html”, format = “html”) oncoEnrichR::write(report = myc_report, oeDB = oeDB, file =
“LOCAL_FOLDER/myc_report_oncoenrichr.xlsx”, format = “excel”)
今天小果给大家带来了oncoEnrichR,不知道小伙伴们有没有收获呢?快运行代码亲自体会 吧~
五 、参考文献
Sigve Nakken, Sveinung Gundersen, Fabian L. M. Bernal, Dimitris Polychronopoulos, Eivind Hovig, and Jørgen Wesche. Comprehensive interrogation of gene lists from genome-scale cancer screens with oncoEnrichR (2023). Int J Cancer.