1、随机森林简介:
”三个臭皮匠顶上一个诸葛亮“非常适合形容随机森林。小果在这里简单通俗的说一下,随机森林就是通过多个决策树模型(森林)组成的集成算法,决策树各不相同,是随机从样本中抽取一部分组成的,每个决策树虽然是一个弱的分类器,都把他们最优的部分集成在一块,就形成了一个强分类器。
2、随机森林的作用:
就我们生物信息学而言,随机森林在分类(二分类)中发挥着十分重要的作用。例如,大家想寻找一些在癌症中发挥重要功能的基因,只要有两组数据,比如这些基因在癌症和癌旁的表达数据就可以进行分析,小果就可以帮大家实现;再比如大家想找出一些在细胞增殖、分化过程中比较重要的因子,只要有这些因子在这两个时期的表达数据,小果也可以帮大家实现!!这么好用的工具,还等什么,赶快跟着小果学起来吧!!!
3、随机森林具体操作代码
rm(list = ls())
library(“randomForest”)
########设置一个种子
set.seed(1243)
#################读取数据
data = read.csv(“lncRNAdatas.csv”,header = T,row.names = 1)
#############数据格式

########随机森林代码读入,并绘制随机森林图片
rf = randomForest(as.factor(data$Group)~.,data=data,importance=T,proximity=T,ntree=500)
plot(rf,main=”Romdom Forest”,lwd=2)
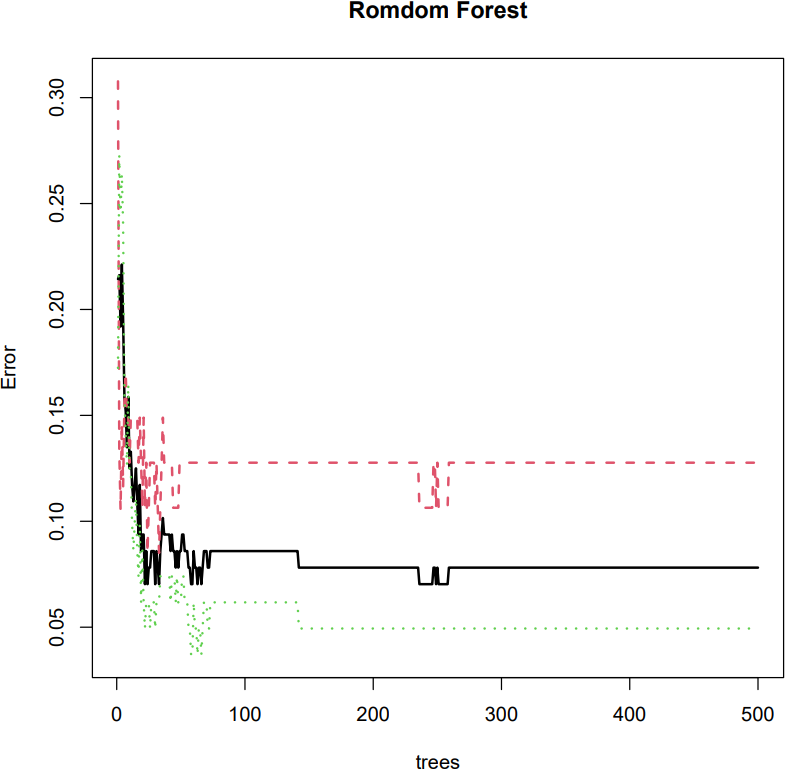
#########寻找误差最小的trees
optionTrees = which.min(rf$err.rate[,1])
rf2 = randomForest(as.factor(data$Group)~.,data=data,importance=T,proximity=T,ntree= optionTrees)
#########查看基因重要性,并绘图
importance = importance(x=rf2)
varImpPlot(rf2,main=””)
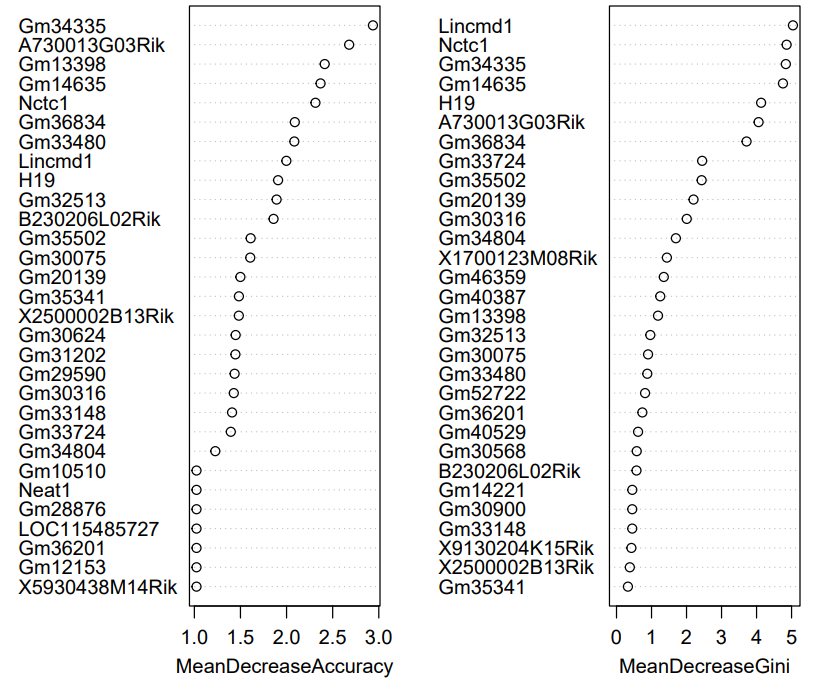
看完小果的代码之后,是不是觉得特别简单!赶快行动起来吧!!如果有什么不明白的地方,欢迎来和小果讨论哦!!!