Hello,大家好,小果又跟大家见面啦!
大家对Hi-C了解多少呢?这里小果首先给大家简单介绍一下Hi-C。Hi-C是一种用于测量基因组中不同位置之间的三维相互作用的技术。它可以用于研究基因组的结构和功能,比如基因调控、染色体域、环状结构等。
Hi-C的原理如下图所示
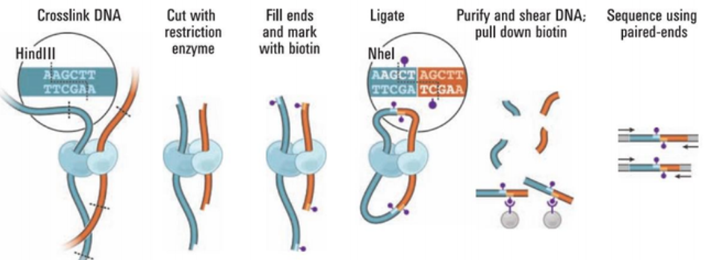
Hi-C的原理是基于染色质连接捕获(3C)的方法,它利用形式甲醛将空间上相邻的染色质片段交联起来,然后用限制性内切酶将交联的染色质切割成小片段,再用连接酶将切割后的片段连接起来,形成嵌合的连接产物。Hi-C的特点是它可以全面地测量基因组中任意两个位置之间的相互作用,而不是局限于特定的区域或目标。它通过在连接处引入生物素标记的核苷酸,然后用亲和纯化的方法,将含有连接处的DNA片段富集并进行深度测序。Hi-C的结果是一个相互作用矩阵,其中每个元素表示两个染色质片段之间的联系频率,也就是它们在空间上相互接近的概率。联系频率可以反映染色质片段之间的距离和紧密程度,从而揭示基因组的三维结构特征。
那么今天小果给大家分享一个处理Hi-C十分方便的软件:juicer
juicer是一个处理Hi-C数据的软件,是一种用于分析千碱基分辨率的Hi-C数据的平台。
juicer包括了一个从fastq原始数据文件生成Hi-C图的流程,以及一些用于在Hi-C图上进行特征注释的命令行工具。它可以用于研究基因组的三维结构和功能。
juicer的流程主要包括以下几个步骤:
- 对fastq文件进行质量控制和过滤,去除低质量的读段和重复的读段。
- 对过滤后的读段进行比对,将它们映射到参考基因组上,并生成sam或bam格式的文件。
- 对比对后的文件进行后处理,将它们分割成不同的染色体,并生成hic格式的文件。
- 对hic文件进行归一化,消除系统性偏差,并生成kr或vc格式的文件。
- 对归一化后的文件进行分析,提取感兴趣的特征,比如拓扑结合域、环状结构、相互作用矩阵等。
那么小果教大家如何使用juicer去处理Hi-C数据,这里带大家生成hic文件,生成hic文件之后大家可以根据自己的想法来做一些分析,例如AB区室划分,TAD分析等。Juicer官网区分了各种使用场景,这里小果就默认大家是CPU版本了嗷,详细的细节大家可以去看juicer的wiki嗷:https://github.com/aidenlab/juicer/wiki
juicer的脚本(juicer.sh)大概处理流程是这样的:
文件输入->bwa比对->排序->合并->去除PCR重复->生成hic文件
上面的流程中需要用到bwa比对软件,所以没安装的小伙伴记得使用之前安装bwa哦
下面是小果的代码:
#创建工作目录,并下载juicer
mkdir ./opt
cd opt
git clone https://github.com/theaidenlab/juicer.git
# 创建软连接
ln -s juicer/CPU scripts
cd scripts/common
wget http://hicfiles.tc4ga.com.s3.amazonaws.com/public/juicer/juicer_tools.1.7.6_jcuda.0.8.jar
ln -s juicer_tools.1.7.6_jcuda.0.8.jar juicer_tools.jar
cd ../..
# 参考基因组建立索引,这里小果使用的是hg38的参考基因组
mkdir references
cp YOUR_hg38_PATH/hg38.fasta references/
bwa index hg38.fasta
cd ..
# 添加限制性内切酶位点信息,小伙伴们注意自己的是不是 MboI 酶哦
mkdir restriction_sites
cd restriction_sites/
# 生成 hg38_MboI.txt 文件
python ../juicer/misc/generate_site_positions.py MboI hg38_MboI ../references/hg38.fasta
# 生成chrom.size文件
awk ‘BEGIN{OFS=”\t”}{print $1, $NF}’ hg38_MboI.txt > hg38.chrom.sizes
cd ..
# 添加 fastq 文件,小果采用是官方的测试数据集
mkdir fastq && cd fastq
wget http://juicerawsmirror.s3.amazonaws.com/opt/juicer/work/HIC003/fastq/HIC003_S2_L001_R1_001.fastq.gz
wget http://juicerawsmirror.s3.amazonaws.com/opt/juicer/work/HIC003/fastq/HIC003_S2_L001_R2_001.fastq.gz
cd ..
# 运行 juicer
scripts/juicer.sh -D YOUR_juicer_PATH -y restriction_sites/hg38_MboI.txt -z references/hg38.fasta -p restriction_sites/hg38.chrom.sizes -s MboI
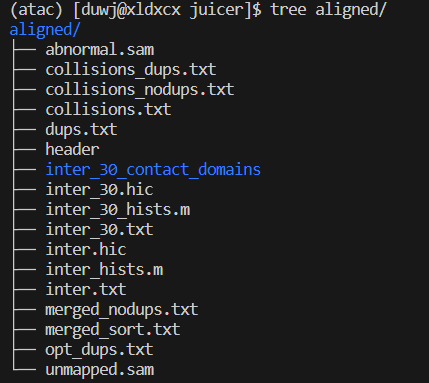
最后生成的结果文件生成在aligned文件夹中,流程中生成的文件在splits文件夹中(例如比对生成的sam文件),aligned文件夹中中就有大家最关注的hic文件啦,大家仔细看会发现有两个hic文件,其中的inter_30.hic是设置了mapQ threshold > 30过滤后的结果。
hic文件的格式大家可以看下面的链接:
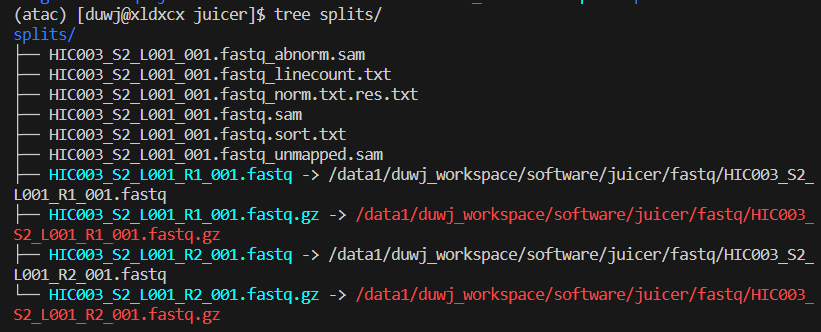
小果的splits文件夹是这样的:
小果的aligned文件夹是这样的:
今天的关于juicer软件的学习就到这里啦,感兴趣的小伙伴可以找小果讨论,有感觉生信分析复杂的小伙伴可以直接使用我们的生信小工具哦,链接在这:http://www.biocloudservice.com/home.html,我们明天见咯~
强力推荐!Juicer处理Hi-C数据
Hello,大家好,小果又跟大家见面啦!
大家对Hi-C了解多少呢?这里小果首先给大家简单介绍一下Hi-C。Hi-C是一种用于测量基因组中不同位置之间的三维相互作用的技术。它可以用于研究基因组的结构和功能,比如基因调控、染色体域、环状结构等。
Hi-C的原理如下图所示

Hi-C的原理是基于染色质连接捕获(3C)的方法,它利用形式甲醛将空间上相邻的染色质片段交联起来,然后用限制性内切酶将交联的染色质切割成小片段,再用连接酶将切割后的片段连接起来,形成嵌合的连接产物。Hi-C的特点是它可以全面地测量基因组中任意两个位置之间的相互作用,而不是局限于特定的区域或目标。它通过在连接处引入生物素标记的核苷酸,然后用亲和纯化的方法,将含有连接处的DNA片段富集并进行深度测序。Hi-C的结果是一个相互作用矩阵,其中每个元素表示两个染色质片段之间的联系频率,也就是它们在空间上相互接近的概率。联系频率可以反映染色质片段之间的距离和紧密程度,从而揭示基因组的三维结构特征。
那么今天小果给大家分享一个处理Hi-C十分方便的软件:juicer
juicer是一个处理Hi-C数据的软件,是一种用于分析千碱基分辨率的Hi-C数据的平台。
juicer包括了一个从fastq原始数据文件生成Hi-C图的流程,以及一些用于在Hi-C图上进行特征注释的命令行工具。它可以用于研究基因组的三维结构和功能。
juicer的流程主要包括以下几个步骤:
- 对fastq文件进行质量控制和过滤,去除低质量的读段和重复的读段。
- 对过滤后的读段进行比对,将它们映射到参考基因组上,并生成sam或bam格式的文件。
- 对比对后的文件进行后处理,将它们分割成不同的染色体,并生成hic格式的文件。
- 对hic文件进行归一化,消除系统性偏差,并生成kr或vc格式的文件。
- 对归一化后的文件进行分析,提取感兴趣的特征,比如拓扑结合域、环状结构、相互作用矩阵等。
那么小果教大家如何使用juicer去处理Hi-C数据,这里带大家生成hic文件,生成hic文件之后大家可以根据自己的想法来做一些分析,例如AB区室划分,TAD分析等。Juicer官网区分了各种使用场景,这里小果就默认大家是CPU版本了嗷,详细的细节大家可以去看juicer的wiki嗷:https://github.com/aidenlab/juicer/wiki
juicer的脚本(juicer.sh)大概处理流程是这样的:
文件输入->bwa比对->排序->合并->去除PCR重复->生成hic文件
上面的流程中需要用到bwa比对软件,所以没安装的小伙伴记得使用之前安装bwa哦
下面是小果的代码:
#创建工作目录,并下载juicer
mkdir ./opt
cd opt
git clone https://github.com/theaidenlab/juicer.git
# 创建软连接
ln -s juicer/CPU scripts
cd scripts/common
wget http://hicfiles.tc4ga.com.s3.amazonaws.com/public/juicer/juicer_tools.1.7.6_jcuda.0.8.jar
ln -s juicer_tools.1.7.6_jcuda.0.8.jar juicer_tools.jar
cd ../..
# 参考基因组建立索引,这里小果使用的是hg38的参考基因组
mkdir references
cp YOUR_hg38_PATH/hg38.fasta references/
bwa index hg38.fasta
cd ..
# 添加限制性内切酶位点信息,小伙伴们注意自己的是不是 MboI 酶哦
mkdir restriction_sites
cd restriction_sites/
# 生成 hg38_MboI.txt 文件
python ../juicer/misc/generate_site_positions.py MboI hg38_MboI ../references/hg38.fasta
# 生成chrom.size文件
awk ‘BEGIN{OFS=”\t”}{print $1, $NF}’ hg38_MboI.txt > hg38.chrom.sizes
cd ..
# 添加 fastq 文件,小果采用是官方的测试数据集
mkdir fastq && cd fastq
wget http://juicerawsmirror.s3.amazonaws.com/opt/juicer/work/HIC003/fastq/HIC003_S2_L001_R1_001.fastq.gz
wget http://juicerawsmirror.s3.amazonaws.com/opt/juicer/work/HIC003/fastq/HIC003_S2_L001_R2_001.fastq.gz
cd ..
# 运行 juicer
scripts/juicer.sh -D YOUR_juicer_PATH -y restriction_sites/hg38_MboI.txt -z references/hg38.fasta -p restriction_sites/hg38.chrom.sizes -s MboI
最后生成的结果文件生成在aligned文件夹中,流程中生成的文件在splits文件夹中(例如比对生成的sam文件),aligned文件夹中中就有大家最关注的hic文件啦,大家仔细看会发现有两个hic文件,其中的inter_30.hic是设置了mapQ threshold > 30过滤后的结果。
hic文件的格式大家可以看下面的链接:
https://github.com/aidenlab/hic-format/blob/master/HiCFormatV9.md
小果的splits文件夹是这样的:

小果的aligned文件夹是这样的:

今天的关于juicer软件的学习就到这里啦,感兴趣的小伙伴可以找小果讨论,有感觉生信分析复杂的小伙伴可以直接使用我们的生信小工具哦,链接在这:http://www.biocloudservice.com/home.html,我们明天见咯~