GWAS分析时,拿到基因型数据,拿到表型数据时,首先应该想到要对基因型和表型数据的质量进行检测和控制。然后再考虑这组数据该怎么分析,如何分析。基因型和表型的质控是非常重要的,如果数据的质量不佳,那么计算的结果的有效性和准确性就无法保证。即使后续分析的非常详细,最后的结果也还是不理想的,相当于做了无用功。所以对基因型和表型质控是至关重要的。那么如何对数据质控呢,需要对那些指标进行指控呢?这都是需要注意的。今天和小果学习一下基因组数据如何清洗。
首先介绍一下需要对那些指标进行质控。
1.个体基因分型检出率(call rate)
样本检出率:是指对于某种样本而言,通过测序并成功判刑的snp与所有检出的snp的比值,通常标准在90%或以上。
- 单个位点基因分型检出率(call rate)
snp检出率:指对某个snp位点被成功检测到的样本与所有样本比值,一般要求在90%或以上。
最小等位基因频率(Minor Allele Frequency,MAF)
最小等位基因频率:对那些MAF较小的snp,能得到信息量较少而且目前对这些snp检出效率也不高,通常要求maf值在5%及以上。
哈代温伯格平衡 (Hardy-wenberg Epuilibrium,HWE)
哈代温伯格平衡:HWE有助于确定哪些有明显基因分型错误的snp,因此一般要求位点snp符合HWE.
在PLINK中做这几步的质量控制,可以直接使用命令+参数完成筛选,也可以使用PLINK生成检测结果来手动筛选。当然,一般对数据要求严格的实验都是先生成结果,经过查看以后确定参数再使用PLINK进行筛选。
具体来讲,根据小果的习惯一般先进行个体和SNP的筛选,次等位基因频率、哈代平衡的检测,再做一遍上述的筛选。
对个体和SNP的检测和筛选
–missing 生成个体和SNP缺失的检测结果。
个体缺失位点结果文件为plink.imiss:
第一列为家系ID,第二列为个体ID,第三列是否表型缺失,第四列缺失的SNP个数,第五列总SNP个数,第六列缺失率。

单个SNP缺失的结果文件为plink.lmiss:
第一列为染色体,第二列为SNP名称,第三列为缺失个数,第四列为总个数,第五列为缺失率
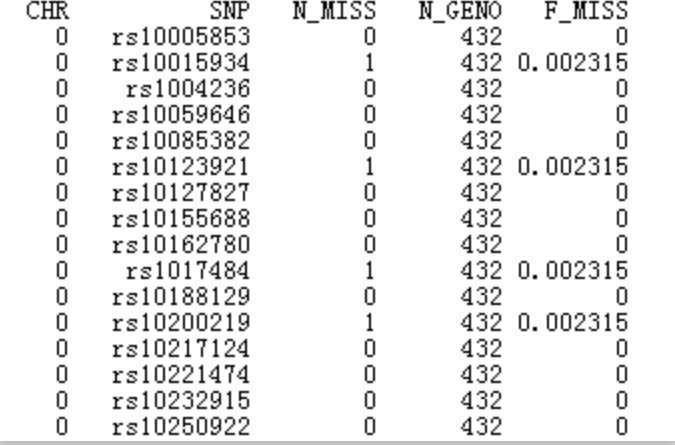
哈代温伯格平衡
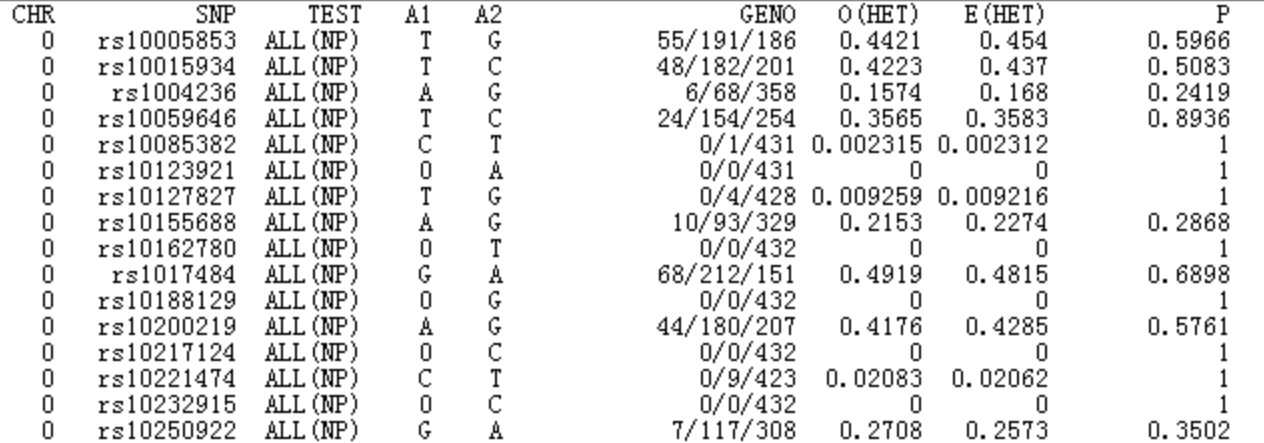
–hardy 计算所有位点的哈温检测结果
结果文件为plink.hwe:
第一列为染色体,第二列为SNP的ID,第三列为TEST类型,第四列A1为 minor 位点,第五列A2为 major 位点,第六列为基因型分布,对应A1A1, A1A2, A2A2的个数,第六列为观测杂合度频率,第七列为期望杂合度频率,第八列为哈温平衡的卡方检验P值。
次等位基因频率
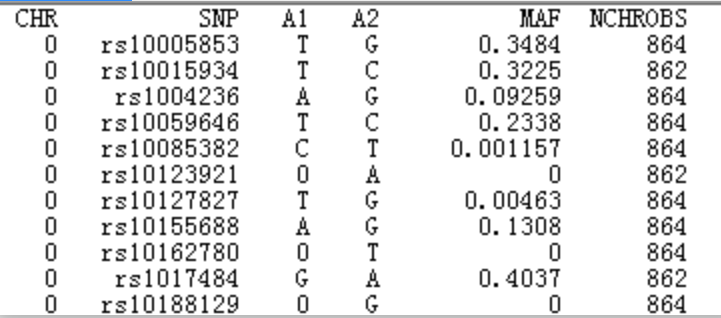
–freq 计算每个SNP位点的次基因频率
结果文件为plink.frq:
第一列为染色体号,第二列为SNP ID,第三列为 minor 位点,第四列为 major 位点,第五列为次等位基因频率,第六列为等位基因观察数。
知道了检测的结果一切就好办了,接下来介绍一下各个指标的质控方法(利用PLINK软件)
call rate >90%
plink –file 1-data-holstein –geno 0.01 –recode –out 2-data-holstein_qc ###这一步就是要求snp检出率要在90%以上
plink –file 1-data-holstein –mind 0.01 –recode –out 3-data-holstein_qc ##这步就是对样本检出率进行质控,控制在90%以上
MAF>0.05
plink –file 1-data-holstein –maf 0.05 –recode –out 4-data-holstein_qc ##这步是对最小等位基因频率进行质控,要求要大于0.05.
HWE < 0.00001
plink –file 1-data-holstein –hwe 0.00001 –recode –out 5-data-holstein_qc ####此步要求哈代温伯格平衡要小于0.00001。
好了,看了上述的质控流程,基本上就能解决问题了,不过一步一步的来很繁琐,可以把所有的过程合并到一起例如:
plink –file 1-data-holstein –mind 0.1 –geno 0.1 –maf 0.05 –hwe 0.00001 –recode –out 2-data-holstein_qc #这条稍微长一点的命令就把所有指标的全部都包含了可以一步就出来质控结果。
在生成的日志文件中可以查看质控结果。

上述就是基因型和表型质控的内容,请结合自己的数据情况选择质控指标和质控指标的阈值。学完了这款软件是不是想马上操作一番,大家可以测试一下,让知识更加牢固。
后面小果将给小伙伴们继续分享PLINK软件进行GWAS分析操作流程,如有需要继续关注生信果和云生信生物信息学平台( http://www.biocloudservice.com/home.html)