大家好,小果又来啦,大家还记得motif是什么吗?思考三秒钟,如果你还记得的话,那小果给你大拇哥!
下面小果给大家详细介绍下motif:motif是指一段具有特定功能或结构的分子序列或元素,通常是保守的、重复的、与其他分子相互作用的。Motif可以存在于DNA、RNA或蛋白质中,例如转录因子的结合位点、RNA酶的活性中心、蛋白质的结构域等。Motif可以用序列logo图、一致性序列或PFM矩阵等方式来表示和分析。Motif的研究可以帮助我们理解基因调控、分子进化、功能预测等生物学问题。
序列基序在基因调控分析中越来越重要。Motif并不局限于基因组DNA序列还是RNA序列,甚至蛋白质序列也可以提取出相应的motif。当某一个大类的序列中,大量重复出现相同的序列结构的时候,就存在探索它意义的价值。基于motif序列的提取,我们可以预测潜在的结合位点等等,有助于我们进一步理解各生物学过程中涉及的生物学意义。
看到这,大家是不是对motif充满了好奇呢
大家经常看到的motif的logo图如下图所示

那怎么去理解motif的logo图呢?小果来告诉大家:motif的logo图经常用于描述序列特征,如DNA中的蛋白质结合位点等。motif的logo图由每个位置的一堆字母组成。字母的相对大小表示它们在序列中的频率。字母高的就说明该位置出现这个碱基的频率更大。
那么如何使用homer去预测motif呢?下面小果带大家一探究竟。
首先说一下用到的软件HOMER,HOMER 是一套用于Motif查找和二代数据分析的工具。HOMER 中的工具是使用Perl 和C++编写的,是Linux command line based。HOMER 这个软件是一个大杂烩,能解决几乎所有的高通量测序数据的分析。
HOMER 一个常用的motif分析软件。它通过比较两个序列集,并使用ZOOPS scoring和超几何分布(或者负二项分布)进行motif的富集分析。它主要用于ChIP-seq和ATAC-seq和promoter分析,但也可以用于核酸序列的motif分析问题。
HOMER软件可以进行多种类型的motif分析,如 promoter motif analysis ,基因组位置motif分析(ChIP-seq分析中的motif分析),利用自定义的fasta文件进行motif分析,RNA序列的motif分析(分析CLIP-seq数据中的RNA binding elements)。
这是HOMER软件的官网地址:
http://homer.ucsd.edu/homer/ngs/peakMotifs.html,大家一定要养成多看官方文档的好习惯嗷
HOMER的安装可以使用conda来完成:
conda install -c bioconda homer
下面需要configureHomer.pl完成Homer软件的配置:
- 先下载configureHomer.pl:
wget http://homer.ucsd.edu/homer/configureHomer.pl
- 使用configureHomer.pl配置Homer(小伙伴们注意文件路径问题嗷)
perl configureHomer.pl [options]
大家可以使用configureHomer.pl来配置Homer,例如下载人类,小鼠的参考基因组等,人和小鼠的研究居多,这里只以这两者举例
# 下载 hg19 人的参考基因组,将hg19替换为mm10可下载mm10 小鼠的参考基因组
perl configureHomer.pl -install hg19
不过这里小果研究的是非模式生物,那么小果就带大家一起用可爱的家蚕来学习HOMER软件吧
首先小果看网上小伙伴说使用本地的参看基因组直接命令行使用就行,可把小果害苦了,一直error!不过不慌,小果debug是最强的,小果又查到是因为参考基因组的问题,看了看homer的版本:

小果通过查阅官网发现从homer v4.4 开始就可以自定义homer了,所以我们需要使用loadGenome.pl或loadPromoters.pl命令创建自己的基因组和启动子 FASTA 或注释文件
查阅帮助文档发现:
Required Parameters:
-name <genome name> (i.e. hg19, tair10, etc.)
-fasta <genome fasta file> (Single genome sequence, preferrabley soft masked, unzipped)
-gtf <gene annotation file> (Transcript annotation in gtf format, -gff/-gff3 to use them)
-org <organism name, ok to use ‘null’>
所以我们需要参考基因组fasta文件和gtf文件
下面是小果的代码:
loadGenome.pl -gtf test.gtf -name test -fasta test_assembly.fa -org null
下面就是预测motif的过程了,以CHIP-seq流程为例,输入文件可以是MACS2软件callpeak的bed文件(可以直接使用)或者是HOMER软件指定的peak文件格式。peak文件格式:使用Tab分隔,共五列,分别是 peak ID , chr , start , end ,strand
下面是小果的代码:
findMotifsGenome.pl test.bed test sample_motif_dir -len 8,10,12
参数的解释如下:
test.bed : bed格式文件
test : 自定义的参考基因组(loadGenome.pl命令中的name参数)
sample_motif_dir : 输出文件夹
-len :motif的长度,默认8,10,12,越大越消耗计算资源
HOMER的输出结果中有名为homerResults.html的网页文件,在里面可以详细的看到motif的结果,下面是小果的结果
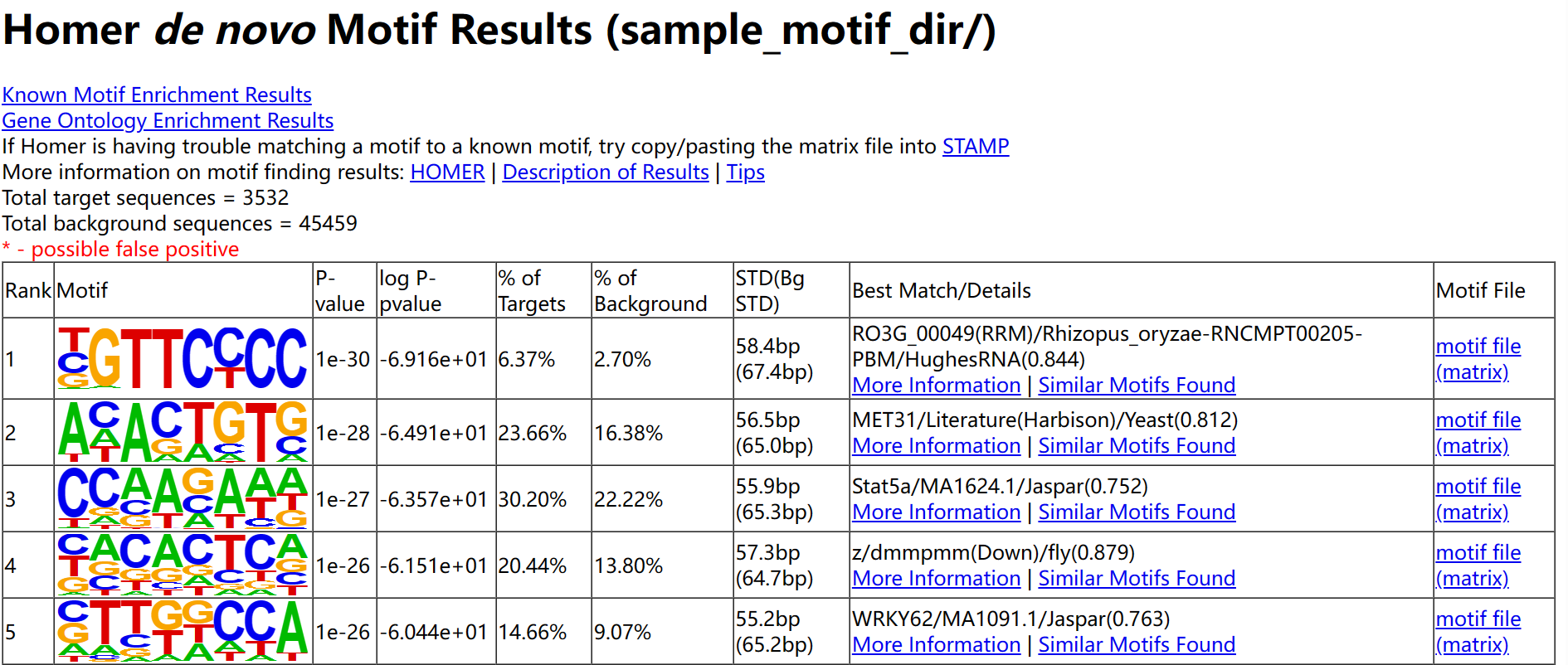
输出结果按照p-value排序,最后一列是一个链接到motif文件的超链接,可以从这个文件中找到包含此motif的其他序列。在Best Match/Details 列中,HOMER展示与denovo motif最匹配的已知motif。
今天的关于homer预测motif的学习就到这里啦,感兴趣的小伙伴可以找小果讨论,有感觉生信分析复杂的小伙伴可以直接使用我们的生信小工具哦,链接在这:http://www.biocloudservice.com/home.html,我们明天见咯~