hello,你知道如何使用edgR包来对RNA-seq数据进行基因表达差异分析嘛?今天小果就来叫你如何操作,感兴趣的话就和小果一起看下去吧!
什么是edgR?
edgeR包主要用于处理RNA-seq或其他高通量测序技术生成的数字化基因表达数据。它提供了一套统计学方法和工具,用于鉴定和分析不同条件下基因表达的差异。edgeR包被广泛应用于生物医学研究和生物信息学领域~
以下是edgeR包的一些主要功能和特点:
- 高度精确的差异表达分析:edgeR使用负二项分布模型和泊松分布模型来准确地估计基因表达的离散度,并计算差异表达的显著性统计量。
- 优化的统计方法:edgeR提供了针对不同实验设计和数据类型的优化统计方法。
- 数据预处理和标准化:edgeR包提供了常用的数据预处理和标准化方法,如去除低表达基因、消除库大小差异和标准化样本间技术变异等。
- 灵活的结果解读和可视化:edgeR包提供了一系列函数和图形工具,用于解读和可视化差异表达结果。
- 高度可扩展性:edgeR可以处理包含数百万个基因和数十个样本的大规模数据集。
edgR分析基因表达数据
- 读取数据
这里我们提供的数据为”GSE168466_series_matrix.txt”,小果后面也会附上该表达矩阵的具体文件,当然也可以导入你自己感兴趣的数据哦~
library(edgeR)
x <- read.table(“GSE168466_series_matrix.txt”, header = TRUE, sep = “\t”, check.names = FALSE)
- 提取表达矩阵
从读取的数据中提取表达矩阵。基因表达数据的表达矩阵是以基因为行,样本为列的形式,相信这一点大家都已经熟悉~
rawdata <- as.matrix(x[, -1])
- 检查和处理缺失值
对于数据中的缺失值,可以选择移除包含缺失值的行或列。
# 检查是否存在缺失值
anyNA(rawdata)
# 移除包含缺失值的行或列
rawdata <- na.omit(rawdata)
我们来一起看下最后处理后的表达矩阵吧!
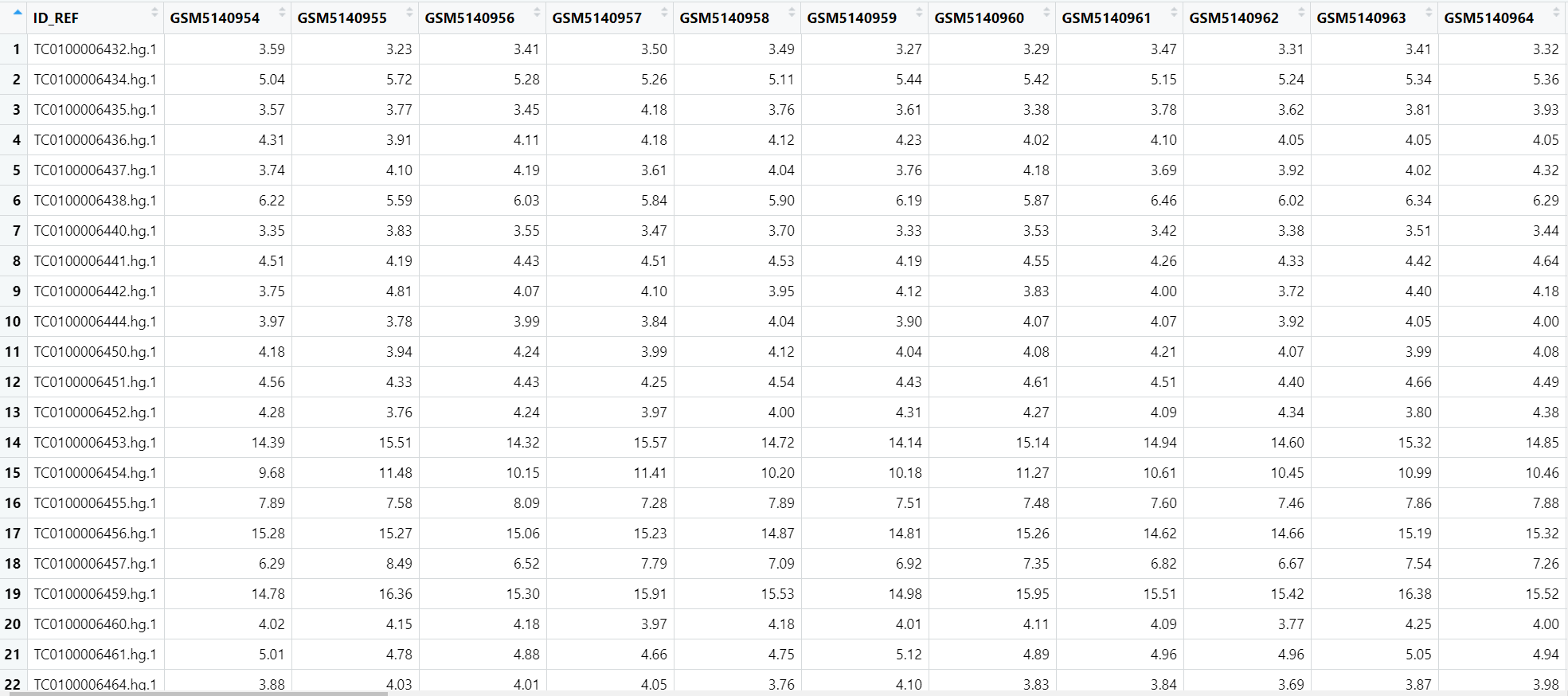
- 设计分组信息
根据实验设计,为每个样本分配一个组别标签。这些标签将用于后续的差异分析。例如,在你的示例中,将”CK”和”Treat”两个分组分别分配给49个样本。
group <- factor(c(rep(“CK”, 49), rep(“Treat”, 49)))
注意:一定要确保分组总数和表达矩阵总列数一致哦!小果的数据集中共有98个样本,所以分别设置两个49个样本的分组:

- 创建DGEList对象
用 DGEList() 函数创建一个 DGEList 对象 y,其中包括原始计数数据 rawdata、基因名称(从行名中提取)和分组信息 group。
y <- DGEList(counts = rawdata, genes = rownames(rawdata), group = group)
- 过滤表达矩阵
如果有需要,可以根据一定的过滤条件来选取感兴趣的基因哦:
# 使用cpm函数计算每个基因的读数,然后选择大于等于1的基因
keep <- rowSums(cpm(y) > 1) >= 1
# 根据过滤条件对表达矩阵进行子集操作
y <- y[keep, , keep.lib.sizes = FALSE]
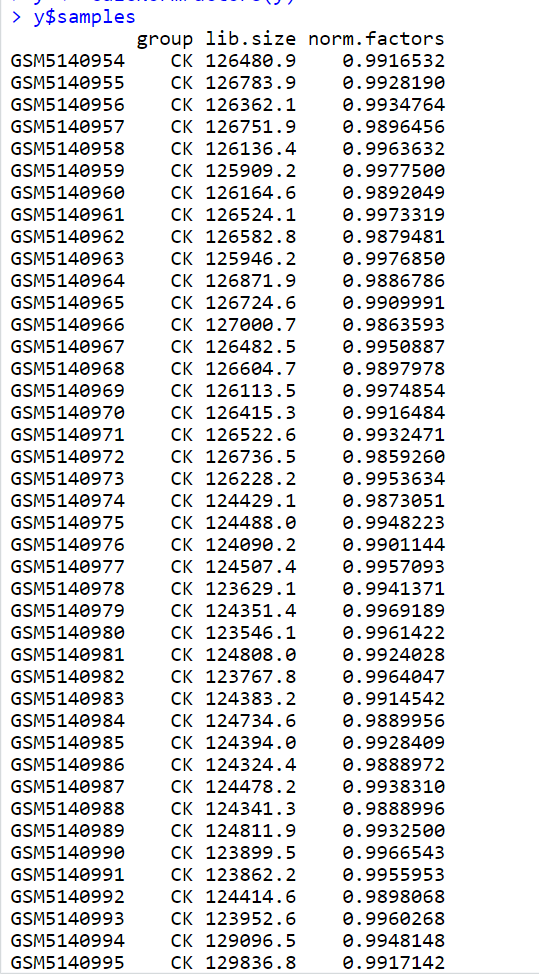
- 数据标准化
接下来,我们需要对表达矩阵进行标准化以消除样本间的技术差异。edgeR提供了TMM(trimmed mean of M-values)方法进行标准化。
y <- calcNormFactors(y)
y$samples
- 差异分析
我们在这里使用exactTest()函数进行差异分析。可以通过指定推测的离散度(bcv)来控制对差异的敏感性。较高的bcv值适用于更保守的差异检测,反之亦然。
###推测离散度,若样本是人,设置bcv = 0.4,模式生物设置0.1(此处是根据有相关教程进行设置,也可以你根据你的结果自行设置,最终得到你的理想值)
bcv <- 0.2
et <- exactTest(y, dispersion = bcv^2)
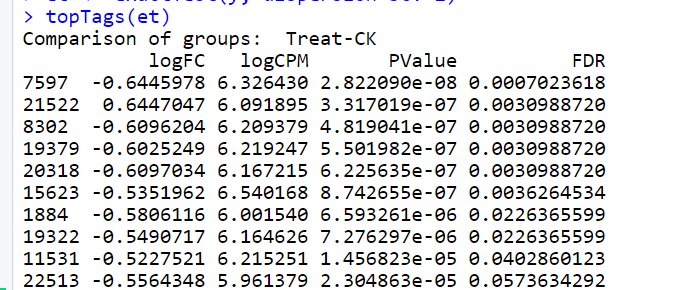
- 结果解读
使用topTags()和summary()函数来查看差异基因的结果统计摘要:
results <- topTags(et)
print(results)
summary(de <- decideTestsDGE(et))
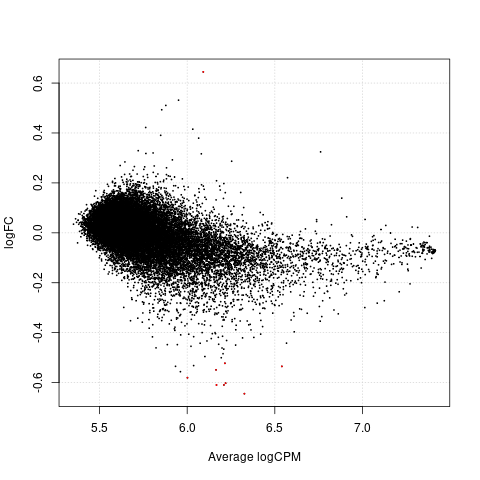
- 结果可视化
使用plotSmear()函数绘制MA图,用于可视化差异表达结果。该图可以帮助我们直观地了解不同基因在样本之间的差异哦
detags <- rownames(y)[as.logical(de)]
png(‘0h_vs_2h_MAplot.png’)
plotSmear(et, de.tags = detags)
abline(h = c(-4, 4), col = “blue”)
dev.off()
接下来让我们一起来看看可视化后的结果吧!
以上教程就是使用edgeR包进行基因差异表达分析的简单步骤。根据你自己的研究问题和数据集,大家可能需要进一步了解和调整方法哦。希望能对你的差异表达分析提供一些帮助!怎么样,你学会了嘛!?