你知道如何使用clusterProfiler对DOSE包中的数据进行功能富集分析嘛?今天小果就来教你轻松完成富集分析的流程并通过不同的R包来实现可视化!感兴趣的话就一起看下去吧!
了解clusterProfiler
clusterProfiler包是用于更全面和灵活的基因功能富集分析的工具。它提供了比DOSE更多的功能和可定制选项,能够进行更深入的功能解释和分析。
主要功能有:
- 富集分析:clusterProfiler包支持多种生物信息学数据库(如GO、KEGG、Reactome)提供的注释数据,可以进行更广泛的功能富集分析。
- 可视化:clusterProfiler包提供了丰富的可视化功能,包括柱状图、散点图等,用于直观展示富集分析的结果,帮助研究人员更好地理解和解释数据。
- 分类和聚类:clusterProfiler包还提供了一系列分类和聚类功能,可以对基因列表进行分组和聚类,并进行富集分析,以发现不同基因子集之间的功能差异和相似性。
什么是DOSE包?
DOSE包主要用于基因功能富集分析,它能够通过比较输入的基因列表与已知的基因功能注释信息,找出在特定功能类别中富集的基因。这些功能类别包括Gene Ontology (GO)注释、KEGG通路、Reactome通路等。DOSE包提供了一系列函数,用于计算富集分析的结果,并生成易于理解和可视化的结果。
主要功能为:
- GO功能富集分析:DOSE包可以执行基于Gene Ontology的功能富集分析,包括三个方面的富集:生物过程(biological process)、分子功能(molecular function)和细胞组分(cellular component)。
- 通路富集分析:DOSE包还能进行基于KEGG和Reactome通路的富集分析,帮助研究人员了解基因在特定生物学路径中的作用和相互关联。
- 基因集合比较:DOSE包可以比较不同基因集合之间的差异,帮助研究人员确定哪些功能或通路在两个基因组之间有显著差异。
clusterProfiler富集分析
- 安装与导入R包
首先,我们需要安装并加载DOSE和clusterProfiler包:
install.packages(“DOSE”)
install.packages(“clusterProfiler”)
- 准备基因数据
在这个示例中,我们将使用DOSE包中附带的geneList数据集哦。使用以下命令加载数据集~
data(geneList, package = “DOSE”)
然后,选择前100个基因作为我们的分析对象:
![]() g_list <- names(geneList)[1:100]
g_list <- names(geneList)[1:100]
head(g_list) # 查看选择的基因列表的前6行
- 富集分析
接下来,我们将使用org.Hs.eg.db数据库进行功能富集分析。大家一定要确保已安装此数据库,然后再加载它哦~
library(“org.Hs.eg.db”)
好啦,现在我们可以我们使用以下代码执行GO富集分析,并将富集结果写入文件:
eG <- enrichGO(gene = g_list,
OrgDb = org.Hs.eg.db,
pvalueCutoff = 0.01,
qvalueCutoff = 0.01,
ont = “all”,
readable = TRUE)
write.table(eG, file = “eG.txt”, sep = “\t”, quote = FALSE, row.names = FALSE)
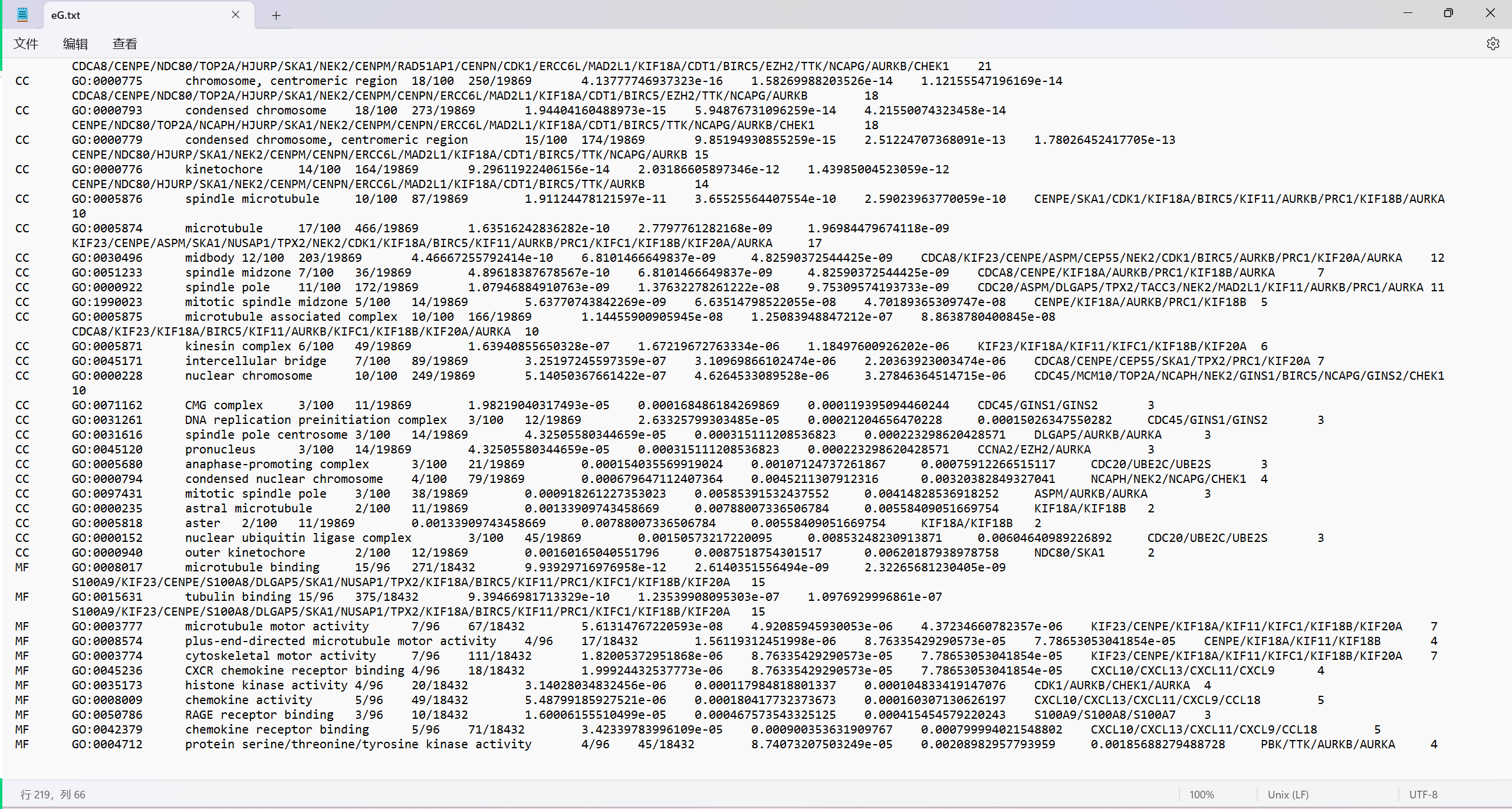
在这个例子中,小果设置了p值和q值的阈值为0.01,并选择了所有的功能富集类型。分析结束后将生成一个名为”eG.txt”的文本文件,其中包含我们的富集结果哦。我们一起来看看分析的结果吧!
多种方法可视化富集分析结果
接下来,小果要教大家使用enrichplot包对富集结果进行可视化:
- barplot
pdf(file = “eGO_barplot.pdf”, width = 8, height = 10)
barplot(eG, x = “GeneRatio”, color = “p.adjust”,
showCategory = 10,
split = “ONTOLOGY”) +
facet_grid(ONTOLOGY ~ ., scale = ‘free’)
dev.off()
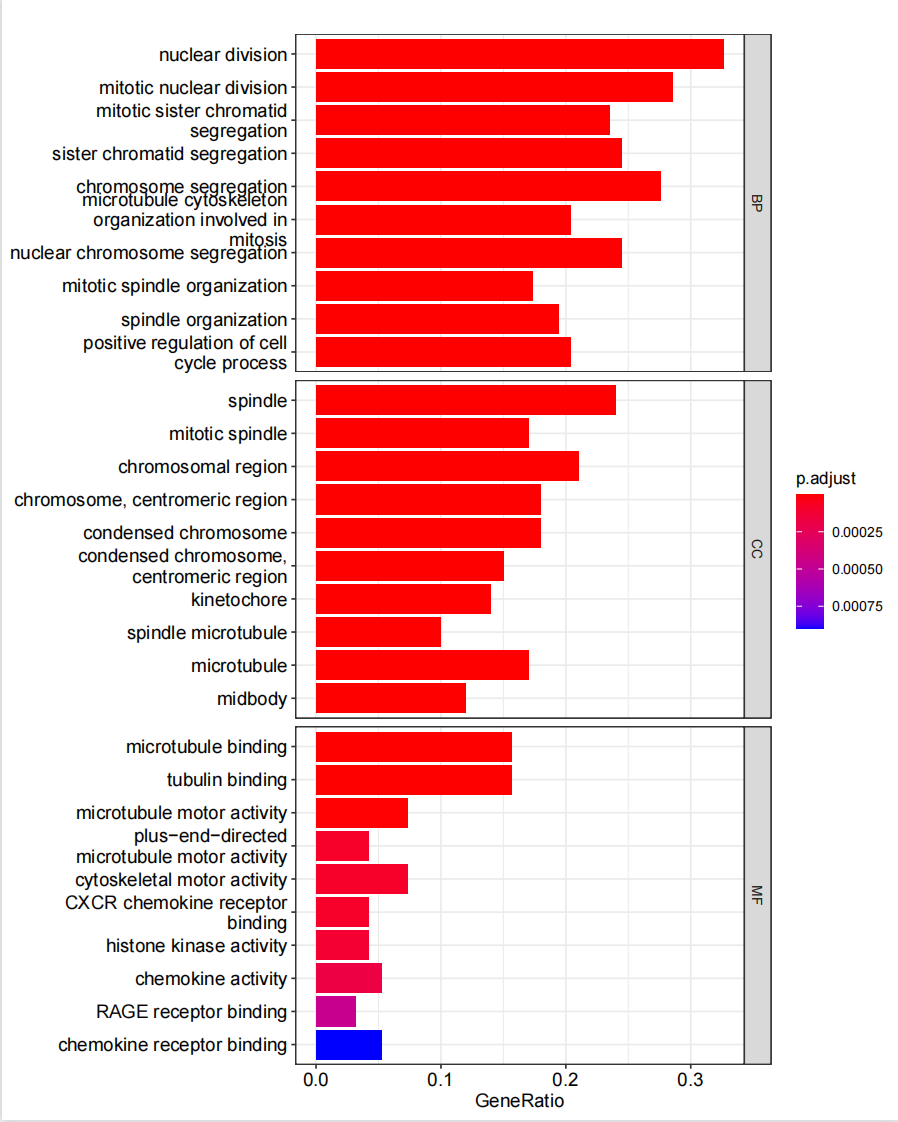
此时在工作文件夹中得到了pdf格式的GO富集绘图:,其中包含基于各功能类型的柱状图。每个类型中显示了前10个具有调整后p值颜色编码的基因比例。我们一起来看看效果吧!
- dotplot
dotplot(eG, x = “GeneRatio”, color = “p.adjust”, size = “Count”,
showCategory = 5,
split = “ONTOLOGY”) +
facet_grid(ONTOLOGY ~ ., scale = ‘free’)
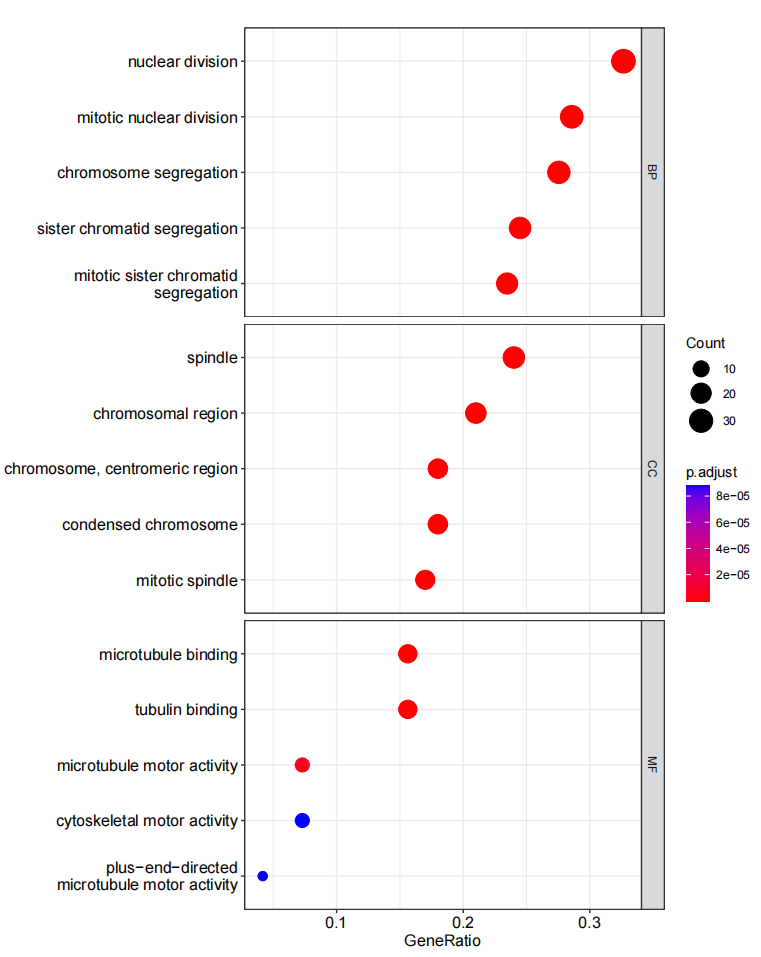
这将生成另一个散点图,其中包含基于各功能类型的点图。每个类型中显示了前5个基因比例,并根据调整后p值来设置点的颜色和大小。我们一起来看看效果:
通过以上步骤,你已经完成了基于DOSE和clusterProfiler的基因功能富集分析啦。大家可以根据的自己需求进一步调整和解释结果哦。怎么样,你学会了嘛!?