对于基因进化树的构建,大家应该都很苦恼吧。最近小果刚好需要构建进化树,在经过学习之后,也掌握了如何建树。那么现在就让小果来带大家一步步从序列数据的下载,再到基因进化树的建立,详细地手把手教学吧!
一、蛋白质序列数据的下载
在这里我们将在NCBI数据库中,搜寻Syp synaptophysin蛋白质的fastq序列作为数据准备。其中Synaptophysin(SYP)是一种神经元内高度保守的膜蛋白,它主要存在于突触小泡膜上,并参与突触传递和神经递质释放的过程。它被广泛用作神经末梢和神经内分泌肿瘤的免疫组化标记物,可以用于帮助鉴定和分类这些肿瘤。因此,在病理学和临床诊断中,鉴定SYP的表达水平可以提供有用的信息,对于诊断和治疗策略的制定具有重要意义。
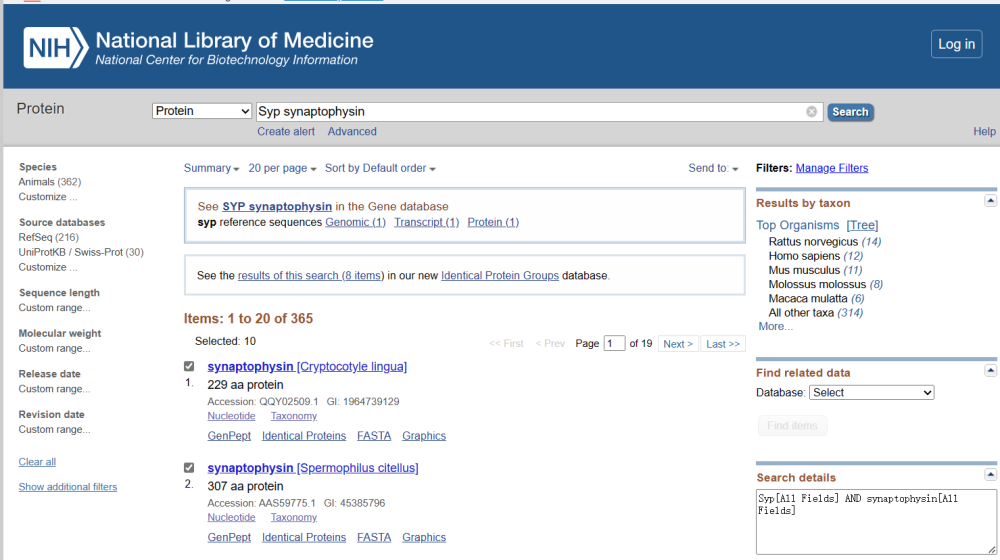
如图所示,在选择了Protein之后,我们在NCBI的搜索框中搜寻Syp synaptophysin的界面如下:
二、搜寻检索的处理
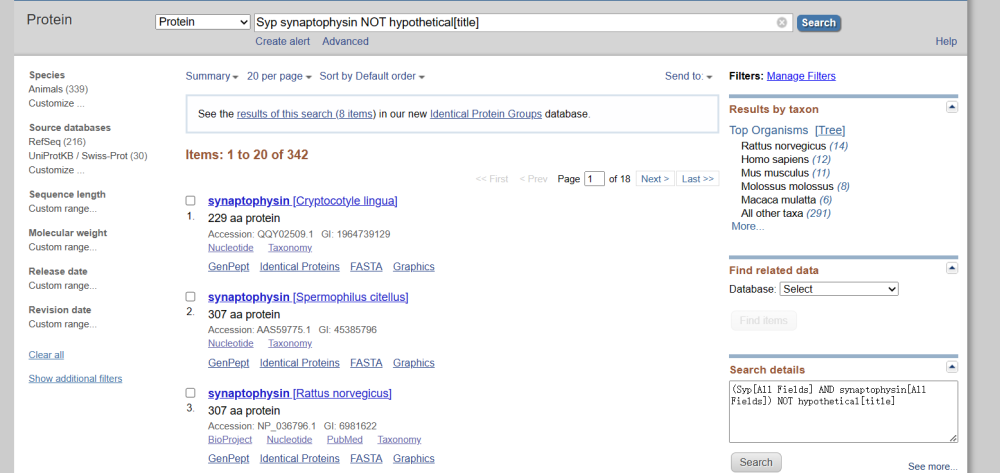
因为在搜寻的过程中,搜寻的结果太多,为了能够进一步的筛查数据,我们可以在搜寻栏中增添逻辑运算符,如NOT、OR、AND等。如图所示,我们为了进一步地去除标题中包含hypothetical的数据,将在检索框中,输入Syp synaptophysin NOT hypothetical[title] ,然后需要注意的是,我们在后面需要备注[title]
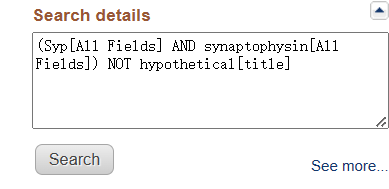
与此同时,我们也可以在Search details中复制搜索的结果并保存,在日后需要再次搜寻同样的结果的时候,可以直接复制上去
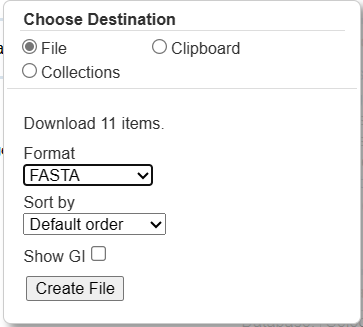
紧接着,我们点击Send to按钮,对已经选取不同物种的蛋白质序列进行下载,在这里我们选取十种左右不同的物种的SYP蛋白序列。选择FASTA的格式,点击Create File按钮,将下载到sequence.fasta的文件中
三、下载序列的处理
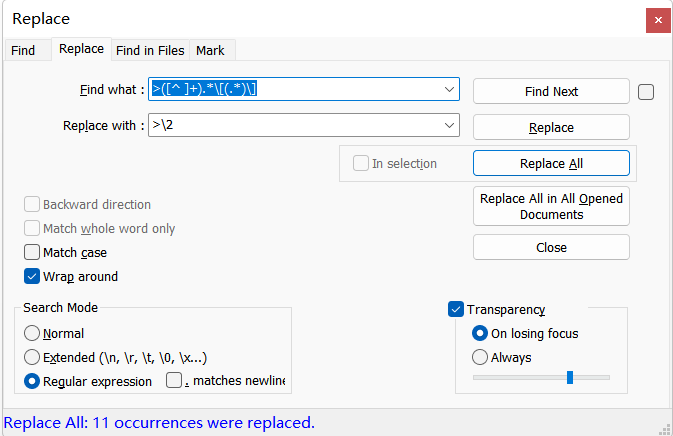
为了进一步地使得后面构建的进化树中,能够很好地观察到物种名,我们在这里需要对>后面的id和物种名进行处理,将id名替换成为物种名
所以在此我们将使用正则表达式进行替换,如图所示为替换的方式,其中的Find what中的字符为>([^ ]+).*\[(.*)\]
>([^ ]+).*\[(.*)\]的意思是:匹配以大于符号(>)开头,之后是一个或多个非空格字符的字符串,然后是一个方括号([])括起来的任意字符。
而当使用\2时,它表示对第二个捕获组的引用。这意味着,如果正则表达式中的模式匹配了一个字符串,并且第二个捕获组捕获了一些内容,那么\2将被替换为该捕获组中所捕获的内容。
例如,如果应用正则表达式>([^ ]+).*\[(.*)\]于字符串>Hello [World],那么\2将被替换为World,因为World是第二个捕获组所捕获的内容。
所得的结果如下:
好啦,对于已经下载好的蛋白质序列也处理了,我们接下来就可以进行基因进化树的构建了!在下一篇中,小果将给大家带来更详细的,去使用mega软件进行进化树的构建的过程!是不是非常期待呢?如果大家对生信知识感兴趣的话,欢迎大家点击http://www.biocloudservice.com/home.html来进行学习哦!