非模式生物的注释信息检索
自RNA测序(RNA-seq)的技术被发明以来,它在分子生物学的领域内得到了广泛的应用。与传统的方法相比,RNA-seq更能够全面和精准地揭示基因在不同层面上的功能和作用。这一技术突出了mRNA剪接的复杂性,并阐明了非编码RNA以及增强子RNA对于基因表达的调控机制,增强了我们对于生物过程的了解,推动了生物学在基因组、转录组以及表观遗传学等方面的研究进步。
然而,当我们通过RNA-seq得到了一系列差异基因的geneid后,一个问题摆在我们面前:我们并不清楚这些geneid实际上对应的是哪些基因。此时,我们需要对这些基因做进一步的功能注释,也就是找出这些基因在生物体中实际上起了什么作用,这些差异表达的基因是否与特定的表型相关?要解答这些问题,就需要我们把得到的geneid与gene数据库进行比对,挖掘出每一个geneid背后的基因信息。这个过程就好比查字典一样,我们有了页码,接下来就需要知道这个页码对应的是什么内容。
对于像人类、小鼠、拟南芥、大肠杆菌等模式生物来说,进行基因注释的过程相对容易,因为它们都有相对完善的数据库可以参考。但是,对于非模式生物,由于通常缺乏良好的参考基因库,进行基因注释的过程就显得十分困难。此时,R包AnnotationHub提供了一种非常有效的解决方案。它能够轻松地检索非模式生物的注释信息,对过去在注释非模式生物基因时遇到的问题提供了有效的解决方式。
下面小果用玉米作为示范
下载AnnotationHub包并加载
BiocManager::install(“AnnotationHub”) #包下载
library(AnnotationHub) #加载
有两种方式可以找到想要的非模式生物注释信息
第一种
ah <- AnnotationHub()
query(hub, “zea”)
AnnotationHub with 12 records
# snapshotDate(): 2022-10-31
# $dataprovider: ftp://ftp.ncbi.nlm.nih.gov/gene/DATA/, NCBI,DBCLS, WikiPathways, …
# $species: Zea mays, Gibberella zeae, Zea mays_var._japonica, Heliothis zea, Heli…
# $rdataclass: OrgDb, SQLiteFile, Tibble, Inparanoid8Db
# additional mcols(): taxonomyid, genome, description, coordinate_1_based,
# maintainer, rdatadateadded, preparerclass, tags, rdatapath, sourceurl,
# sourcetype
# retrieve records with, e.g., ‘object[[“AH10514”]]’
title
AH10514 | hom.Gibberella_zeae.inp8.sqlite
AH91642 | MeSHDb for Zea mays (Corn, v001)
AH91804 | wikipathways_Gibberella_zeae_metabolites.rda
AH91817 | wikipathways_Zea_mays_metabolites.rda
AH97909 | MeSHDb for Zea mays (Corn, v002)
… …
AH107469 | org.Zea_mays.eg.sqlite
AH107470 | org.Zea_mays_var._japonica.eg.sqlite
AH108747 | org.Helicoverpa_zea.eg.sqlite
AH108748 | org.Heliothis_zea.eg.sqlite
AH109103 | org.Gibberella_zeae_PH-1.eg.sqlite
通过检索,org.Zea_mays.eg.sqlite就是小果想找的玉米Org数据库。
第二种
我们也可以访问它的官方接口来更快地获取

再然后我们就可以点击species按照物种来进行快速检索
从左下角可以清楚地看到有将近6000种可用的物种,这是不是比你想象得多很多呢?
输入小果的好朋友玉米的学名zea mays
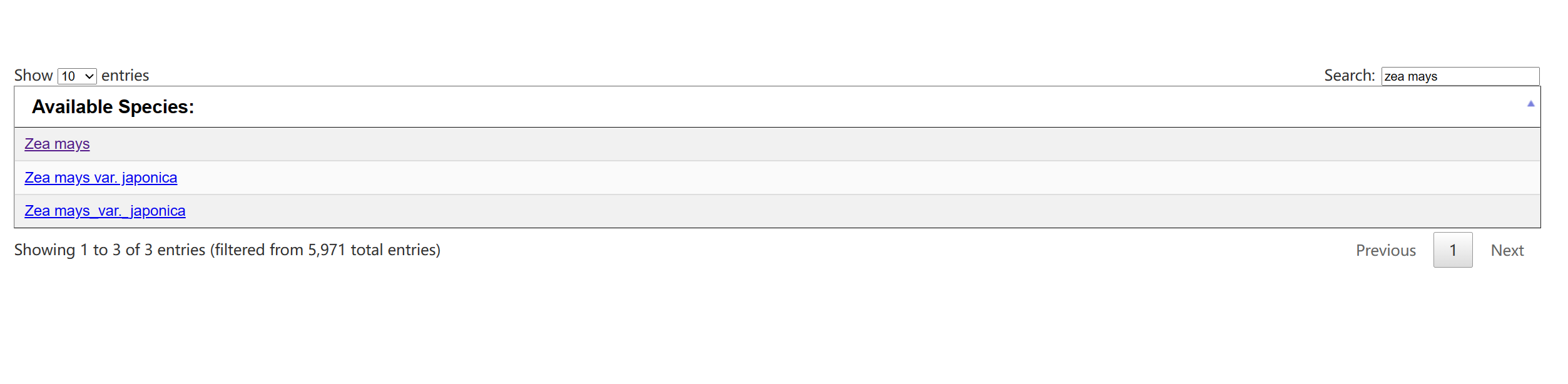
点击zea mays,太好了小果找到了玉米的org数据库~
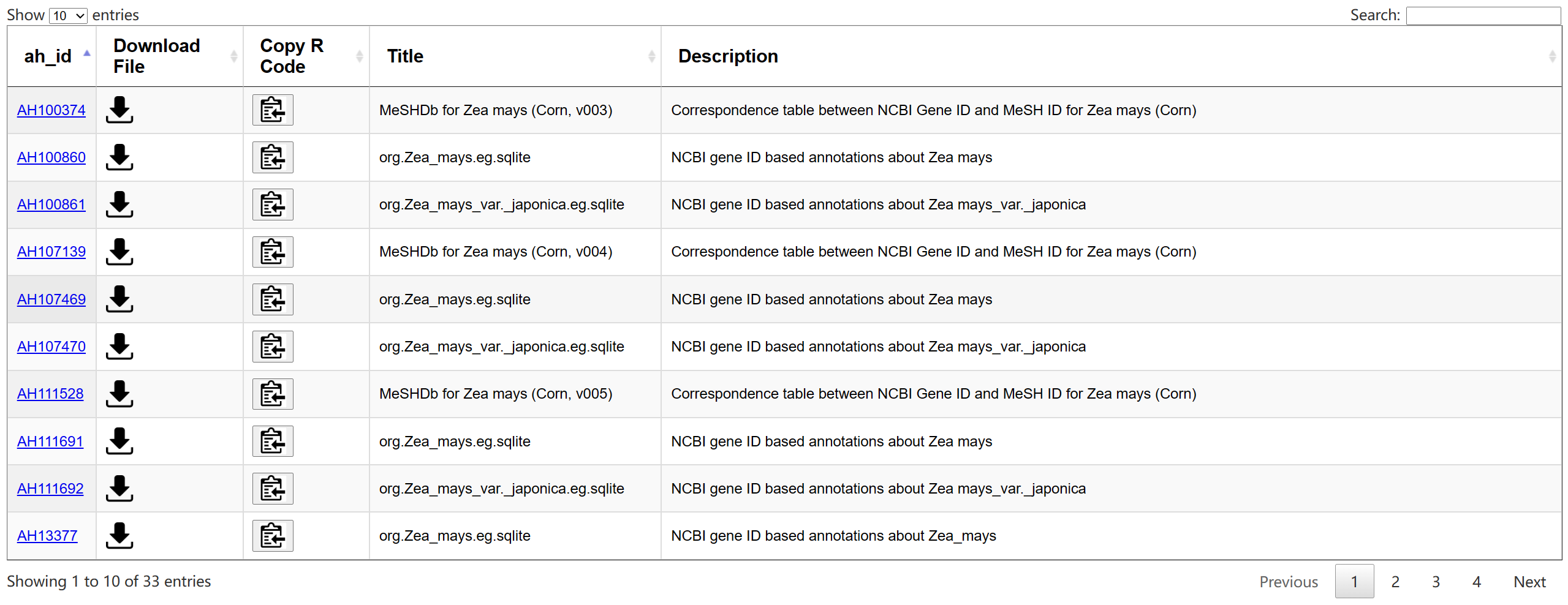 |
获取玉米注释信息
通过对应的accession number, 获取文件AH107469 。
并将文件存入maize对象中,我们可以看到它包含了玉米49905个基因的注释。
> maize <- hub[[‘AH107469’]]
downloading 1 resources
retrieving 1 resource
|==============================================| 100%
loading from cache
> length(keys(maize))
[1] 49905
这个Org数据库,包含有以下一些注释信息(好多小果都还未接触过):
> columns(maize)
[1] “ACCNUM” “ALIAS” “CHR”
[4] “ENTREZID” “EVIDENCE” “EVIDENCEALL”
[7] “GENENAME” “GID” “GO”
[10] “GOALL” “ONTOLOGY” “ONTOLOGYALL”
[13] “PMID” “REFSEQ” “SYMBOL”
[16] “UNIGENE”
有了Org数据库之后就可以进行后续的操作了。比如不同数据库的ID转化,基因/转录本/蛋白的ID转化,GO注释以及其他注释。
怎么样是不是很方便呢,欢迎和小果讨论~
小果的云生信平台还有更多方便实用的工具,快来瞧瞧吧