hello,今天小果来教大家如何对KEGG富集分析的结果通过ggplot2实现可视化并绘制对应的KEGG通路网络图 ,让你的分析结果更加美观!感兴趣的话就和小果一起来看一下吧!
准备需要的R包
先来简单介绍一下我们今天用到的R包,分别是DOSE、clusterProfiler、org.Hs.eg.db、ggplot2、tidyverse以及enrichplot包。同学们要提前下载好哦!在这里小果为大家简单的介绍一下其中几个R包:
- DOSE:DOSE包主要用于基因功能富集分析,它能够通过比较输入的基因列表与已知的基因功能注释信息,找出在特定功能类别中富集的基因。
- clusterProfiler:clusterProfiler是一个用于生物信息学分析的R包,主要用于功能注释和富集分析。它提供了针对多种生物数据类型的功能富集分析方法,包括GO、KEGG、Reactome等。该包还提供了可视化工具,使用户能够更好地理解和解释功能富集结果。
- org.Hs.eg.db:org.Hs.eg.db是一个R语言中用于人类基因注释的数据库包。它提供了对人类基因组的注释信息,包括基因ID、基因名称、基因符号、通路信息等。使用org.Hs.eg.db包,用户可以将基因ID转换为易读的基因符号,从而更容易理解和解释功能富集结果。
- tidyverse:tidyverse是一个由多个R包组成的集合,旨在提供一套一致且易于使用的数据处理和可视化工具。这些包包括ggplot2、dplyr、tidyr、readr等。其中,ggplot2是用于数据可视化的重要组成部分,提供了强大且灵活的绘图功能,使用户能够创建高质量的图表,用于数据探索和展示。
- enrichplot:enrichplot是一个用于生物信息学分析的R包,主要用于功能富集结果的可视化。它提供了多种绘图函数,可以绘制基因集的富集分析结果,如GO条形图、KEGG通路网络图等。enrichplot提供了丰富的参数设置,以满足用户对结果可视化的个性化需求。
KEGG富集分析
enrichKEGG进行富集分析
首先,加载DOSE包并使用该包中的基因集作为我们分析的对象:
library(“DOSE”)
data(geneList, package = “DOSE”)
g_list <- names(geneList)[1:100]
head(g_list)
![]()
利用enrichKEGG函数进行富集分析,对指定的基因列表进行KEGG通路富集分析,并设置p-value和q-value的阈值。
library(“clusterProfiler”)
library(“org.Hs.eg.db”)
ek <- enrichKEGG(gene = g_list,
organism = “hsa”,
pvalueCutoff = 0.05,
qvalueCutoff = 0.05)
write.table(ek, file = “ek.txt”, sep = “\t”, quote = FALSE, row.names = FALSE)
注意:
- pvalueCutoff = 0.05:设置p-value的阈值,用于筛选富集分析结果中统计显著的通路。在这里,设定为0.05,即p-value小于0.05的通路被认为是显著富集的通路。
- qvalueCutoff = 0.05:设置q-value的阈值,q-value是经过FDR校正的p-value,用于控制多重假设检验的错误发现率。同样,设定为0.05,即q-value小于0.05的通路被认为是显著富集的通路。
至此我们已经完成了富集分析的流程,让我们一起来看一下分析的结果吧!
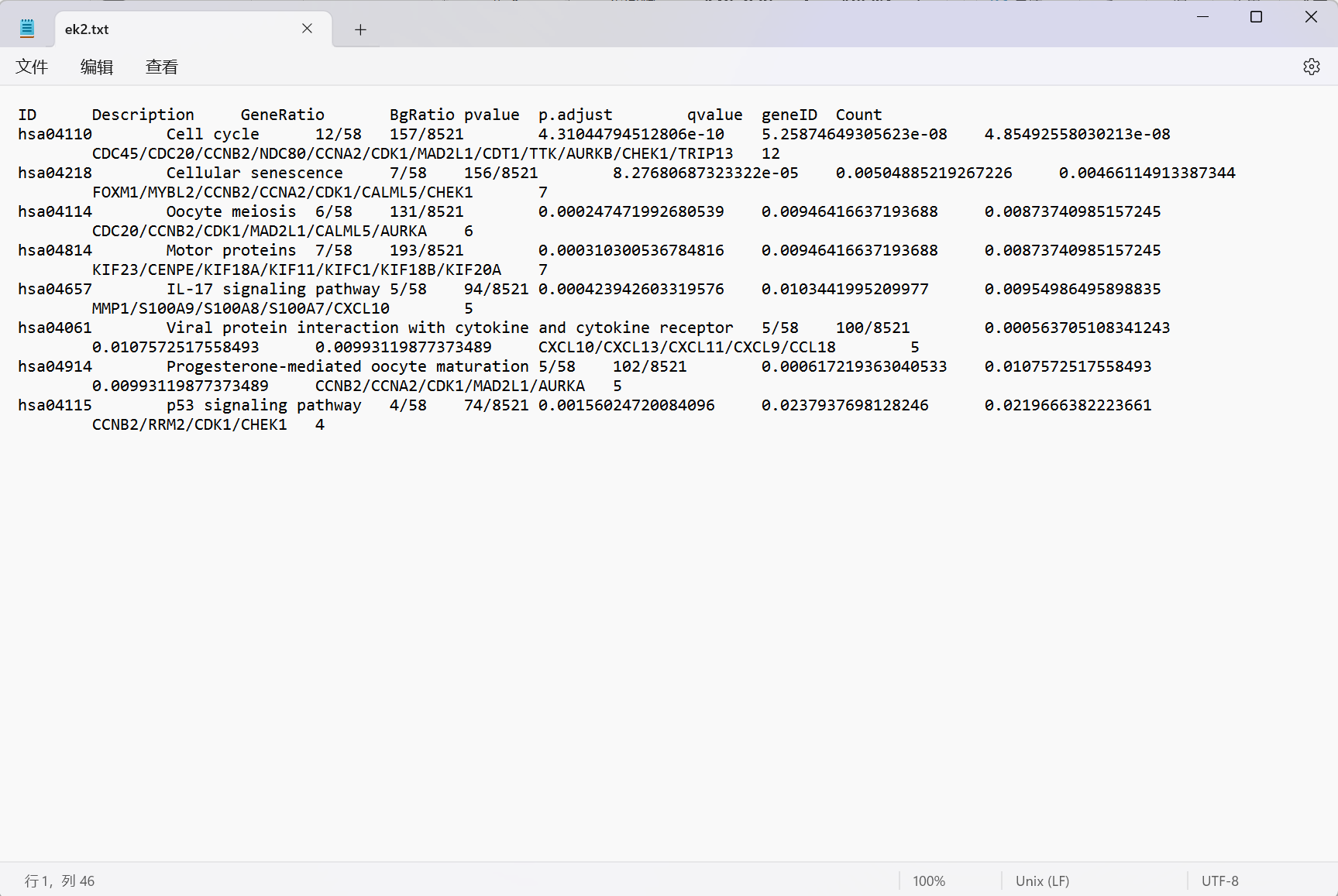
需要注意的是结果中geneID为EntrezID。
head(ek@result$geneID)

将EntrezID转换为Symbol
使用setReadable函数将EntrezID转换为Symbol:
ek2 <- setReadable(ek,
OrgDb = “org.Hs.eg.db”,
keyType = “ENTREZID”)

ggplot2可视化结果
首先,我们需要通过安装tidyverse包并读取保存的富集分析结果文本文件,对表格进行处理和计算Enrichment Factor,在这里小果用tidyverse包的separate函数将GeneRatio和BgRatio的分子分母先分开,再进行计算:
library(tidyverse)
ek.rt <- read.table(“ek.txt”, header = TRUE, sep = “\t”, quote = “”)
ek.rt <- separate(data = ek.rt, col = GeneRatio, into = c(“GR1”, “GR2”), sep = “/”)
ek.rt <- separate(data = ek.rt, col = BgRatio, into = c(“BR1”, “BR2”), sep = “/”)
ek.rt <- mutate(ek.rt, enrichment_factor = (as.numeric(GR1) / as.numeric(GR2)) / (as.numeric(BR1) / as.numeric(BR2)))
筛选出前10个通路进行可视化:
ek.rt10 <- ek.rt %>% filter(row_number() >= 1, row_number() <= 10)
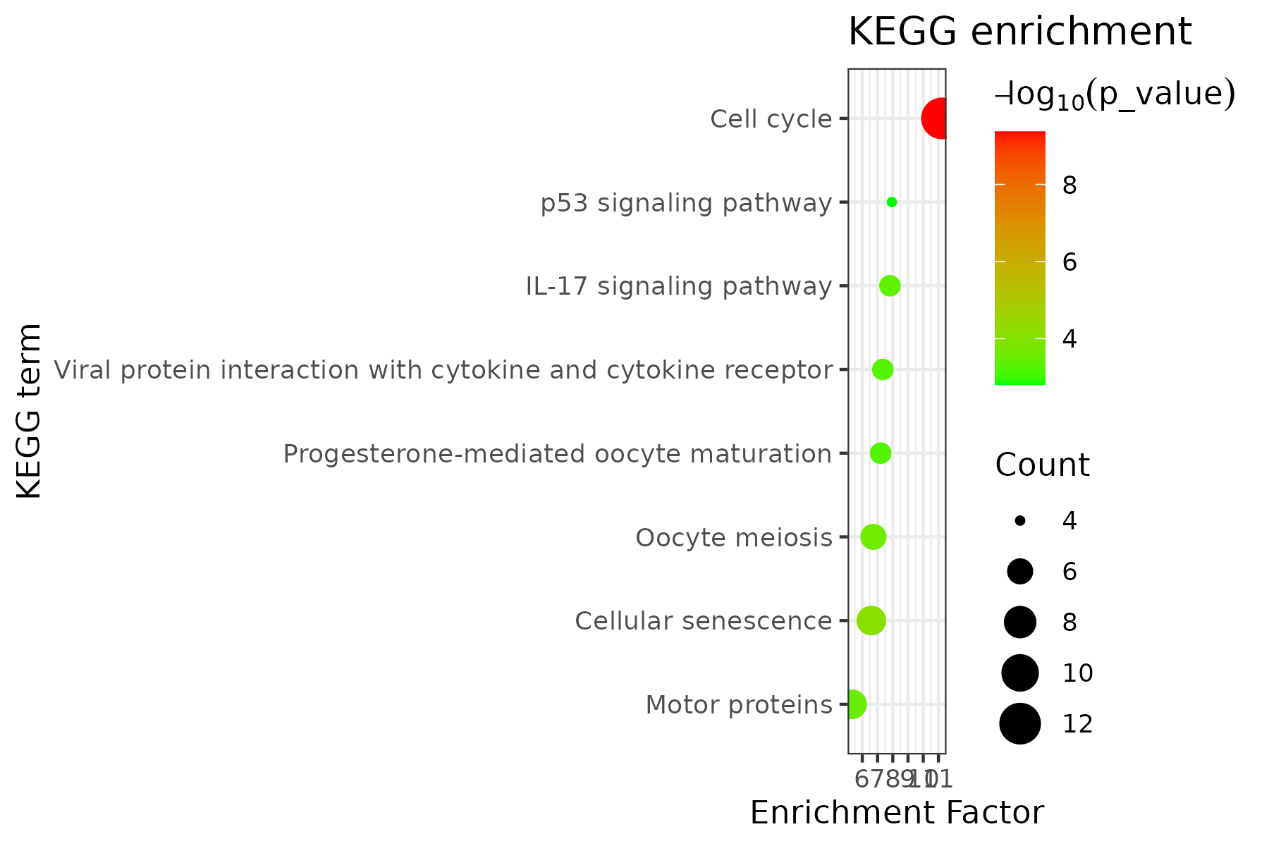
ggplot2绘制dotplot
使用ggplot2包绘制Enrichment Factor与KEGG term之间的关系图:
library(“ggplot2”)
p <- ggplot(ek.rt10, aes(enrichment_factor, fct_reorder(factor(Description), enrichment_factor))) +
geom_point(aes(size = Count, color = -1 * log10(pvalue))) +
scale_color_gradient(low = “green”, high = “red”) +
labs(color = expression(-log[10](p_value)), size = “Count”,
x = “Enrichment Factor”, y = “KEGG term”, title = “KEGG enrichment”) +
theme_bw()
ggsave(“er.rt10_KEGG.png”, width = 6, height = 4)
我们一起来看看效果图吧!
绘制KEGG通路网络图
使用enrichplot包的cnetplot函数绘制KEGG通路网络图,并将结果保存为PDF文件。
library(“enrichplot”)
pdf(file = “net-pathway.pdf”, width = 8, height = 10)
cnetplot(ek2)
dev.off()
效果图如下:

以上教程小果介绍了如何使用R语言中的DOSE和clusterProfiler包进行基因集的KEGG通路富集分析,并通过ggplot2和enrichplot实现可视化化。通过这些代码,你可以将自己的基因列表进行富集分析,并进一步将结果进行可视化以帮助解释和理解数据哦!怎么样,你学会了嘛?