许多生信分析报告里都会有上一张PCA图,那它是什么,又为什么会如此重要呢?小果今天就为大家讲解。
主成分分析(Principal Component Analysis,PCA)是做生信分析的一种非常常用的数据分析算法,它在做高维数据分析时极为有用广泛应用于多领域,如医学研究、市场分析和财务分析等。
它的原理是通过对协方差矩阵进行特征分解,以得出数据的主成分与它们的权值,即特征值和特征向量。它产生的结果可以理解为对原数据中的方差做出解释:哪一个方向上的数据值对方差的影响最大?
小果这里举个栗子 ,比如我们得到了一组转录组表达数据,其中有n个转录本的表达量信息。那么我们就有n个变量了,这个n有时会非常大,会到上百万千万。若是直接去对比两个或多个数据,就显得非常困难。经过主成分分析后,就可以将这n个变量降维到r个量(r<n)。
这r个主成分会依据方差的大小进行排序,称作主成分(PC)1、主成分2、……主成分r。而每个主成分的方差在这一组变量中的总方差中所占的比例,即是主成分的贡献度。通常来说,我们仅考察贡献度前2或者前3的主成分,经过可视化后,即得到了二维或三维PCA散点图。
PCA提供了一种降低数据维度的有效办法;如果分析者在原数据中除掉最小的特征值所对应的成分,那么所得的低维度数据必定是最优化的(也即,这样降低维度必定是失去信息最少的方法)。
下面小果通过一个案例来让大家更深入地学习。
加载R包和数据
#加载R包和数据
library(tidyverse)
library(“factoextra”)
data(“decathlon2”)
df <- decathlon2[1:23, 1:10] #删除分类变量
观察一下数据,数据类型为数据框”data.frame”,23行,10列。

数据的变量分别是100米、400米、跳远、铅球等体育项目

行是不同人的名字

接下来我们开始正式对这个数据集进行主成分分析
主成分分析
1.分析
本次案例使用的是R语言自带的主成分分析函数,prcomp和 princomp,它们可以很好的与其他的包兼容。后续会结合factoextra包进行数据的可视化。
#主成分分析
res.pca <- prcomp(df,scale. = T)
通过这样一行代码就可以对数据完成主成分分析
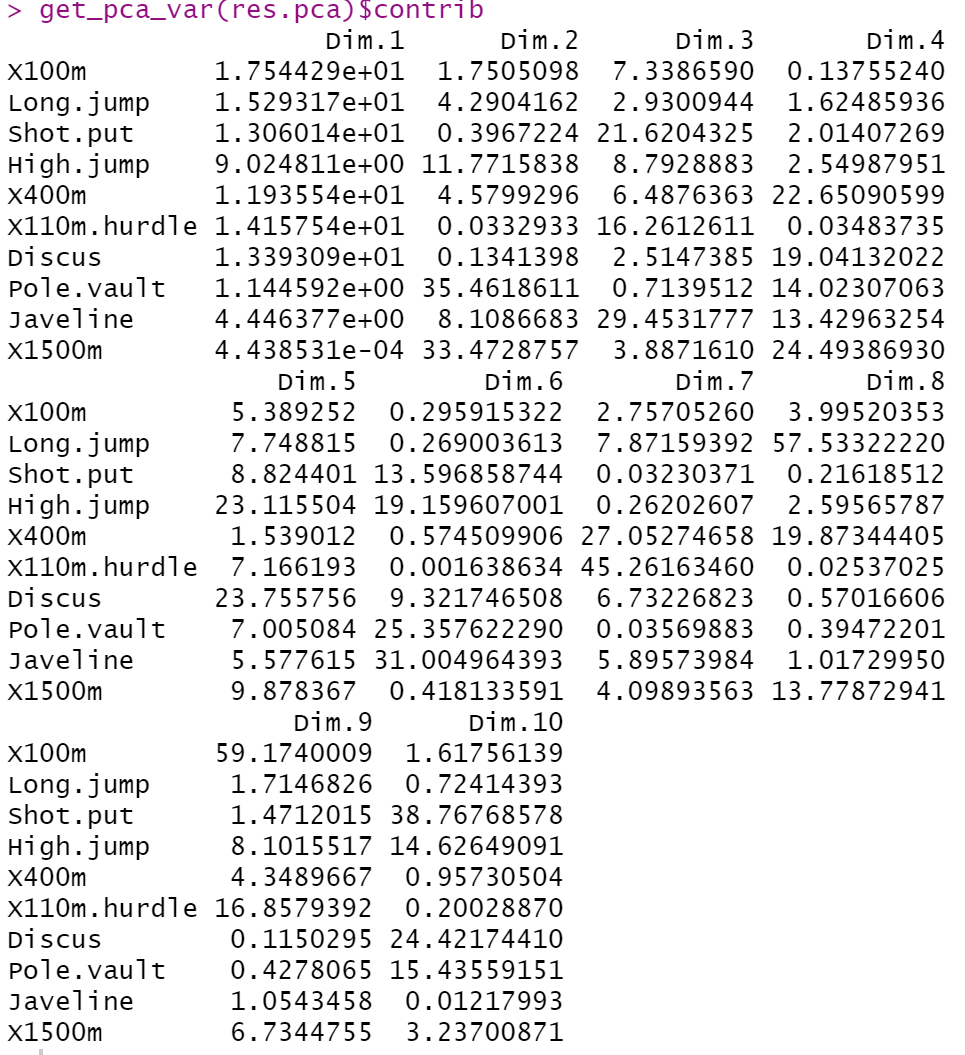
用get_eig()函数获得分析的结果。

返回的三个列,分别是特征值(eigenvalue)、方差贡献率(variance.percent)和累积方差贡献率(cumulative.variance.percent)。总共有十个变量也就是10个维度。特征值依次从大到小排列,对应的方差贡献率也是从大到小排列的趋势。这意味着排名在前面的就是数据的主成分。
数据的方差等于协方差所有特征值之和,第i个主成分的方差等于协方差矩阵的第i个特征值。然后从前到后求和就得到累积方差贡献率。所以累积方差贡献率的大小表示了当前选择的所有主成分携带原数据的信息的比例。
一般情况下会选择90%为限,但是小果提示这里还是要具体情况具体分析哦。
2.结果可视化
2.1 碎石图
fviz_screeplot(res.pca, addlabels = TRUE, ylim = c(0, 45))
反映各成分方差解释比例,非常直观。
2.2特征值
fviz_screeplot(res.pca,
choice=”eigenvalue”,
addlabels = TRUE,
ylim = c(0, 4.5))
十个变量的特征值排序
2.3查看变量的分布
# 变量分布可视化
fviz_pca_var(res.pca,
col.var=”contrib”, # 根据贡献度着色
gradient.cols = c(“#00AFBB”, “#E7B800”, “#FC4E07”),
repel = TRUE
)
这个图小果教你怎么看。
变量贡献图显示了每个变量在主成分上的贡献。线条越长越接近红色,对应的主成分贡献较大。横纵坐标分别代表主成分1 ,主成分2,百分比是主成分1 2的方差解释。
对Dim1来说要横着看,对其影响最大的是100m和Long.jump两个变量。
对Dim2来说要竖着看,对其影响最大的是1500m和Pole.vault两个变量。
2.4特征对主成分1的贡献
#特征贡献
fviz_contrib(res.pca, choice = “var”, axes = 1, top = 10)
2.5样本PCA
样本PCA分布,通常也是论文里出现频次最高的PCA图。小果这里选择了有三组数据的鸢尾花数据集进行展示。
#样本pca图
iris.pca <- prcomp(iris[,-5])
fviz_pca_ind(iris.pca,
label = “none”, # hide individual labels
habillage = iris$Species, # color by groups
palette = c(“#00AFBB”, “#E7B800”, “#FC4E07”),
addEllipses = TRUE # Concentration ellipses
)
通过贡献最高的两个主成分,在一个二维的平面上,就能在图像上显示这些数据的不同组。
好您是否明白了呢,如果有不懂的来和小果讨论吧~
PCA分析相关其他分析内容欢迎尝试本公司新开发的云平台生物信息分析小工具,零代码完成分析,云平台网址:
http://www.biocloudservice.com/home.html