Lasso(Least Absolute Shrinkage and Selection Operator)算法是一种用于特征选择和模型正则化的线性回归方法。它通过在损失函数中引入L1正则化项,迫使模型系数稀疏化,将一些系数压缩为零,从而实现特征选择的效果。
想要深入了解Lasso算法原理,需要我们一步一步寻找Lasso算法的来源,如下是小果的学习思考总结:
- 线性回归模型:
我们先回顾一下简单的线性回归模型:y = β0 + β1×1 + β2×2 + … + βnxn,其中y是因变量,x1, x2, …, xn是自变量,β0, β1, β2, …, βn是模型的系数。
- 最小二乘法:
在普通的线性回归中,我们使用最小二乘法来估计模型的系数,即通过最小化实际观测值和模型预测值之间的残差平方和来求解系数。这种方法没有对系数进行任何约束。
- 引入L1正则化项:
在Lasso算法中,我们引入L1正则化项作为约束条件,以促使模型系数稀疏化。L1正则化项是模型系数的绝对值之和与一个调节参数λ的乘积,可以写为:λ * (|β1| + |β2| + … + |βn|)。
- Lasso的优化目标:
Lasso的目标是将最小化的损失函数与L1正则化项相结合,形成优化目标函数:minimize(平方损失函数 + λ * (|β1| + |β2| + … + |βn|))。
- 最小化目标函数:
我们使用优化算法来最小化目标函数,一种常用的方法是坐标下降法(coordinate descent)。在坐标下降法中,我们逐个更新模型系数,通过迭代优化来逐步逼近最优解。
- 坐标下降法的迭代过程:
①初始化模型系数β为零或一个较小的值。
②针对每个系数βi,固定其他系数不变,通过求解最小化目标函数的一维子问题来更新βi。
③重复上述步骤,直到满足收敛条件。
- 子问题的求解:
在每个子问题中,通过求解最小化目标函数关于βi的一阶导数为零的方程来更新βi。这个方程可以写为:2 * (Σ(xi * (yi – (β0 + β1×1 + β2×2 + … + βnxn)))) + λ * sign(βi) = 0,其中sign(βi)是βi的符号函数。
- Lasso算法的特点:
Lasso算法的特点在于它引入了L1正则化项,促使模型系数稀疏化,实现特征选择的效果。Lasso可以通过调节正则化参数λ来控制稀疏性程度,较大的λ会导致更多的系数被压缩为零。
总结一下,Lasso算法通过在损失函数中引入L1正则化项,迫使模型系数稀疏化,实现特征选择和模型正则化的效果。它使用坐标下降法迭代优化,通过解决子问题来更新模型系数。Lasso算法在机器学习中被广泛应用,特别适用于高维数据和特征选择的场景,尤其是在选择特征基因用于疾病诊断时。
R包glmnet,提供了高效的Lasso和Elastic-Net正则化方法,用于拟合广义线性模型。它支持线性回归、逻辑回归、多项式回归、泊松回归、Cox模型等多种模型。glmnet的算法基于循环坐标下降,可以快速计算整个正则化路径。此外,glmnet还支持自定义的GLM family对象和relax选项。通过glmnet,可以进行特征选择、模型正则化和预测等任务。
以下是一个简单的示例,展示如何使用R包glmnet进行糖尿病预测模型的训练和评估:
代码具体包括:
Step1 导入数据集以及数据预处理
# 这里的”inputdata.txt”是自行准备的本地文件,小果给大家附在最后。
# 1.导入数据集以及数据预处理
# 安装和加载所需的库
# install.packages(“glmnet”)
library(glmnet)
# 导入数据集
data <- read.table(file = “D:/wanglab/life/ziyuan/20230628/inputdata.txt”,header = T) # 替换为实际数据集文件的路径
data[, 1:8] <- scale(data[, 1:8])# 数据标准化
data$diabetes <- as.factor(data$diabetes)# 将目标变量转换为因子
# 划分数据集为训练集和测试集
set.seed(123) # 设置随机种子,保证结果可复现
indices <- sample(1:nrow(data), nrow(data)*0.8) # 80%的数据用于训练,20%的数据用于测试
train_data <- data[indices, ]
test_data <- data[-indices, ]
Xtrain<-as.matrix(trainingset[,1:8])
Ytrain<-as.matrix(trainingset[,9])
Xtest<-as.matrix(testingset[,1:8])
Ytest<-as.matrix(testingset[,9])
Step2创建lasso回归模型
# 2.创建lasso回归模型
lsofit<-glmnet(Xtrain,Ytrain,family=”binomial”,alpha=0.1)
print(lsofit)##lasso回归系数
coef.apprx<-coef(lsofit, s=0.017)
coef.apprx
plot(lsofit,xvar=”lambda”,label=TRUE)# 画Lasso筛选变量动态过程图

Step3 K折交叉验证
# 3.K折交叉验证
set.seed(2022) #设置随机种子,保证K折验证的可重复性
lsocv<-cv.glmnet(Xtrain,Ytrain,family=”binomial”,nfolds=10)
lsocv ##print(lsocv)
lsocv$lambda.min
lsocv$lambda.1se
plot(lsocv) #绘制交叉验证曲线
交叉验证曲线图

coef(lsocv,s=”lambda.min”) #获取使模型偏差最小时λ值的模型系数
coef(lsocv,s=”lambda.1se”) #获取使模型偏差最小时λ值+一个标准误时的模型系数
cbind2(coef(lsocv,s=”lambda.min”),coef(lsocv,s=”lambda.1se”)) #合并显示
select_x <- names(which(coef(lsocv,s=”lambda.1se”)[-1,] != 0))
select_x得到对预测结果有影响的因素
Step 4.绘制AUC曲线评估模型
# 4.绘制AUC曲线评估模型
predictions <- lsocv %>% predict(as.matrix(test_data[, 1:8]))# 预测概率
# 计算AUC
library(pROC)
roc_obj <- roc(test_data$diabetes, predictions)
auc_value <- auc(roc_obj)
# 绘制AUC曲线
plot(roc_obj, main = “ROC Curve”)
text(0.5, 0.3, paste0(“AUC = “, round(auc_value, 3)), col = “red”, cex = 1.2)
AUC曲线图

Step5.绘制列线(Nomogram)图
# 5.绘制列线(Nomogram)图
library(rms);library(Hmisc);library(lattice);library(survival);library(Formula);library(ggplot2);library(SparseM)
NM<-datadist(trainingset)
options(datadist=’NM’)
lgrfit<-lrm(diabetes~pregnant+glucose+mass+pedigree+age,data=trainingset,x=T,y=T)
Nomg<-nomogram(lgrfit,fun=plogis,fun.at=c(.001,.01,.05,0.1,seq(.2,.8,by=.2),0.9,.95,.99,.999),lp=F,funlabel=”malignant Rate”)
plot(Nomg)
列线图(Nomogram)图

# 6.绘制多因素Cox回归森林图
# 6.绘制多因素Cox回归森林图
library(survival)
library(survminer)
library(tidyverse)
library(eoffice)
library(forestplot)
trainingset$diabetes <- as.numeric(trainingset$diabetes)
model <- coxph(Surv(diabetes)~pregnant+glucose+mass+pedigree+age,
data = trainingset[,c(‘pregnant’,’glucose’,’mass’,’pedigree’,’age’,”diabetes”)] )
ggforest(model = model,#cox回归方程
data = trainingset[,c(‘pregnant’,’glucose’,’mass’,’pedigree’,’age’,”diabetes”)],#数据集
main = “简单森林图”)#标题
绘制多因素Cox回归森林图 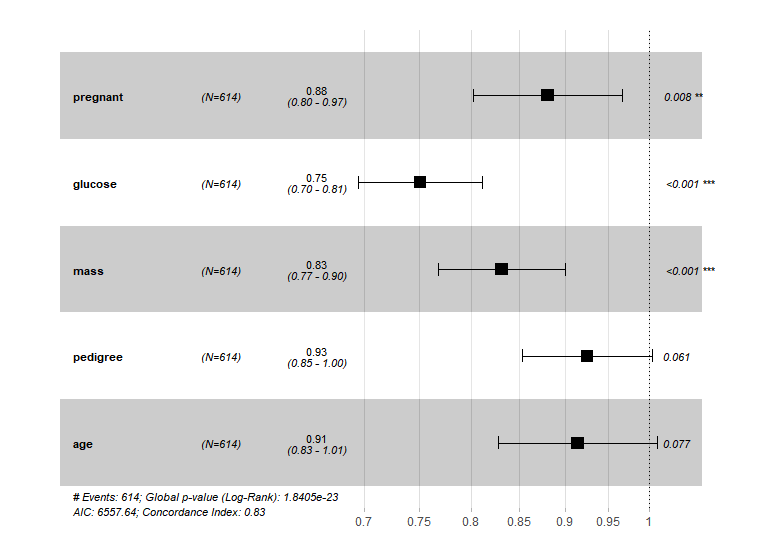
注:
# 请确保将路径”inputdata.txt”替换为实际数据集文件的路径,并根据需要调整模型参数和其他配置。
# 在绘制AUC曲线之前,确保您的模型输出的是预测的概率值,而不是类别标签。
厉害吧,我们这么快就完成了一个用lasso算法建立模型并评估。如果特征比较多,尤其是以基因为特征时lasso算法能从成千上万的基因中挑选出对预测表现影响较大的那一部分,甚至是几个基因。需要注意的是,如果特征值较多,可能需要大量的内存,耗时比较长。因此,小果建议使用我们的云生信平台,我们的平台提供强大的计算资源和深度学习工具。您可以访问我们的云生信平台,链接为:http://www.biocloudservice.com/home.html。