R语言作为数据分析和统计建模的流行工具,提供了许多功能强大的扩展包。其中,mnormt包是一个广泛应用于高级统计分析的重要工具。该包为用户提供了处理多元正态分布和多元t分布的功能,使得在多元数据分析和统计建模中更加方便和高效。
mnormt包的功能涵盖了多个方面,包括多元正态分布的随机抽样、密度估计、分布函数计算以及分位数计算等。此外,该包还提供了多元t分布相关的功能,如随机抽样、密度估计、分布函数计算和分位数计算等。这些功能的结合使得用户可以更加灵活地进行多元数据的分析,并能够利用多元正态分布和多元t分布进行假设检验、参数估计和置信区间构建等统计推断。
在使用mnormt包进行分析时,用户可以通过简单的函数调用完成复杂的统计计算。例如,使用函数dmnorm()可以计算多元正态分布的密度函数值,而函数rmnorm()则可以生成多元正态分布的随机样本。类似地,对于多元t分布,函数dt()可以计算密度函数值,函数rt()可以生成随机样本。这些函数的参数设置灵活,并且提供了丰富的选项,以满足不同分析需求。
此外,mnormt包还提供了一些用于多元数据可视化和模型拟合的功能。例如,函数persp()可以绘制多元正态分布的密度函数三维图像,函数pairs.mvt()可以绘制多元t分布的散点图矩阵。这些功能有助于用户更好地理解多元数据的特征和结构,并辅助模型建立和评估过程。
在实际应用中,mnormt包广泛应用于各个领域的数据分析和统计建模中。例如,在金融领域,研究人员可以利用mnormt包中的函数进行多元投资组合的风险评估和优化;在生物医学领域,研究人员可以使用该包进行多元生物指标的关联分析和模式识别。此外,mnormt包还在社会科学、环境科学和工程领域等多个领域得到了广泛应用。
要使用mnormt包,可以在R中使用以下命令进行安装和加载:
> install.packages(“mnormt”) #安装mnormt语言包
> library(mnormt) #加载语言包
示例:
基因表达数据的差异分析。
假设我们有两个实验组,每个组中包含多个样本,我们对这些样本进行了基因表达水平的测量。现在我们想要确定哪些基因在这两个组之间存在显著差异。
首先,我们需要加载mnormt包和一些其他必要的包,然后读取和准备我们的数据。假设我们的数据是一个矩阵,其中每一行代表一个基因,每一列代表一个样本。
# 加载所需的包
> library(mnormt)
> library(edgeR) # 用于差异表达分析的另一个常用包
# 读取和准备数据
> data <- read.csv(“gene_expression_data.csv”, header = TRUE)
> gene_expression <- data[, -1] # 去掉第一列,即基因名称列
接下来,我们可以使用mnormt包中的函数来比较两个组之间的基因表达差异。常见的方法之一是使用Hotelling’s T-squared检验,它可以在多元正态分布假设下检测组之间的差异。
# 执行Hotelling’s T-squared检验
> group1 <- gene_expression[1:10, ] # 第一个实验组的样本
> group2 <- gene_expression[11:20, ] # 第二个实验组的样本
# 计算每个基因的Hotelling’s T-squared统计量和p-value
> result <- apply(gene_expression, 1, function(x) {
t_stat <- hotelling.test(x ~ group, data = data.frame(x = x, group = c(rep(“Group1”, 10), rep(“Group2”, 10))))$statistic
p_value <- 1 – pt(t_stat, df = ncol(group1) – 1, lower.tail = FALSE)
c(t_stat, p_value)
})
# 将结果保存到一个数据框中
> result_df <- data.frame(Gene = rownames(gene_expression), T_statistic = result[, 1], P_value = result[, 2])
# 打印前几行结果
> print(head(result_df))
上述代码中,我们选择了前10个基因作为第一个实验组的样本,接下来的10个基因作为第二个实验组的样本。然后,我们使用hotelling.test()函数执行Hotelling’s T-squared检验,并计算每个基因的统计量和p-value。最后,我们将结果保存在一个数据框中并打印出前几行。

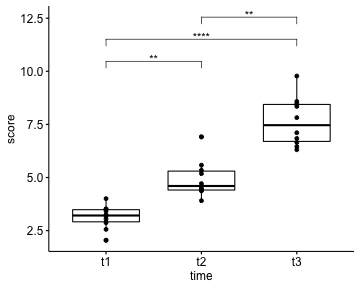

除了Hotelling’s T-squared检验,mnormt包还提供了其他用于多元正态分布的假设检验方法,如Wilks’ lambda检验和Pillai’s trace检验,可以根据具体需求选择适当的方法。

通过使用mnormt包,我们可以方便地进行基因表达数据的差异分析,并找出在不同实验组之间具有显著差异的基因。这有助于我们理解基因在不同生物条件下的调控和功能,并为生物信息学研究提供重要的洞察。
以上就是对R语言包mnormt的简单介绍啦,R语言包mnormt是一款强大的工具,为用户提供了处理多元正态分布和多元t分布的丰富功能。其简洁的函数接口和灵活的参数设置使得用户能够高效地进行多元数据的分析和统计推断。无论是在学术研究还是实际应用中,mnormt包都发挥着重要的作用,为高级统计分析提供了有力的支持。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html
References:
- https://www.educba.com/t-test-in-r/
- https://www.datanovia.com/en/blog/how-to-perform-paired-pairwise-t-tests-in-r/
- https://www.datanovia.com/en/blog/how-to-perform-multiple-t-test-in-r-for-different-variables/
- https://www.researchgate.net/figure/Size-power-curves-for-the-Wilks-Lambda-statistic-L-dashed-line-and-the-robust-Wilks_fig2_46494016
- https://stats.stackexchange.com/questions/557428/ridge-trace-plot-interpretation