上一次小果给大家分享了如何处理FASTQ文件,相信聪明的小伙伴们已经轻松掌握,对下一阶段的调整跃跃欲试啦。正如上一次所说,数据质量评估和预处理步骤在使用FASTQ文件进行进一步分析之前是很重要的,那么这一次小果就来给大家分享如何在一分钟内轻松完成FASTQ文件质量评估,并对质量评估结果附上详细的说明,干货满满,小伙伴们快来看看吧。
为什么要进行质量评估和预处理?
- 错误识别和修正:FASTQ文件包含了测序实验生成的原始碱基序列及其对应的质量值。这些质量值反映了每个碱基测序的可信度。数据质量评估可以帮助识别和修正可能存在的测序错误,例如测序碱基替代、插入或删除错误。通过修复这些错误,可以提高后续分析的准确性。
- 冗余和低质量片段过滤:FASTQ文件通常包含大量的测序片段,其中可能存在冗余序列、PCR重复或者低质量的片段。对数据进行质量评估和预处理可以过滤掉这些不必要的片段,减少数据量和噪音,提高后续分析的效率和准确性。
- 质量控制:数据质量评估可以提供关于测序数据质量的详细统计信息,例如每个碱基的质量分布、测序深度等。这些信息对于选择合适的阈值、确定分析方法和解释结果非常重要。通过对数据质量进行控制,可以确保后续分析的可靠性和可重复性。
- 数据清洗:在进行某些分析之前,需要对数据进行清洗,以去除低质量的碱基或者不可靠的片段。这样可以排除噪音引入和偏差,确保分析结果更加准确和可靠。
FASTQ数据质控过滤软件—SOAPnuke
当今市面上有许多可以对FASTQ数据进行质控过滤的软件,SOAPnuke是其中对新手比较友好,使用起来比较简单的入门级软件。小果这次就在linux中以SOAPnuke为例,教给大家质量评估的步骤。
下面是SOAPnuke的一些常用参数的介绍:
- -1:指定待处理数据的第一对(或唯一)测序文件的路径。
- -2:指定待处理数据的第二对测序文件的路径(如果是双端测序数据)。
- -r:指定保存处理结果的路径。
- -C:用于指定配置文件的路径。配置文件包含了SOAPnuke的详细设置,如处理过程中所使用的质量计算方法、质量阈值等。可以根据需要进行自定义设置。
- -5:指定5’端剪切的碱基数。该参数用于去除测序文库中的低质量末端,提高数据质量。
- -3:指定3’端剪切的碱基数。该参数同样用于去除测序文库中的低质量末端。
- -l:指定最短有效读长(clean read length)。该参数用于去除较短的reads,提高数据质量。
- -q:指定质量阈值。该参数用于将低于指定质量阈值的碱基标记为N,从而去除低质量的碱基。
- -n:指定允许的最大连续低质量碱基数。该参数用于去除具有连续低质量碱基的reads。
- -M:指定保留的最小平均质量值。该参数用于过滤掉平均质量低于指定阈值的reads。
- -d:指定adapter序列文件的路径。该参数用于去除reads中的adapter序列。
- -t:指定线程数。该参数用于指定并行处理的线程数,加快处理速度。
- -D:开启adapter序列的存在性检测和去除。
- -I:开启PCR冗余序列的检测和去除。
- -O:开启PCR冗余序列配对比对的检测和去除。
这些只是SOAPnuke的一些常用参数,还有其他一些可选参数用于更详细地控制预处理过程。使用适当的参数设置可以根据实际需求进行数据质量控制和预处理,提高后续分析的准确性和可靠性。小伙伴们如果想了解的话可以在命令行中运行SOAPnuke filter –help,查看SOAPnuke更多完整参数。
操作步骤
# 创建文件夹
mkdir 00_ref 01_clean 02_align 03_SNPCalling 04_annotation raw_data
之所以创建这么多文件夹是因为质量评估只是我们分析流程的第一步,在后续分析流程还会产生以下文件:
过滤→clean.fq→比对→clean.bwa.sam→数据格式转换→clean.bwa.bam→索引结果排序→clean.bwa.sorted.bam→变异检测→clean.bwa.snp.vcf
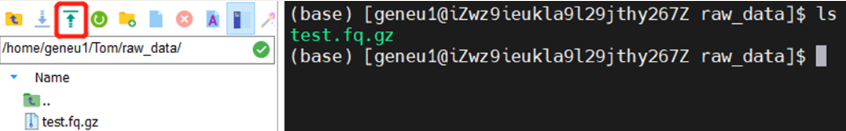
这里小果建议大家使用mobaXterm来连接linux服务器,因为mobaXterm可以直接在命令行窗口左边工具栏实现数据的上传和下载等功能,非常简单方便,如下图这个框中的箭头就是数据上传按钮,而它左边另一个箭头就是数据下载按钮。创建成功后通过左侧工具栏将下机数据拷贝至新建目录raw_data:
# 使用SOAPnuke过滤数据(注意,此命令为连续一行)
SOAPnuke filter -1 ./raw_data/G1211112269_Barcode_e.fq.gz -l 10 -q 0.5 -n 0.01 -T 1 -o ./01_clean/ -C test_clean.fq.gz
# 可在01_clean路径下使用less命令逐一查看测序数据质量评估表,简单展示过滤前后数据总体情况如下
less 01_clean/Basic_Statistics_of_Sequencing_Quality.txt
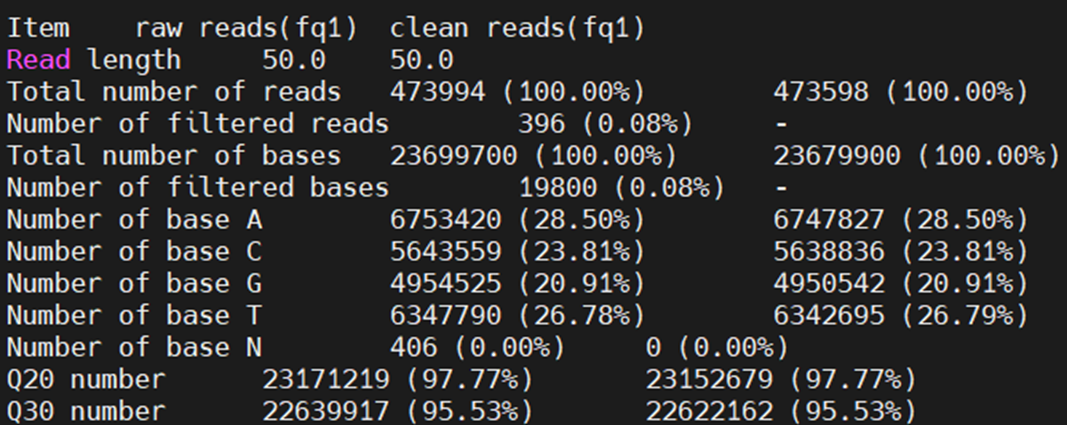
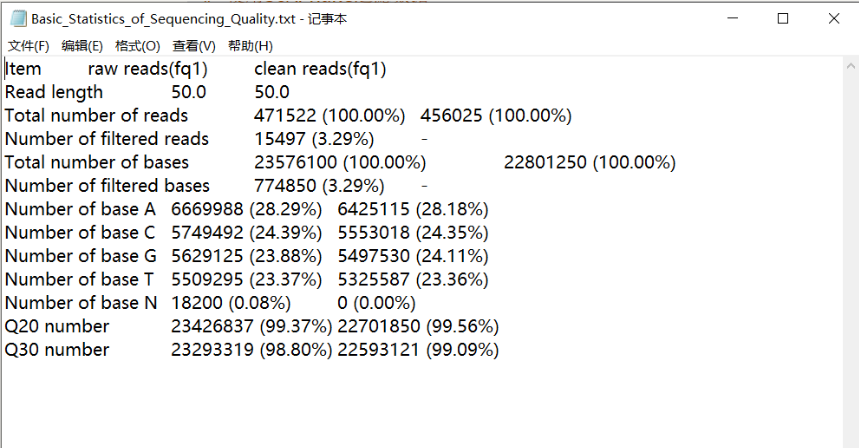
大家也可以把数据下载到本地进行查看,可在01_clean路径下找到”Basic_Statistics_of_Sequencing_Quality.txt”文件。该文件提供了过滤前后数据质量的对比统计信息,可以查看以下指标:数据总数、总碱基数、GC含量、Q20和Q30的比例等,可通过查看该文件了解数据质量与 过滤情况,截图如下
到这里为止质量评估的操作就结束啦,是不是非常简单呢?下一次小果会详细给大家介绍如何观看质量评估结果以及生成的质量评估报告,感兴趣的小伙伴们一定要继续关注哦~
如果小伙伴们平时在生信分析的操作过程中遇到困难,欢迎大家使用小果开发的生信工具平台http://www.biocloudservice.com/home.html,大家在新接触一个知识的时候,与其先花费大量时间死啃知识点,不如先利用好工具先自己上手接触流程,在跑完一遍全流程后再返回去理解知识点,相信可以更好更快地理解,达到事半功倍的效果!