书接上回,上一次小果给大家分享了如何进行FASTQ文件的质量评估,这次就给大家分享一下如何观看质量评估结果,小伙伴们赶紧往下看吧。
# 可在01_clean路径下使用less命令逐一查看测序数据质量评估表,简单展示过滤前后数据总体情况如下
less 01_clean/Basic_Statistics_of_Sequencing_Quality.txt
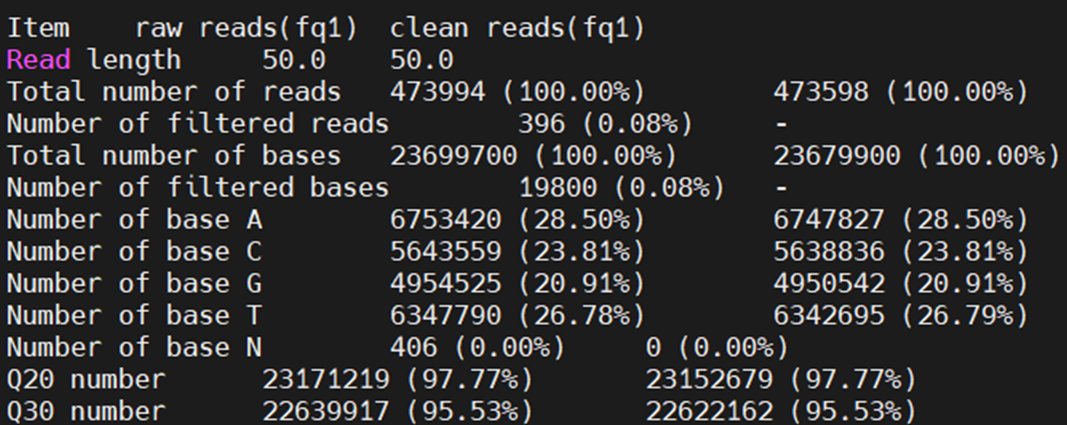
大家也可以把数据下载到本地进行查看,可在01_clean路径下找到”Basic_Statistics_of_Sequencing_Quality.txt”文件。该文件提供了过滤前后数据质量的对比统计信息,可以查看以下指标:数据总数、总碱基数、GC含量、Q20和Q30的比例等,可通过查看该文件了解数据质量与 过滤情况,截图如下
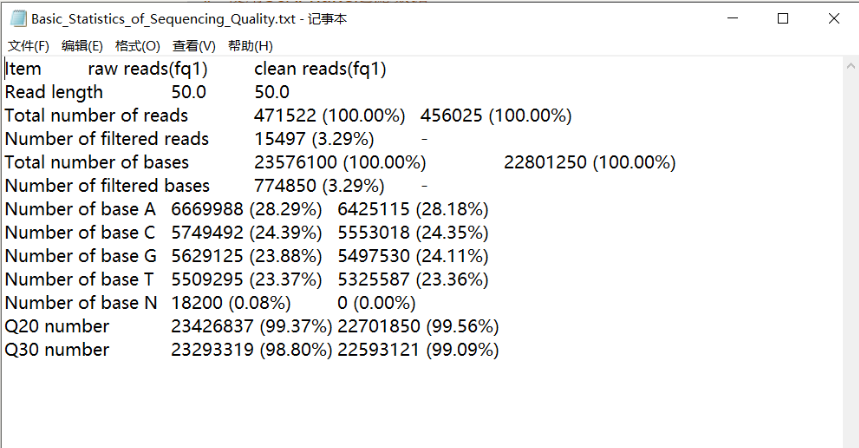
根据实验结果截图,可以得到以下信息:
总reads数量为471522,其中被过滤的reads数量为15497,占总reads的3.29%。 总碱基数量为23576100,被过滤的碱基数量为774850 ,占总碱基的3.29%。
经过筛选过后的数据如下:
碱基A的数量为6425115,占总碱基的28.18%。 碱基C的数量为2977487,占总碱基的27.04%。 碱基G的数量为5497530,占总碱基的24.11%。 碱基T的数量为5325587,占总碱基的23.36%。
Q20质量值的reads数量为22701850,占总reads的99.56%。 Q30质量值的reads数量为22593121,占总reads的99.09%。
以上结果显示,在实验过程中采取了有效的质量控制步骤,SOAPnuke filter工具过滤掉了质量较差的reads 和碱基,从而提高了实验数据的质量,以便进行进一步的分析,挖掘其中的生物信息学特征。
另外,FASTQC工具还生成了一份碱基质量值分布图报告,打开FASTQC工具生成的碱基质量值分布图,可以清晰直观地观察到每个碱基位置上的质量值情况:
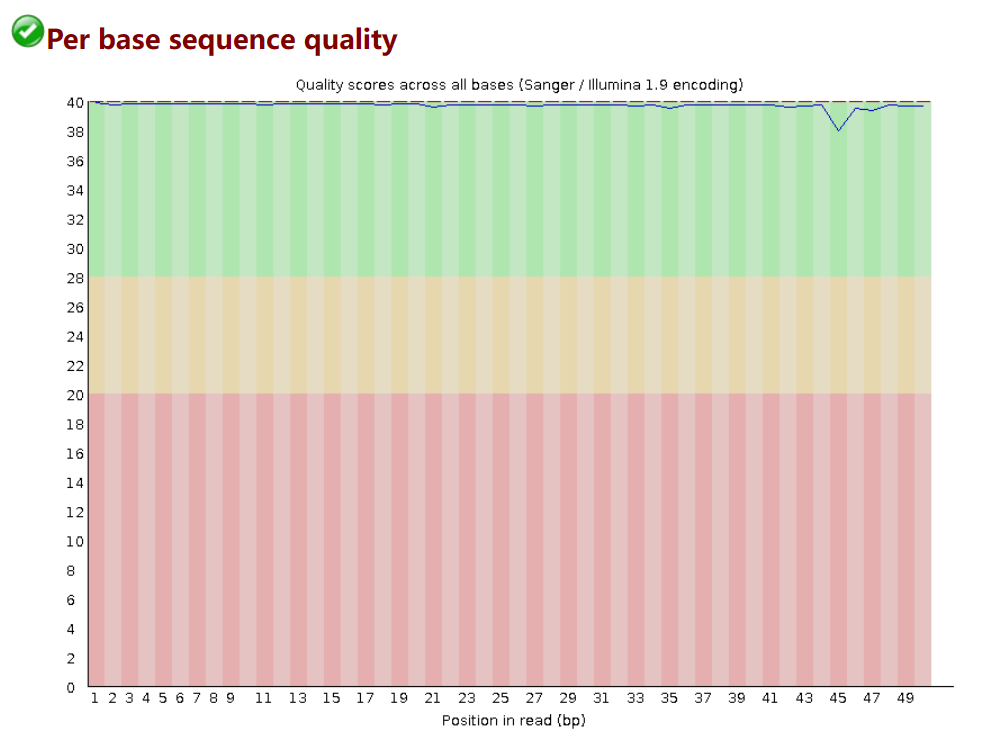
Per base sequence quality说明:各位置碱基质量,每个read各位置碱基的测序质量。横轴碱基的位置,纵轴是质量分数,Quality score=-10log10p(p代表错误率),所以当质量分数为40的时候,p就是0.0001,代表质量很高。红色线代表中位数,蓝色代表平均数,黄色是25%-75%区间,触须是10%-90%区间。若任一位置的下四分位数低于10或者中位数低于25,出现“警告”;若任一位置的下四分位数低于5或者中位数低于20,出现“失败,Fail”。
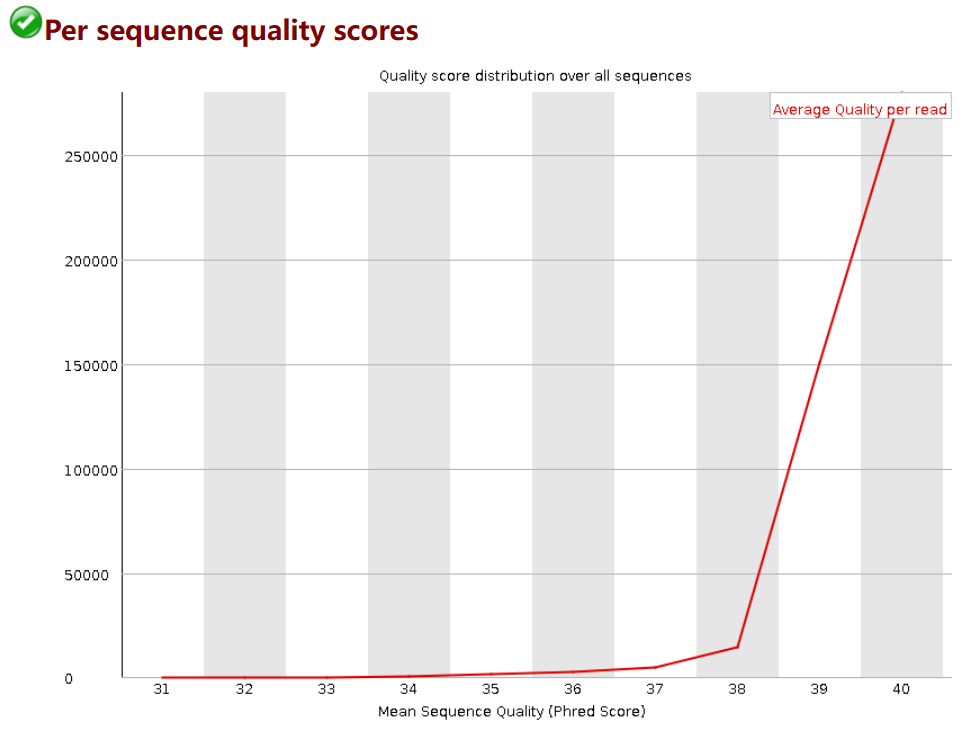
Per sequence quality scores说明:横轴为quality,纵轴为reads计数。
可以看出,小果的数据的质量比较高,质量分数在39以上。
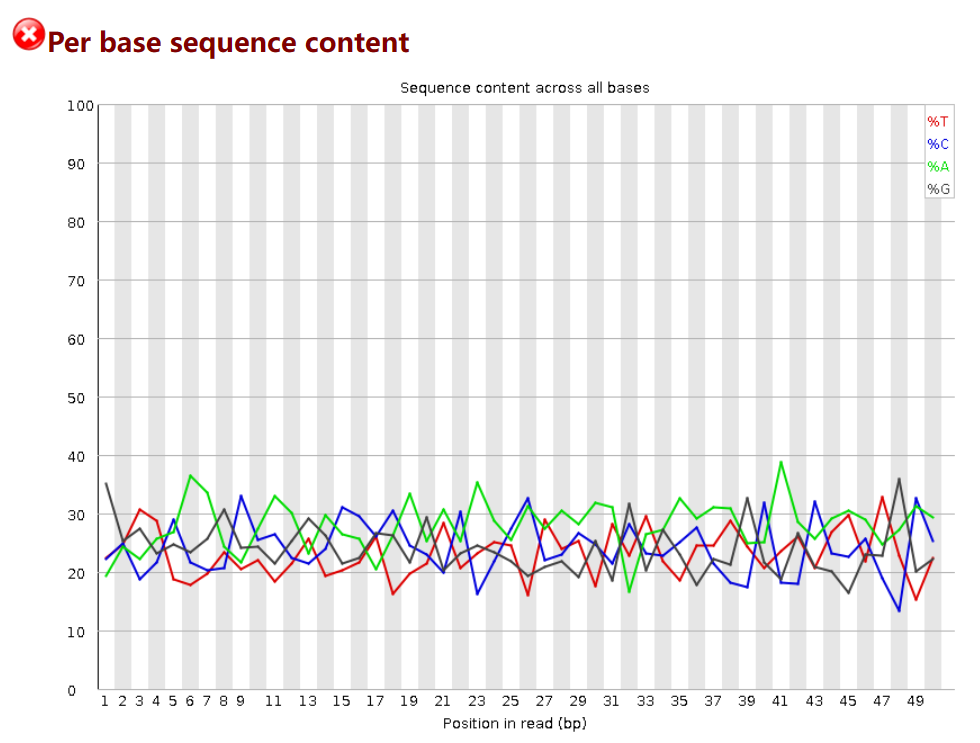
Per base sequence content说明:碱基分布,对所有reads的每一个位置,统计ATCG四种碱基的分布,横轴为位置,纵轴为碱基含量,正常情况下每个位置每种碱基出现的概率是相近的,四条线应该平行且相近。当部分位置碱基的比例出现bias时,即四条线在某些位置纷乱交织,往往提示我们有overrepresented sequence的污染。本结果每种碱基频率有明显的差别,说明有污染。当任一位置的A/T比例与G/C比例相差超过10%,报’WARN’;当任一位置的A/T比例与G/C比例相差超过20%,报’FAIL’。
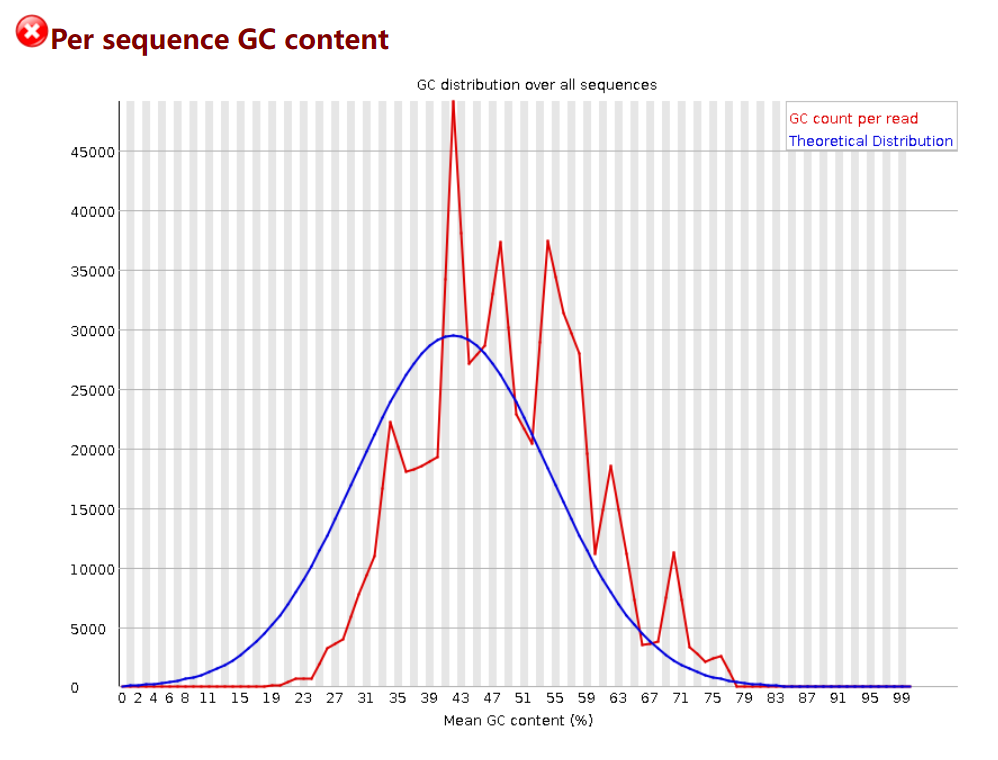
per sequence GC content说明:横轴为GC含量,纵轴为read计数。红色为实际测得,蓝色为理论分布。 偏离大于15%,报WARN;大于30%,报FAIL。 这里与标准曲线偏离较严重,小果经过查阅资料得知,如果曲线形状不符,代表文库污染。
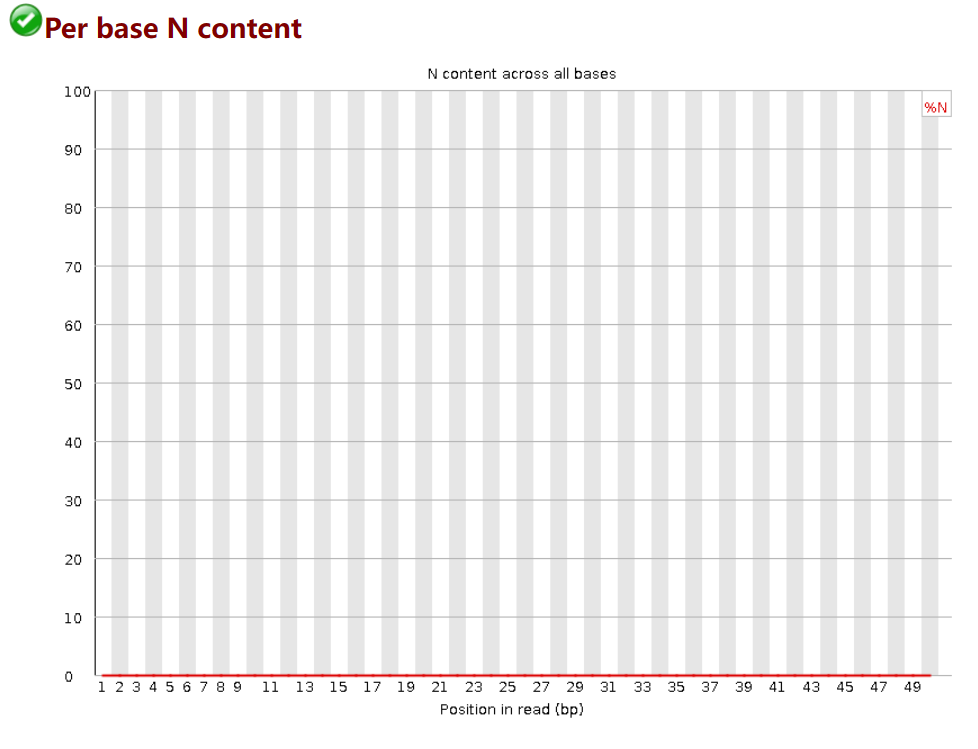
per base N content说明:N 代表测序仪不能识别的碱基,横轴代表read位置,纵轴代表占比。如果正常测序,红线应该是趋近与0的直线。当任意位置N占比大于5%,报WARN;大于20%,报FAIL。这里测序质量没有问题。
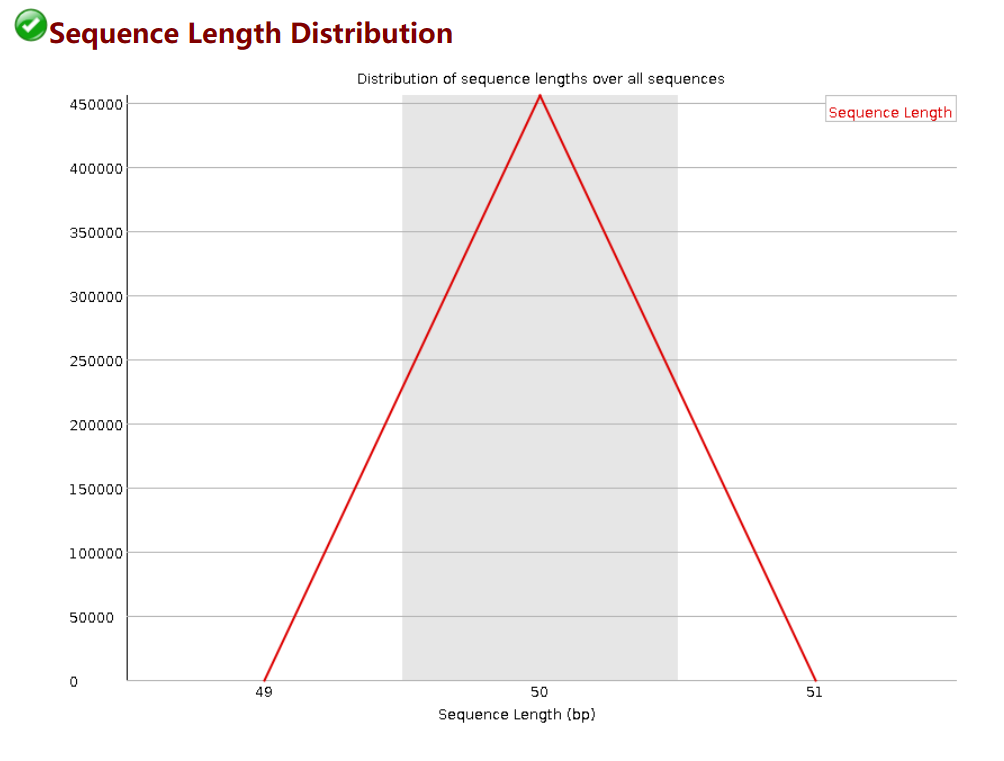
长度一致,没有问题。
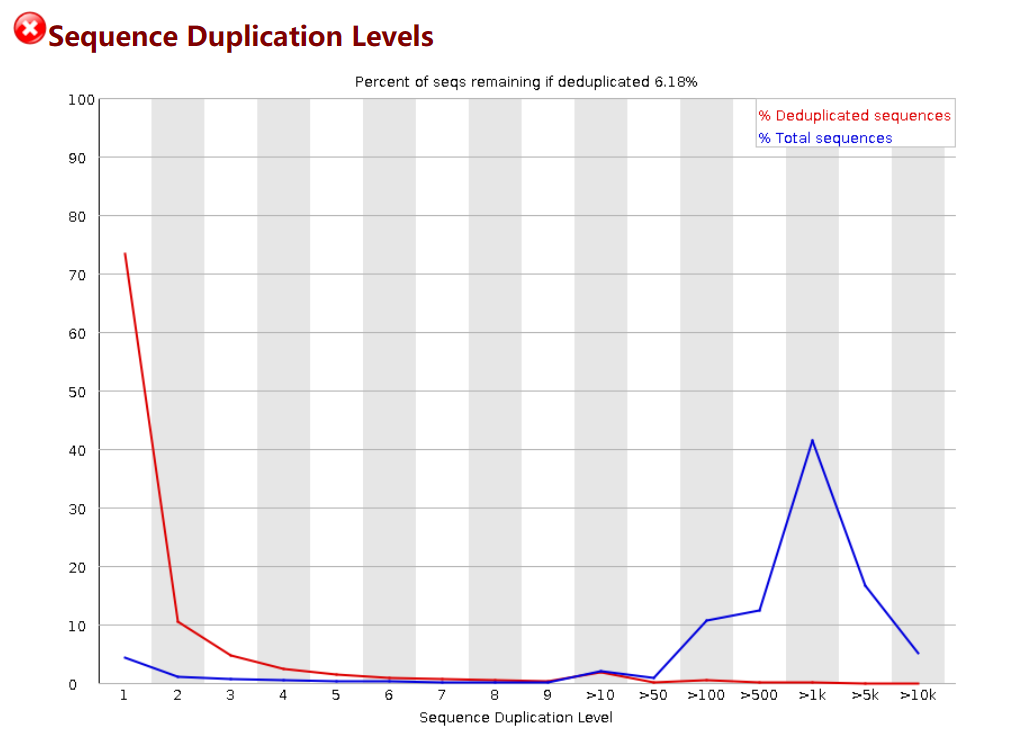
sequence duplication levels说明:横坐标为重复(duplication)的次数,纵坐标为reads的数目,以unique reads的总数作为100%。这里可能意味着存在某种技术问题,如PCR偏倚、文库构建问题或者污染等。
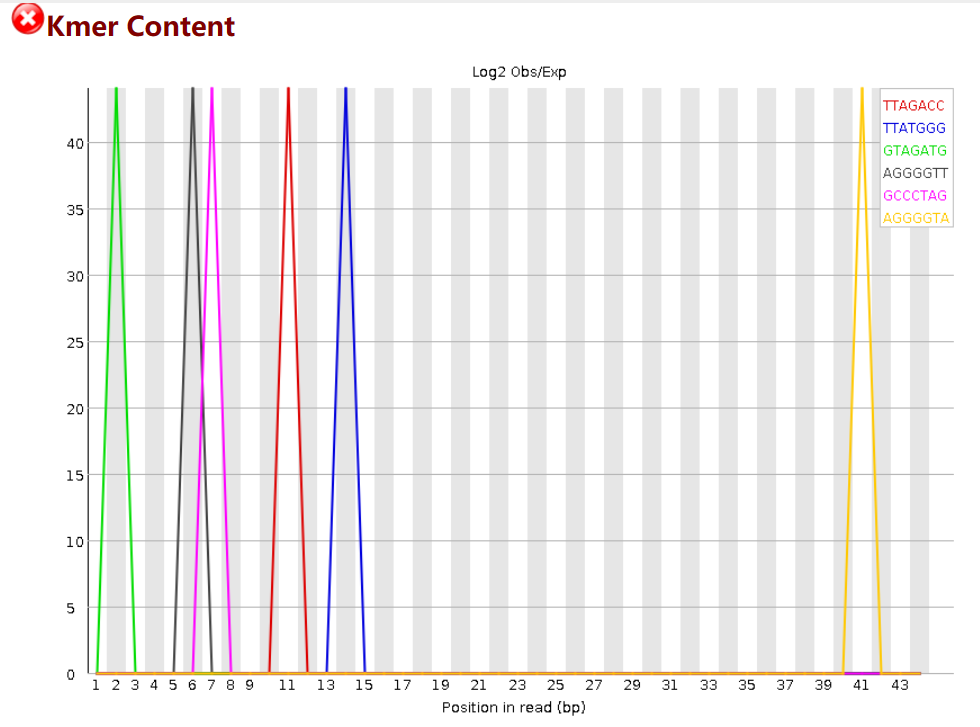
Kmer Content说明:Kmer是指连续的长度为k的核苷酸序列片段,通过计算和比较这些片段的频率,可以了解样本中不同核苷酸的相对丰度。在FastQC的Kmer Content模块中,通常会显示各个Kmer长度(例如,4、5、6等)的频率分布图。每个Kmer长度代表了不同长度的核苷酸序列片段。分析这些片段的频率分布可以得到样本中各种核苷酸组成的相对比例。这里的kmer分布有异常出现,可能是有污染的问题。
本次的分享到这里就结束啦,怎么样,质量评估的操作部分是不是很简单,而观看结果的部分是不是干货满满呢?相信小伙伴们也都是收获满满吧。生信分析经常是这样,操作很简单而分析乍一看比较复杂,但学习后也会发现其实只是“纸老虎”,只要小伙伴们肯认真钻研其实也是很简单的。
如果小伙伴们平时在生信分析的操作过程中遇到困难,欢迎大家使用小果开发的生信工具平台http://www.biocloudservice.com/home.html,大家在新接触一个知识的时候,与其先花费大量时间死啃知识点,不如先利用好工具先自己上手接触流程,在跑完一遍全流程后再返回去理解知识点,相信可以更好更快地理解,达到事半功倍的效果!
终于完成了质量评估,但后面需要我们分析的项目还有很多~下一次小果将给大家分享如何进行序列比对,小伙伴们要持续关注哦~