pbkrtest包的主要功能是提供有效的假设检验方法,用于评估混合效应模型中固定效应的显著性。它通过计算近似的p值,利用参数估计的渐近分布,来进行统计推断。这种近似方法对于大型数据集和复杂的模型结构尤其有用,因为它可以显著降低计算的复杂性。
在使用pbkrtest包时,用户可以指定混合效应模型,并使用适当的函数进行拟合。然后,可以使用summary()函数来查看模型的摘要信息,包括固定效应的估计值、标准误差和置信区间。同时,pbkrtest包还提供了anova()函数,用于比较不同模型之间的显著性差异。
与传统的假设检验方法相比,pbkrtest包的优势在于它能够考虑到混合效应模型的特殊结构,并提供了更准确的假设检验结果。传统的假设检验方法通常假设数据独立同分布,而混合效应模型则能够处理数据中存在的层级结构和相关性。此外,pbkrtest包还提供了模型诊断的功能,可以帮助用户评估模型的拟合程度和假设的合理性。
pbkrtest包的应用范围非常广泛,适用于许多领域的数据分析和研究。例如,在社会科学中,研究人员可以使用混合效应模型来分析家庭、社区或学校之间的差异对个体行为的影响。在生物医学研究中,混合效应模型可以用于考虑实验设计的重复性,从而更准确地评估治疗效果。
要使用pbkrtest包,可以在R中使用以下命令进行安装和加载:
> install.packages(“pbkrtest”) #安装pbkrtest语言包
> library(pbkrtest) #加载语言包
下面是一个使用pbkrtest包进行生物信息相关实际数据分析的示例:
假设我们正在研究一种新药对基因表达的影响。我们有一个实验数据集,其中包含了10个病人,在给予药物治疗前后,测量了他们的基因表达水平。此外,每个病人还有一些基本信息,如性别和年龄。我们想要确定药物是否对基因表达产生显著影响,并且是否存在个体间的差异。
首先,我们需要加载所需的包,并导入数据集。假设我们的数据集命名为”gene_expression.csv”,其中包含了”PatientID”(病人ID)、”Treatment”(治疗前后)、”GeneExpression”(基因表达水平)、”Gender”(性别)和”Age”(年龄)等变量。
# 加载所需的包
> library(lme4)
> library(pbkrtest)
# 导入数据集
> data <- read.csv(“gene_expression.csv”)
接下来,我们可以构建混合效应模型,其中基因表达水平是因变量,治疗和病人是固定效应,而病人是随机效应。我们可以使用lmer()函数来拟合该模型。
# 构建混合效应模型
> model <- lmer(GeneExpression ~ Treatment + (1 | PatientID), data = data)
然后,我们可以使用pbkrtest包提供的summary()函数来查看模型的摘要信息,包括固定效应的估计值、标准误差和置信区间。
# 查看模型摘要信息
> summary(model)
输出的摘要信息将提供固定效应的估计值、标准误差和置信区间等统计量。通过检查固定效应的p值,我们可以确定治疗对基因表达的影响是否显著。
此外,我们还可以使用anova()函数来比较不同模型之间的显著性差异。假设我们还想考虑性别和年龄对基因表达的影响,我们可以构建一个包含这些变量的模型,并使用anova()函数进行比较。
# 构建包含性别和年龄的模型
> model_full <- lmer(GeneExpression ~ Treatment + Gender + Age + (1 | PatientID), data = data)
# 比较两个模型的显著性差异
> anova(model, model_full)
通过比较模型的显著性差异,我们可以确定是否有必要将性别和年龄考虑在内。
在这个示例中,我们使用pbkrtest包来分析生物信息相关的实际数据。我们构建了混合效应模型,考虑了治疗和个体间的差异,并使用summary()函数查看了模型的摘要信息。此外,我们还使用anova()函数比较了不同模型之间的显著性差异。通过这些分析,我们可以得出关于药物对基因表达的影响以及其他因素的结论。
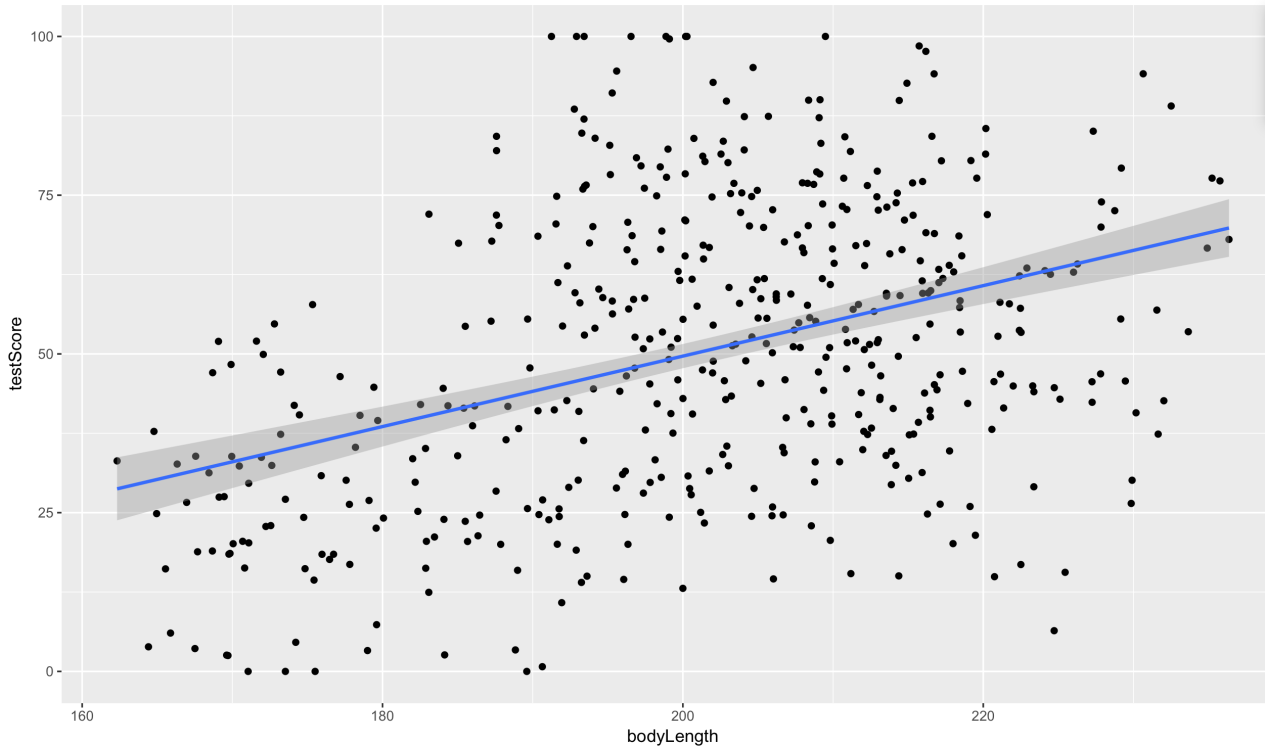
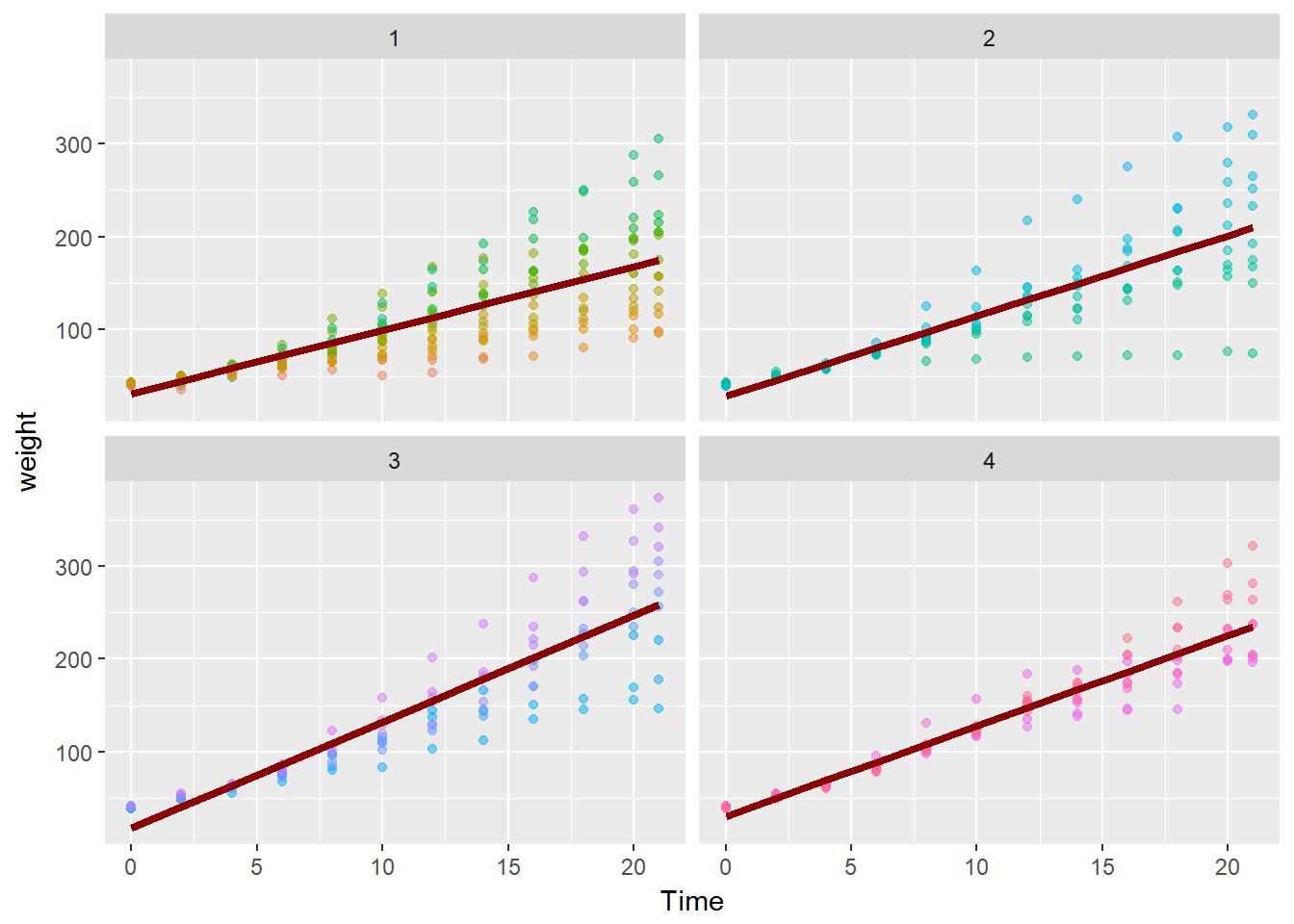
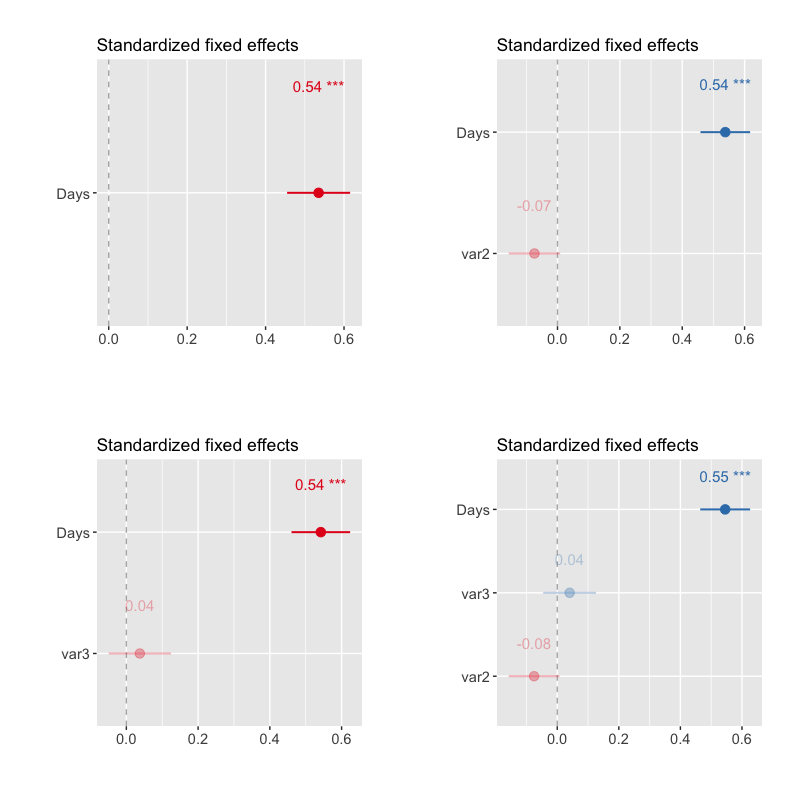
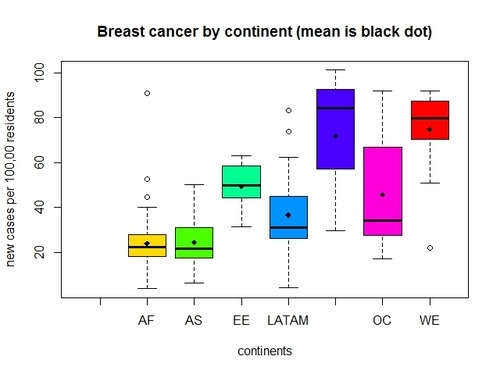
以上就是对R语言包pbkrtest的简单介绍啦,pbkrtest包为研究人员提供了一个强大的工具,用于在混合效应模型中进行假设检验和参数估计。它能够处理复杂的数据结构和相关性,提供准确的统计推断结果。通过使用pbkrtest包,研究人员可以更好地理解数据,并得出更可靠的结论。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html
References:
- https://ourcodingclub.github.io/tutorials/mixed-models/
- https://terpconnect.umd.edu/~egurarie/teaching/Biol709/Topic3/Lecture16_MixedEffectsModels.html
- https://stackoverflow.com/questions/40546716/plotting-mixed-models-regression-coefficients-in-r
- http://www.analyticsforfun.com/2014/06/performing-anova-test-in-r-results-and.html
- https://www.scribbr.com/statistics/anova-in-r/