原理简介
KEGG富集分析是一种生物信息学方法,用于解释和理解基因或蛋白质的功能和参与的代谢通路。该分析使用KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库中的注释信息,将研究中发现的差异表达基因或蛋白质与已知的代谢通路和功能进行关联。
KEGG富集分析通常包括以下步骤:1、将感兴趣的基因或蛋白质列表与KEGG数据库中的基因ID或路径way ID进行比对。2、使用统计方法,如Fisher精确检验或超几何分布法,计算在给定基因或蛋白质列表现特定代谢通路或功能的显著性。3、根据显著性水平对结果进行筛选,确定具有生物学意义的通路或功能。4、通过KEGG富集分析,研究人员可以识别与研究对象相关联的代谢通路、信号通路和功能模块,从而更好地理解基因或蛋白质在生物体内的作用和调控。
今天小果带大家实现KEGG的富集分析~
代码实现
读入一个示例数据
data <- read.delim(“Downloads/kegg_rich.txt”,header = T,na.strings = T)
#加载相应的软件包
library(tidyverse)
colnames(data)
data_1<-subset(data,p.adjust<0.05)
colnames(data_1)

还记得小果在讲R语言的颜色的时候给大家分享的paletteer包嘛,这里可以应用一下啦~
添加自定义配色方案:
library(paletteer)#配色包
d_palettes <- palettes_d_names
paletteer_d(“ggthemes::wsj_dem_rep”,n=3)
col_bar1<-paletteer_d(“ggthemes::wsj_dem_rep”,n=3)
col_bar1

画图
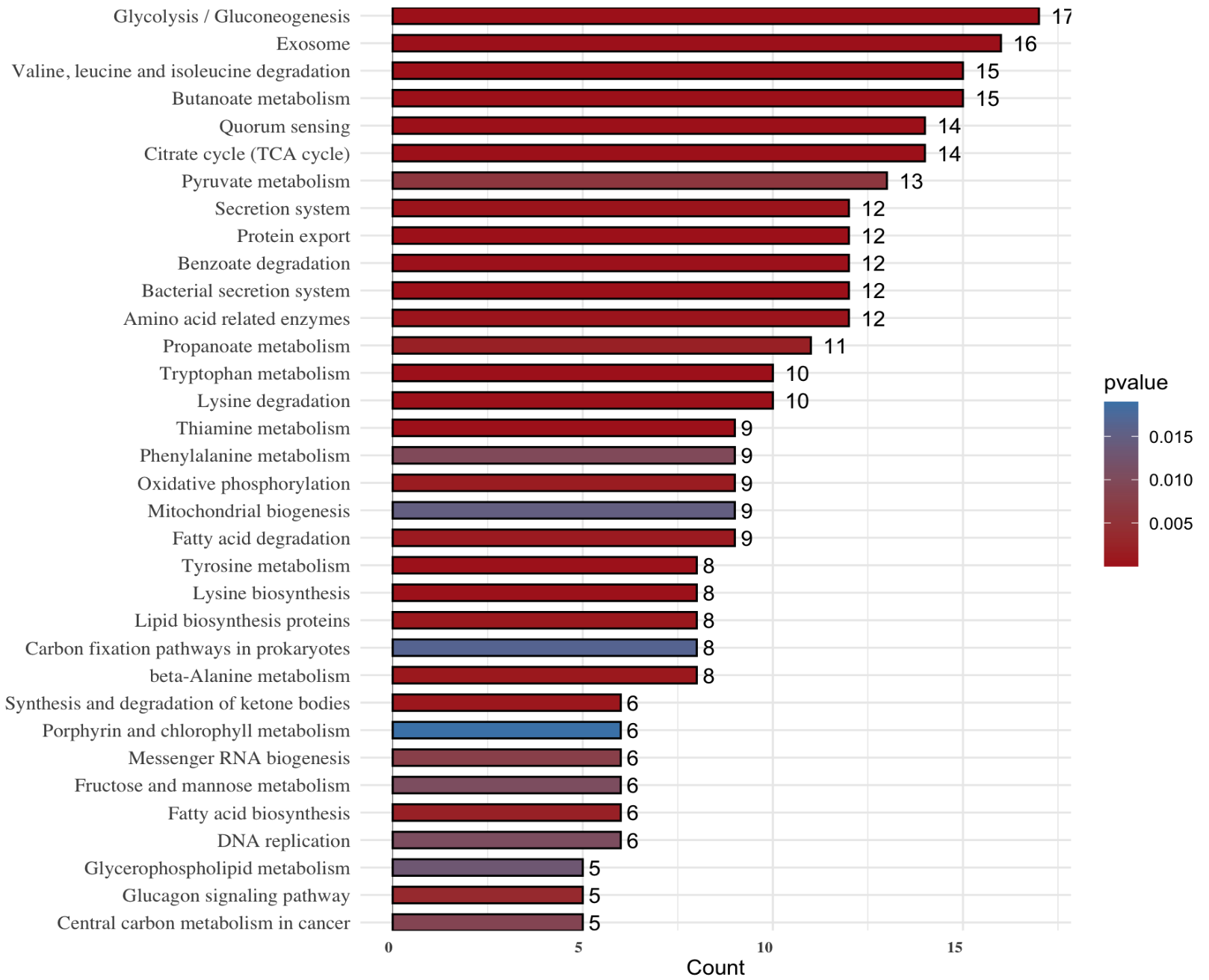
排序柱状图
简单绘制:
ggplot(data_1)+
geom_col(aes(x=Count,y=reorder(Description,Count),fill=pvalue),color=’black’,width = 0.6)
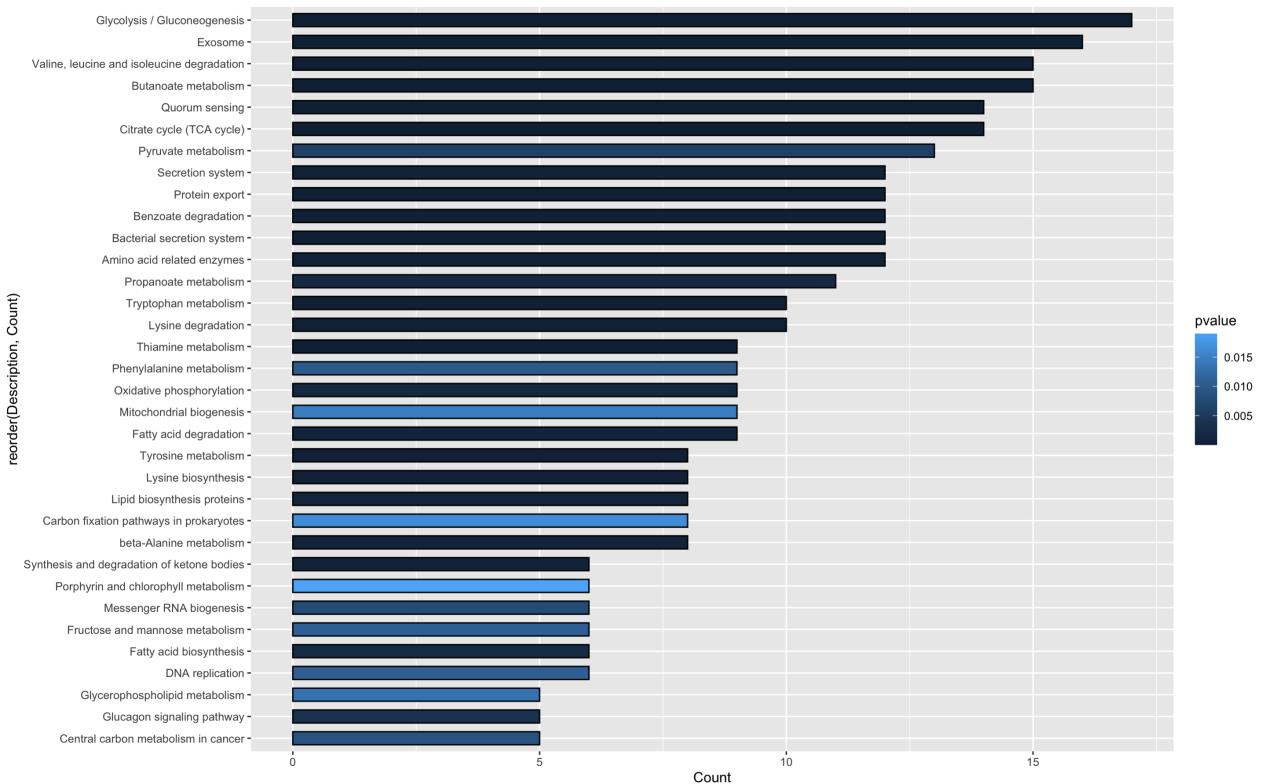
添加count、颜色、和主题
ggplot(data_1)+
geom_col(aes(x=Count,y=reorder(Description,Count),fill=pvalue),color=’black’,width = 0.6)+
geom_text(aes(x=Count,y=reorder(Description,Count),label =Count),hjust=-0.5,vjust =0.5)+
scale_fill_gradientn(colours = c(‘#B1283AFF’,’#006A8EFF’))+
theme_minimal()+
scale_y_discrete(expand = c(0,0))+
geom_vline(xintercept = 0,lwd=0.1,color=”black”)+
theme(legend.position = ‘right’)+ylab(NULL)
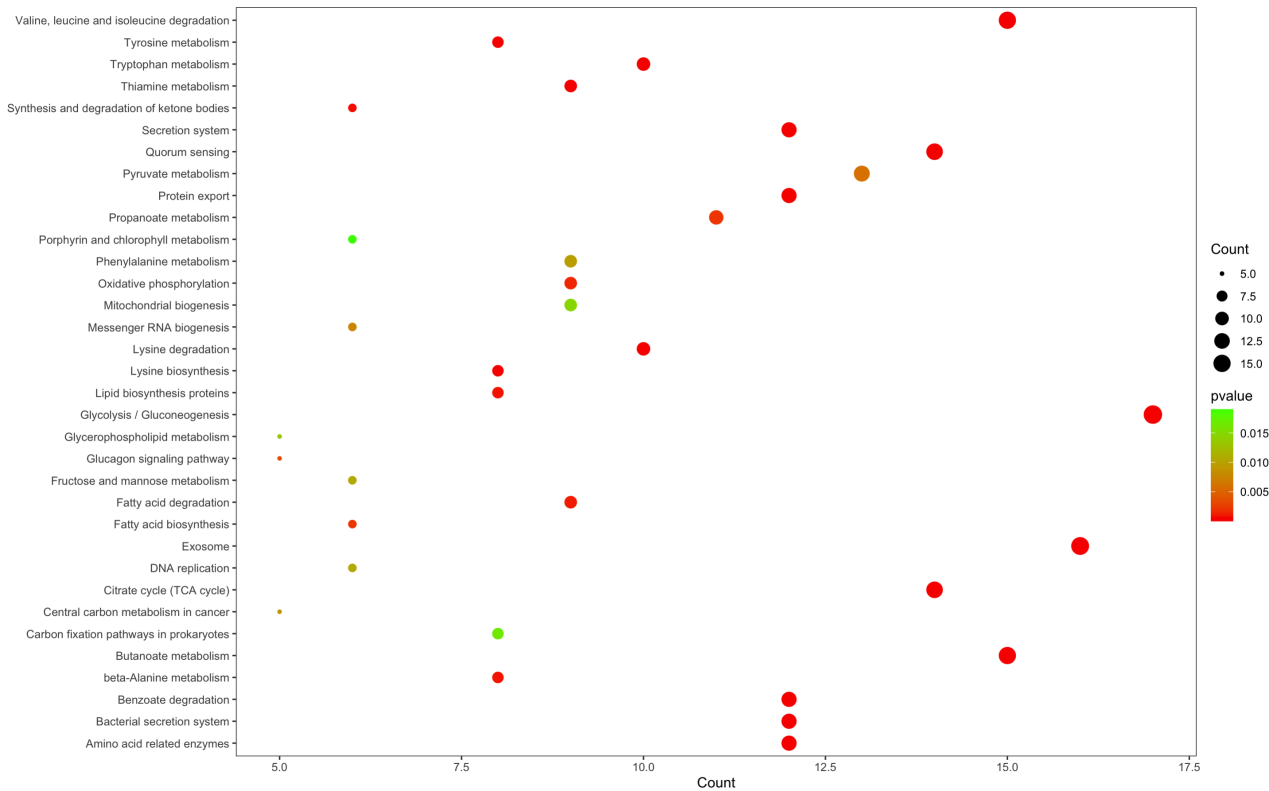
富集气泡图
ggplot(data_1)+
geom_point(aes(x=Count,y=Description,color=pvalue,size=Count))+
scale_color_gradientn(colours = c(‘red’,’green’))+
theme_test()+
ylab(NULL)
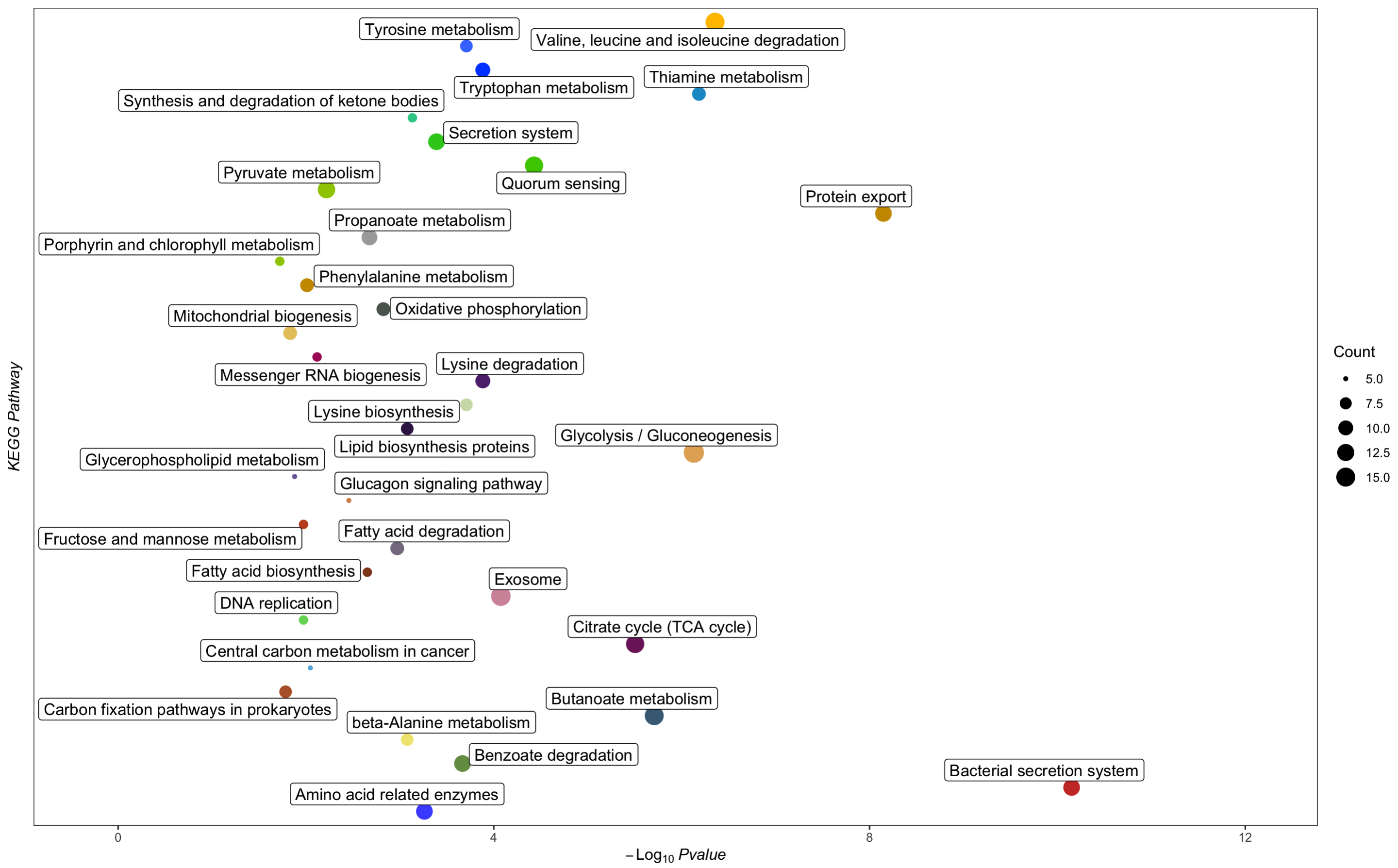
3、带有标签的气泡图
利用ggrepel中的geom_label_repel函数
#取一个-log10变成一列
data_1$logP<- -log10(data_1$pvalue)
library(ggrepel)
col_bar2<-paletteer_d(“ggsci::default_igv”,n=50) #去另外一个色块的颜色
col_bar2
colnames(data_1) 
ggplot(data_1)+
geom_point(aes(x=logP,y=Description,size=Count,color=Description))+
scale_color_manual(values = col_bar2)+
theme_test()+
guides(color=F)+
geom_label_repel(aes(x=logP,y=Description,label=Description),
size=4,nudge_y = 0.1)+
theme(axis.text.y = element_blank(),
axis.ticks.y = element_blank())+
xlim(c(min(data_1$logP)-2,max(data_1$logP)+2))+
xlab(bquote(-Log[10] ~ italic(‘Pvalue’)))+ylab(bquote(italic(‘KEGG Pathway’)))
KEGG富集分析作用
功能注释和解释:KEGG数据库提供了详细的生物通路和功能注释信息,通过KEGG富集分析可以确定基因集合与哪些生物学功能、信号通路或化学反应相关联。这有助于研究人员理解基因表达数据中的关键生物过程和调控机制。
1、生物标志物鉴定:通过比较不同实验组之间基因表达差异,在特定疾病状态下对基因进行KEGG富集分析,可以帮助鉴定与该疾病相关的生物标志物。这有助于诊断和治疗策略的开发。
2、数据可视化:KEGG富集分析结果可以通过生成图形化展示来更好地呈现。例如,使用柱状图、散点图或网络图等方式将富集通路/功能及其关联基因可视化,有助于直观地理解整体结构和相互作用。
3、假设验证和新假设生成:根据已知的KEGG富集分析结果,可以验证预先设定的假设或生成新的生物学假设。例如,如果某个通路在疾病中显著富集,则可能提示该通路在疾病发展过程中起重要作用。
4、数据挖掘和整合:通过将基因表达数据与KEGG数据库进行整合和分析,可以从大规模基因表达数据中提取有关特定通路、功能或代谢途径的重要信息。这有助于揭示潜在的调节机制、相互作用网络和生物学过程。
总之,KEGG富集分析是一种强大而广泛应用的工具,在基因组学、转录组学和代谢组学等领域中发挥着重要作用。它帮助解释基因集合在生物学过程中所扮演的角色,并为进一步研究提供了方向和洞见。
如果遇到不懂的也可以借助线上的云平台哦~http://www.biocloudservice.com/home.html
好了今天的KEGG富集分析就讲到这里,欢迎大家有问题与小果一起讨论哦~