前两次分享中小果细致地给大家讲解了变异检测的操作步骤以及如何分析变异检测得到的结果文件,并且已经给大家预告了这一次的内容:深度解析变异检测。很多小伙伴可能觉得只要懂流程、会分析就已经足够了,对于完成一般的任务来说确实如此,但是如果想要真正成长为大神,理解是万万不能缺少的哦。好了,废话不多说,我们直接开始本次的分享吧~
什么是变异检测?
变异检测是指通过高通量测序技术对某一物种个体或群体的基因组进行测序及差异分析, 获得大量的遗传变异信息,如单核苷酸多态性(single nucleotide polymorphisms,简称SNP)、插入缺失(Insertion–deletion mutations,简称InDel)、结构变异(structural variation,简称SV)、拷贝数变异(copy number variation,简称CNV)等用于开发分子标记建立遗传多态性数据库,为后续揭示进化关系、挖掘功能基因、理解疾病的发病机制、预测疾病风险、指导个性化治疗等奠定数据基础。
以下是变异检测的一般步骤:
- 基因组测序:通过高通量测序技术,获取个体的基因组数据。目前常用的测序方法包括全外显子组测序(whole-exome sequencing)和全基因组测序(whole-genome sequencing)。
- 数据比对:将测序数据与参考基因组进行比对,找到与参考序列不同的位置。比对可以使用多种算法,例如Burrows-Wheeler Transform (BWT) 算法。
- 变异标注:对比对结果进行分析和注释,识别可能的变异位点。常见的变异类型包括单核苷酸变异(Single Nucleotide Variants, SNVs)、插入/缺失(Insertions/Deletions, Indels)和结构变异(Structural Variants, SVs)等。
- 功能注释:对标记出的变异位点进行功能预测和注释,评估其可能的影响。这可以包括确定变异是否在编码区域、是否导致氨基酸改变(非同义突变、错义突变等)、是否在调控元件上等。
- 进一步分析:识别与特定性状、疾病或药物反应相关的变异。这可能涉及将变异与已知数据库进行比较,如1000 Genomes Project、ClinVar、dbSNP等。
- 结果解读:将检测到的变异结果与临床数据结合,以诊断疾病或指导个性化治疗。这涉及到对变异的致病性、遗传模式和风险评估进行综合分析,有时还需要进一步的实验验证。
为何检测DNA?
为什么变异检测要检测DNA呢?如果要溯其根源,就绕不开中心法则。中心法则是指遗传信息从DNA传递给RNA,再从RNA传递给蛋白质的转录和翻译的过程,以及遗传信息从DNA传递给DNA的复制过程,这个过程决定了蛋白质的特异性。这是所有有细胞结构的生物所遵循的法则。
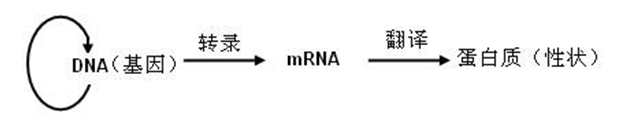
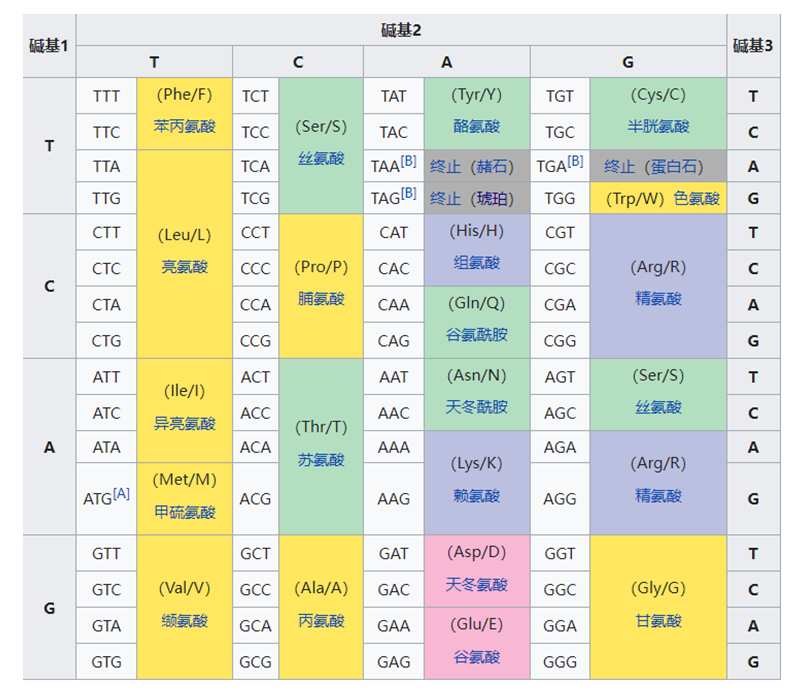
遗传密码(Genetic code)又称遗传编码,是遗传信息的传递规则,将DNA或信使RNA(mRNA)序列以三个核苷酸为一组的“密码子(codon)”翻译为氨基酸序列,以用于指导蛋白质合成。
遗传密码以DNA密码子表的形式表示,这是因为在细胞核糖体制造蛋白质时,指导合成蛋白质的是mRNA。mRNA的序列则由基因组DNA决定。随着计算生物学和基因组学的兴起,现今可在DNA水平上发现大多数基因,因此DNA密码子表变得愈加有用。标准密码子表如下:
变异的概念和分类
人类基因组上的变异主要分为三大类:
1. SNV(Single Nucleotide Variant,单核苷酸变异)是指在基因组中发生的单个碱基的替代,导致了DNA序列的变化。这种变异是最常见的基因组变异类型之一,也是人类遗传变异的主要形式之一。当SNV在人群中的频率大于1%时被称为单核苷酸多态性,简称SNP;
2. Indels,是Insertion和Deletion的简称,表示在基因组上某个位置上所发生的较短长度的线性片段的插入或缺失,长度通常在50bp以下,这个长度范围的变异通常可以采用Smith-Waterman的比对算法来精准获得,可以在目前短读长的测序数据中较好的检测出来;
3. 基因组结构性变异(Structure Variations,简称SVs),包含长度在50bp以上的长片段序列的插入或者缺失、串联重复、染色体倒位。
人类遗传变异类型众多,但SNP变异可解释近90%的个体表型差异,因此成为众多疾病研究关注的热点,也是小果给大家分享的重点。
SNP在群体中的发生频率不小于1 %,其种类包括单个碱基的替换、插入和缺失等类型,其中替换又分为转换和颠换:
转换:同型碱基之间的替换,即嘌呤与嘌呤( G/A) 、嘧啶与嘧啶(C/ T) 间的替换;
颠换:发生在嘌呤与嘧啶(A/T、A/C、C/G、G/T) 之间的替换。
依据排列组合原理,SNP 一共有6种替换情况,即A/ G、A/ T、A/ C、C/ G、C/ T 和G/ T ,但事实上,由于不同碱基的化学结构和分子特性有差异,导致转换的发生频率多于颠换;在转换当中,C/T 转换又多于G/A转换。人类基因组上共有约300万个SNP位点,每隔100至300个碱基就会发生一处SNP变异。每3个SNP变异中有2个会是C/T转换。
SNP的功能分类
根据SNP变异的发生是否会影响个体表型,我们可将其分为2类:
同义突变(Synonymous Mutation)是指在基因组中发生的一种变异,导致一个密码子被另一个编码相同氨基酸的密码子替代,从而不会改变蛋白质的氨基酸序列。
一般来说,同义突变产生的变异是无害的,因为它们通常不会影响蛋白质的结构和功能。然而,同义突变也可能对基因表达和调控产生影响。例如,某些同义突变可能会导致mRNA的稳定性、剪接或转运发生改变,从而影响蛋白质的表达水平。此外,虽然同义突变不直接改变氨基酸序列,但它们可能会影响蛋白质的速度、折叠能力或与其他分子的相互作用。
非同义突变(Non-synonymous Mutation)是指在基因组中发生的一种变异,导致一个密码子被另一个编码不同氨基酸的密码子替代,从而改变了蛋白质的氨基酸序列,这种改变常是导致生物性状改变的直接原因。
非同义突变又可以分为错义突变、无义突变两种类型。错义突变是指编码的某种氨基酸的密码子变成另一种氨基酸密码子,使得多肽链的氨基酸种类和序列发生改变,错义突变通常会使多肽链丧失原有功能。无义突变是指编码某一氨基酸的密码子变成终止密码子UAA、UGA或UAG,导致多肽链翻译中止,从而形成一条不完整的多肽链,使蛋白质的生物活性和功能改变。
通常我们还可按应用对SNP进行分类,以下是常见的几类:
(1)个体识别SNP(IISNPs)
SNP作为第三代遗传标记,具有较高遗传稳定性,几乎为零的循环突变率、所需扩增片段长度短、遍布全基因组,更加适用于高度降解DNA样本的鉴定。但是由于SNP为双等位基因,其多态性的信息含量低,需要联合多个位点才能达到个体识别的要求。在个体识别SNP位点的筛选研究中,耶鲁大学的Kidd实验室开展了系统深入的研究,并于2006年界定了筛选个体识别SNP位点的标准:1.等位基因的平均杂合度>=0.4;2.不同人群间等位基因频率的差别小,Fst<0.06;3.筛选的SNP位点之间互不连锁。
(2)祖先信息SNP(AIMSNPs)
在人类基因组的SNPs中有一部分是与种族起源相关的,不同人群之间基因频率差异非常大的多态性基因位点,被称为祖先信息标记-AIMs(Ancestry Informative Markers). AIMs包含有种群结构、种内和种间差异相关信息,可以定量的估计某个体可能的地域种族来源。本科课程中涉及的AIMs的筛选标准[6]:1.筛选出来的位点构建的体系符合H-W平衡和连锁平衡;2.人群特异性位点的选择中次等位基因频率(MAF)>0.01;3.等位基因频率差异(AFD)>0.5;4.群体遗传分化指数Fst>0.3;
(3)表型相关SNPs
SNP是影响基因表达和功能的最主要遗传标记类型,能够预测个体的外部可视化特征,比如身高、肤色、瞳孔颜色、发色、脸部形态等。
(4)连锁信息性SNP
线粒体(mtDNA)及Y染色体上的遗传标记带有家族特征,又被称为“谱系标记”。Y染色体作为父系遗传,具有单倍型保持完整、突变率低、遗传稳定的特点,所以适合用作法医遗传标记,也可以用于人类进化中的物种起源、物种迁移以及遗传推断。由于集中在mtDNA和Y染色体上的SNPs,对于个体识别能力较低,所以限制了这一类SNPs的应用范围。
SNP与表型的关系
表型(phenotype),又称性状,是指一个生物体(或细胞)可以观察到的性状或特征,是特定的基因型与环境相互作用的结果。包括个体形态、功能等各方面的表现,如身高、肤色、血型、酶活力、药物耐受力乃至性格等。经典遗传学(genetics)是指由于基因序列改变(如基因突变等)所引起的基因功能的变化,从而导致表型发生可遗传的改变,基因对形状的控制可以通过酶的表达或者蛋白质的合成来实现,生物的表型主要通过蛋白质来表现。SNP也决定了生物表型的多样性。而表观遗传学(epigenetics)则是指在DNA序列没有发生改变的情况下,基因功能发生了可遗传的变化,并最终导致了表型的变化。
SNP是影响基因表达和功能的最主要遗传标记类型,不仅能够预测个体的外部可视化特征,还可以对个体的营养代谢能力以及疾病风险进行预测。
为了更直观的感受,小果举例一些控制人类乳糖代谢能力、运动能力以及耳垢类型的SNP如下,感兴趣的小伙伴们可以去查阅资料哦:
| 基因 | SNP | 基因型 | 表型 |
|---|---|---|---|
| MCM6 | rs4988235 | GG | 乳糖不耐受 |
| MCM6 | rs4988235 | AA | 乳糖耐受 |
| MCM6 | rs4988235 | GA | 乳糖耐受 |
| MCM6 | rs182549 | CC | 乳糖不耐受 |
| MCM6 | rs182549 | TT | 乳糖耐受 |
| MCM6 | rs182549 | CT | 乳糖耐受 |
| ACTN3 | rs1815739 | TT | 耐力型 |
| ACTN3 | rs1815739 | CC | 爆发型 |
| ACTN3 | rs1815739 | CT | 爆发型 |
| ALDH2 | rs671 | GG | 喝酒不会有或有较轻的脸红反应 |
| ALDH2 | rs671 | AA | 喝酒会有脸红反应 |
| ALDH2 | rs671 | AG | 喝酒不会有或有较轻的脸红反应 |
| ABCC11 | rs17822931 | CC | 很可能为湿型耳垢 |
| ABCC11 | rs17822931 | TT | 很可能为干型耳垢 |
| ABCC11 | rs17822931 | TC | 很可能为湿型耳垢 |
以上就是小果本次为大家带来的变异检测的全部信息啦~很多小伙伴看完前两期内容后虽然会操作和分析结果,但是对细节方面还有些一知半解,现在是不是豁然开朗、融会贯通了呢?如果小伙伴们平时在生信分析的操作过程中遇到困难,欢迎大家使用小果开发的生信工具平台http://www.biocloudservice.com/home.html哦。本次的分享就到这里啦,我们下次再见~