上期小果带大家介绍了Cell Ranger处理分析后输出的文件,并解读了网页版的结果报告。虽然从网页版的报告中我们能够的到诸多的信息,例如:单细胞质控,测序,比对等信息,但是我们不能很好的进行样本的数据探索。这一期小果带大家使用10×genomics平台官方出的Loupe Browse来进行数据探索。
Loupe Browser下载
软件我们可以通过10×的官网下载到。下载安装好即可。
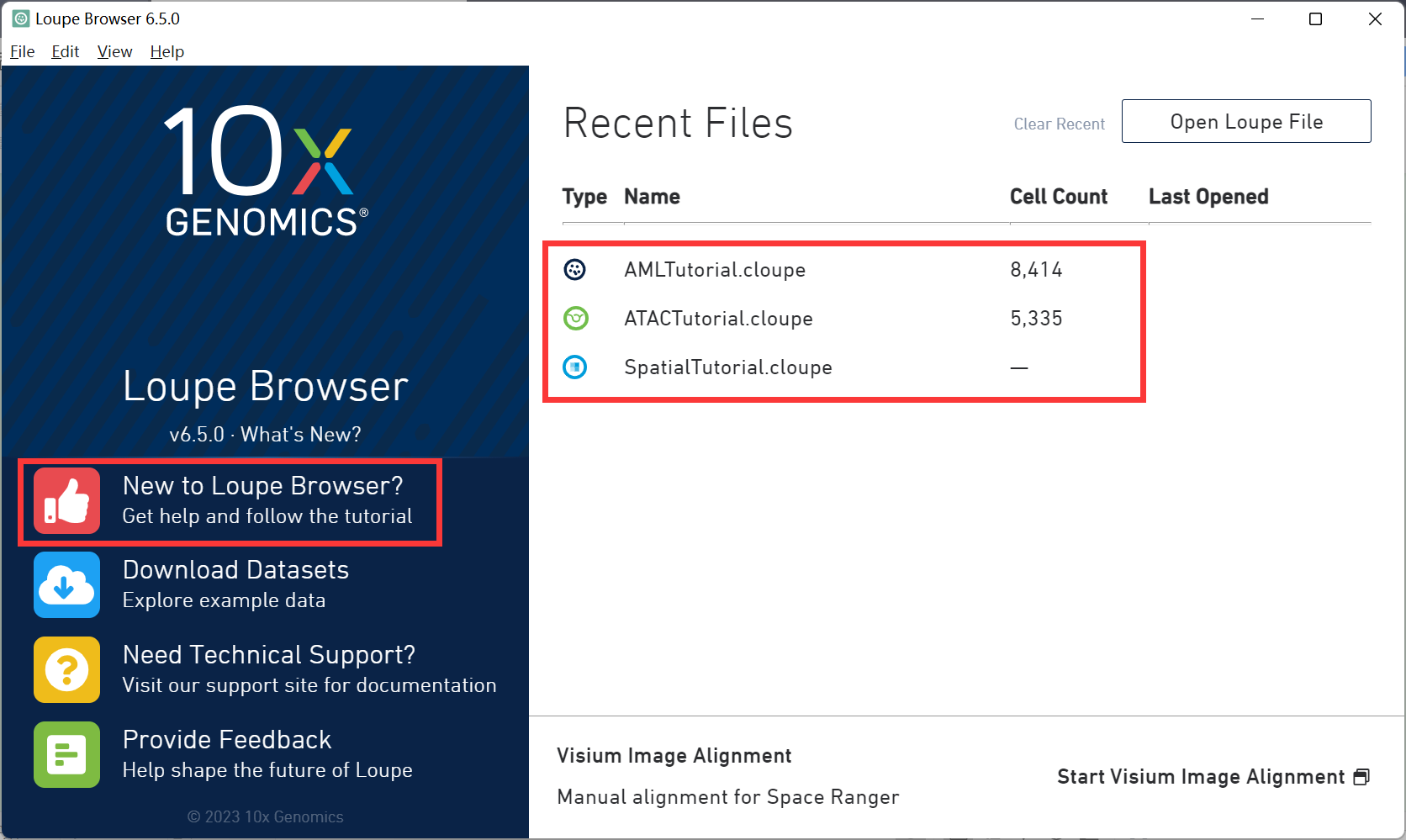
中间有自带的案例数据,有ATAC数据以及空间转录组数据。左边有详细的新手教程。并且可以下载其他的示例数据集。下方有反馈的途径。
Loupe browse教程
Loupe browse较网页板的分析报告,功能更多,更易于数据可视化与多维的数据探索。
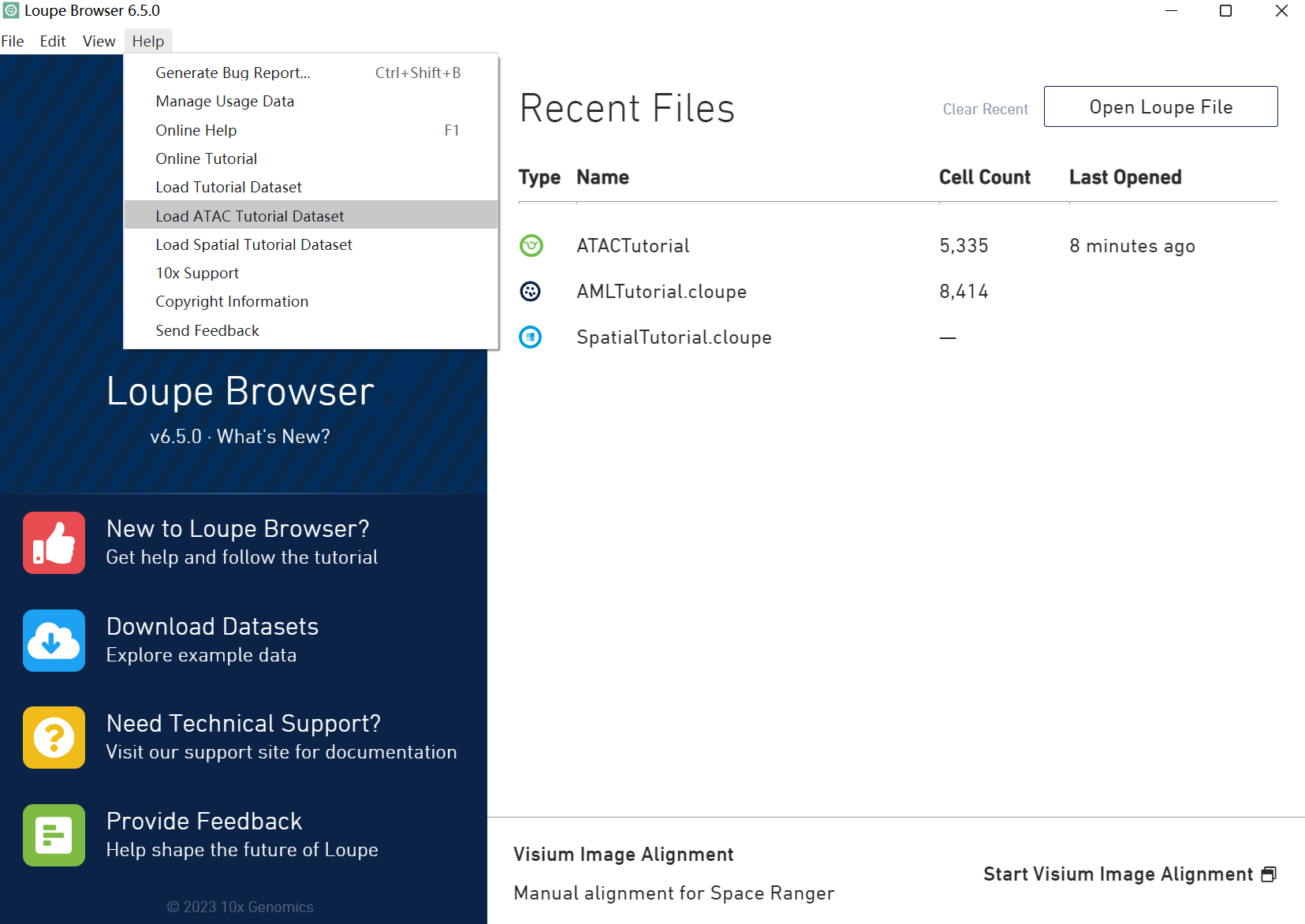
选择加载教程数据集。
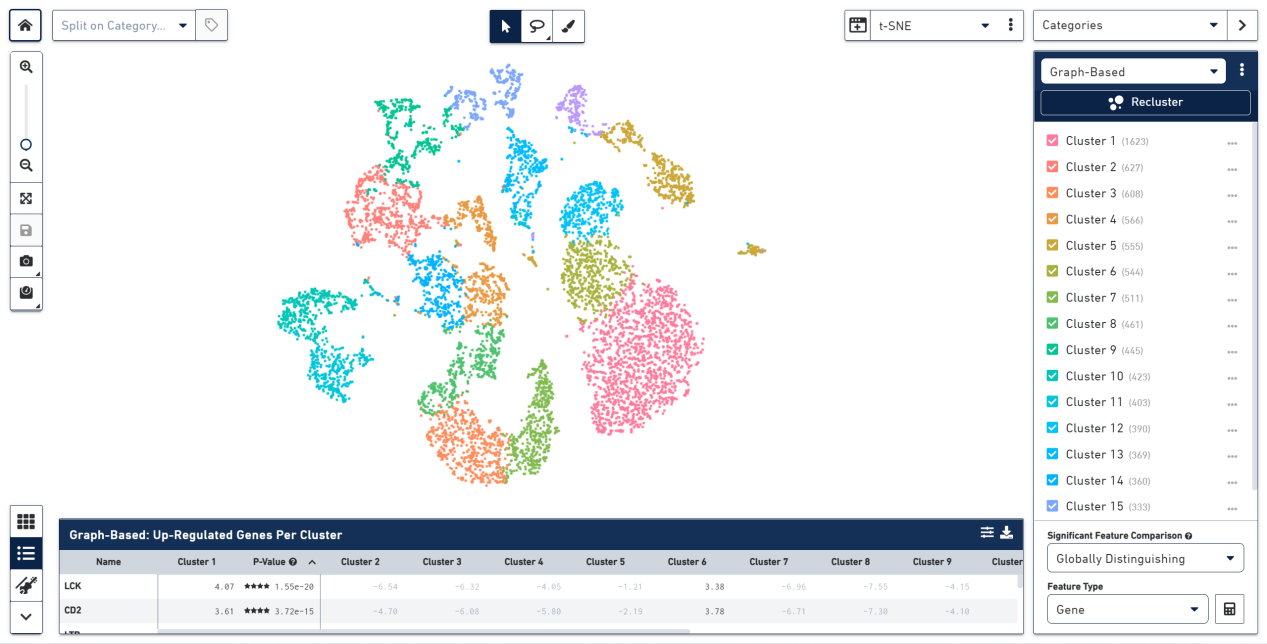
这是主界面。图中的每个点代表一个细胞,对其进行分类。示例的数据分了16组。
左边有探索数据的工具 。
上方有套索工具,可以对细胞群进行选择,进行进一步的操作。

右上角可以切换图形。
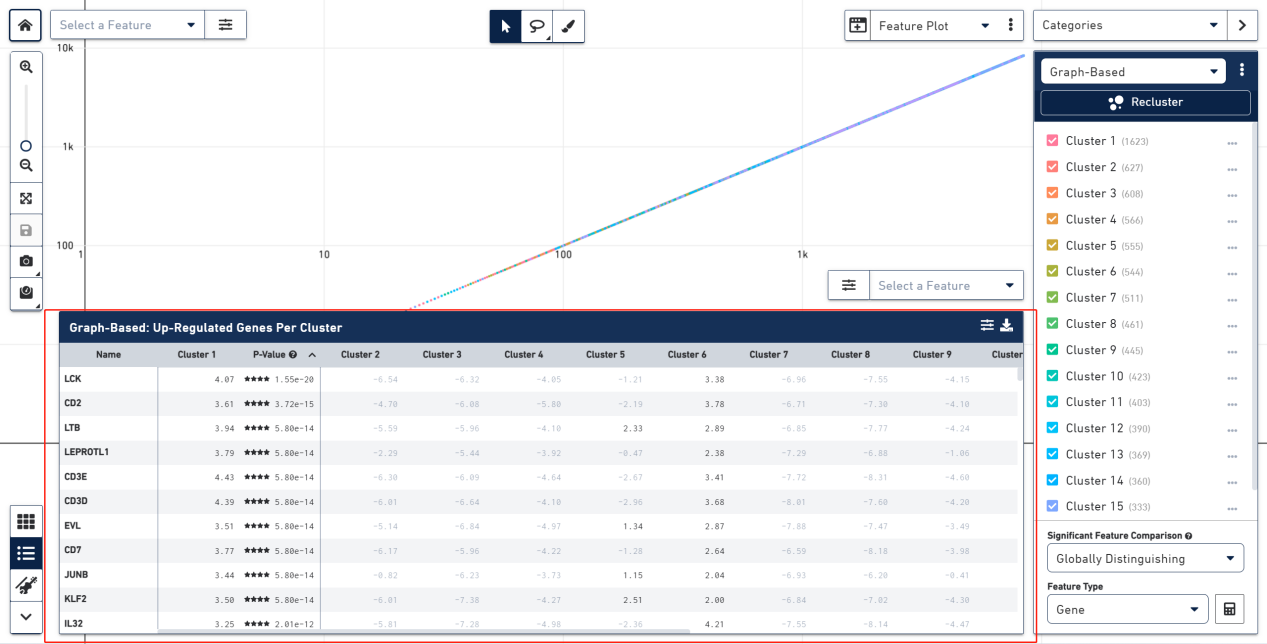
下方是基因的面板。
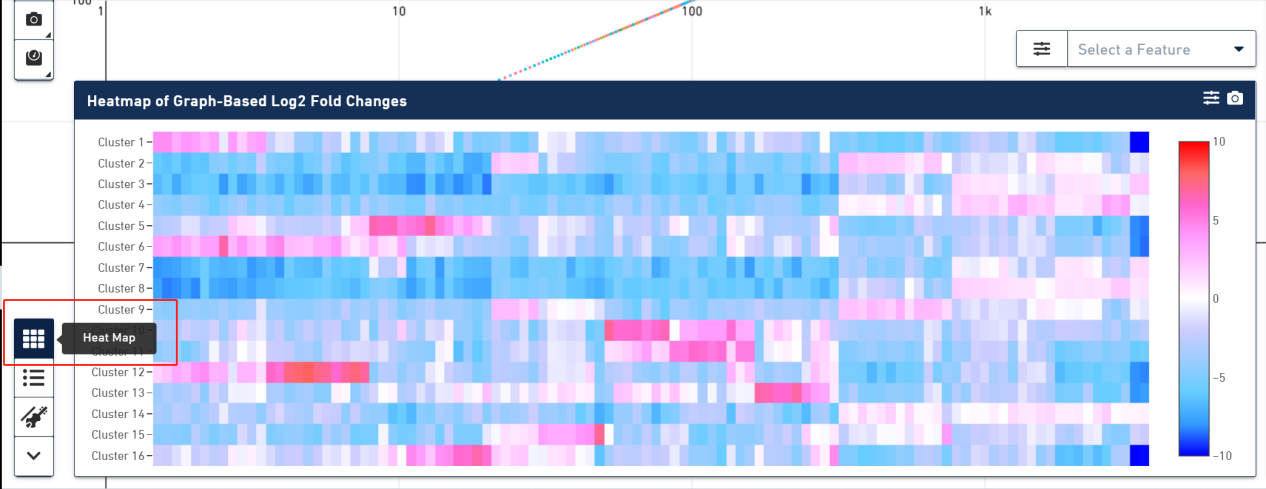
点击左侧网格图标,可以一键将基因显示成热图。

我们选择LCK基因,然后点击左侧小提琴图标。可以绘画出小提琴图,这个基因在各个细胞组中的基因表达情况,是如何分布的。
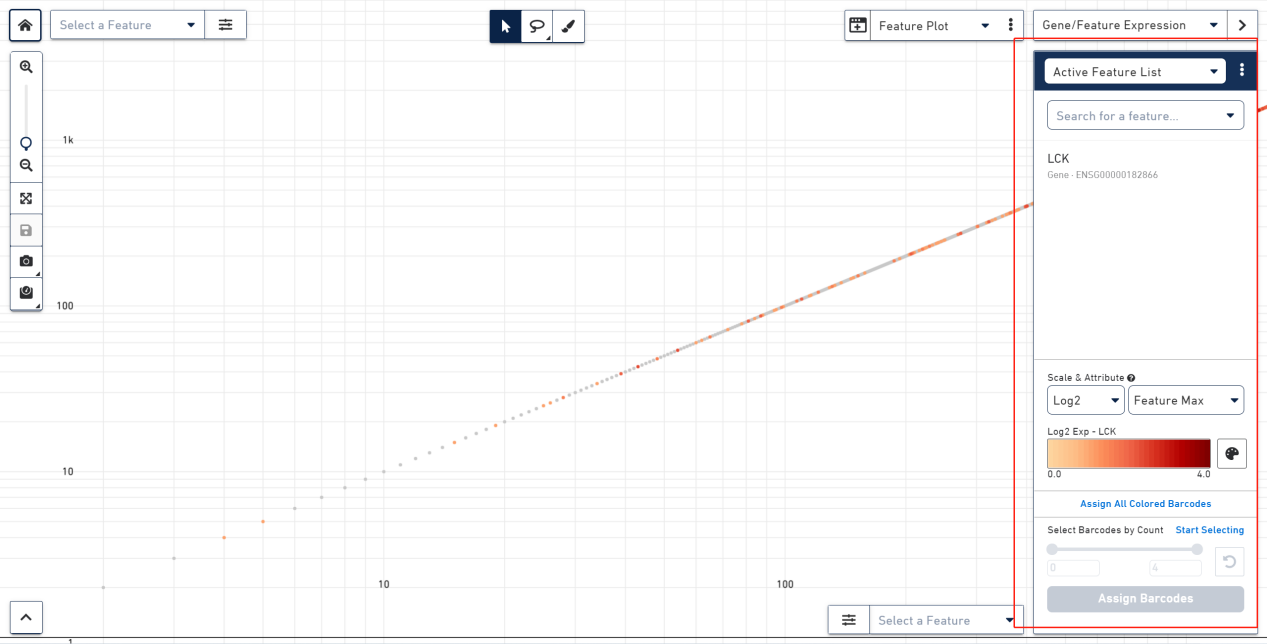
右侧框为切片器,我们可以选择或筛选感兴趣的基因。
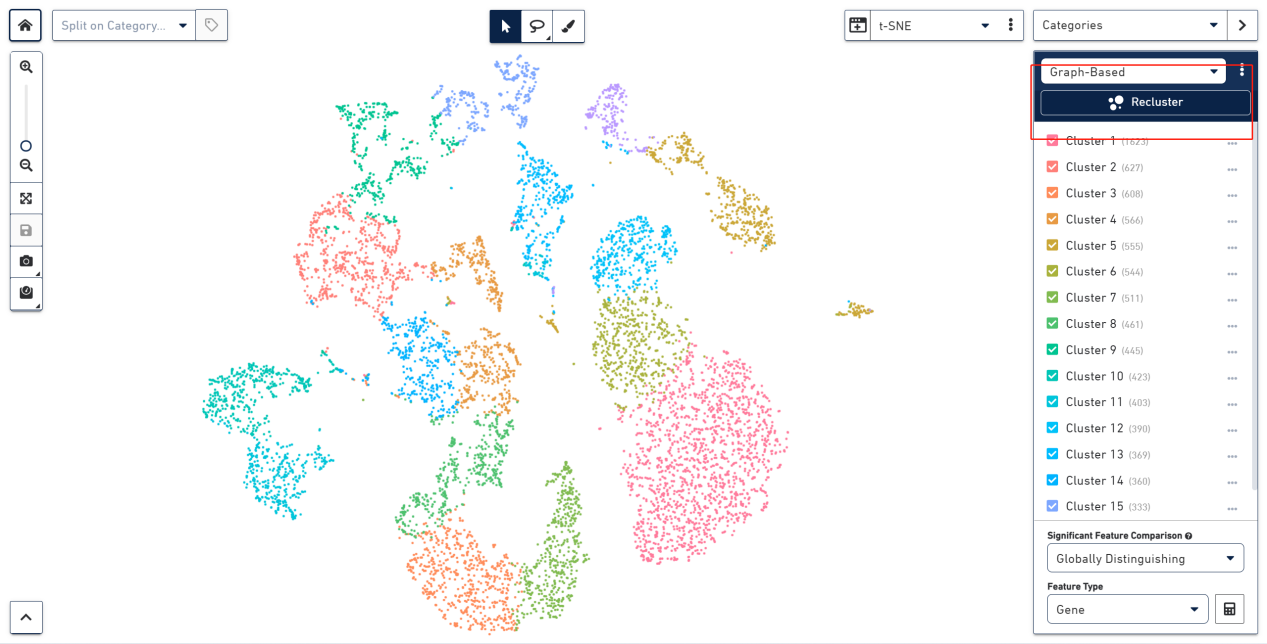
我们可以通过选择Recluster对细胞重新进行聚类。
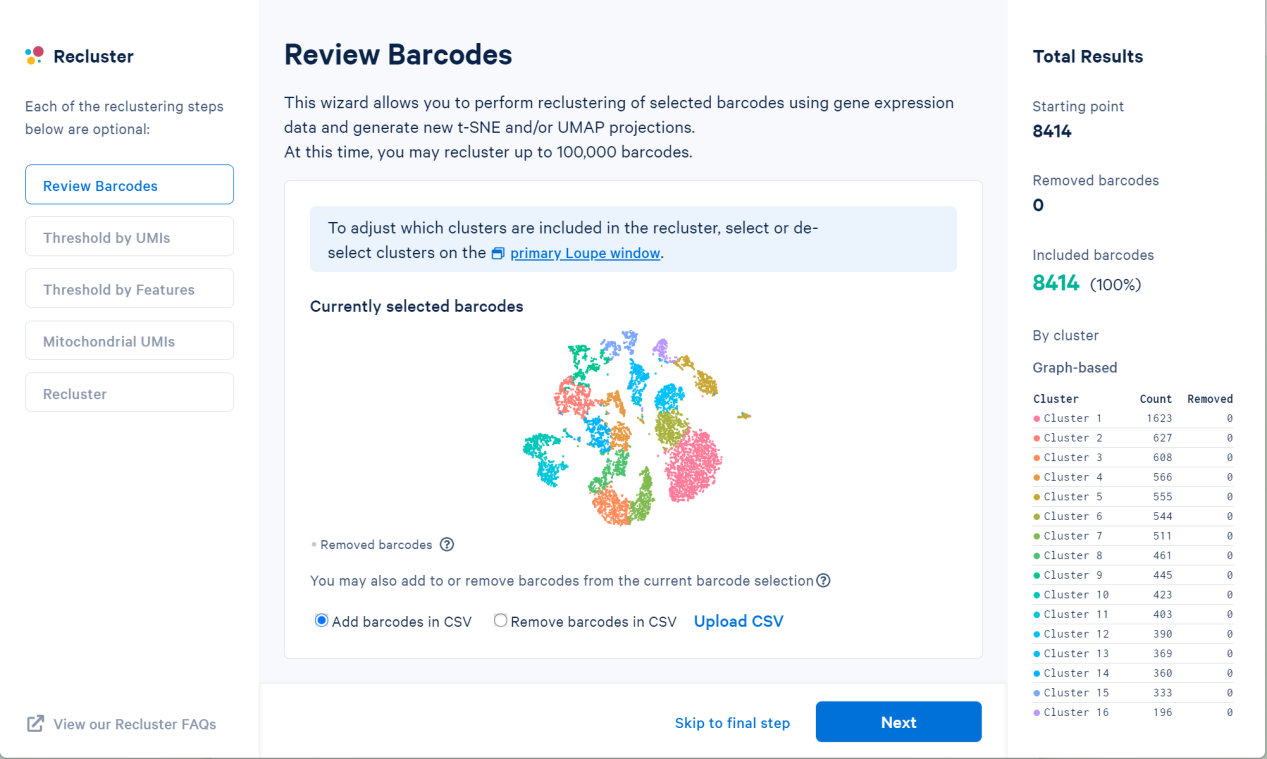
选择不同的算法。下方可以根据提供的barcodes列表,选择或者去除细胞。
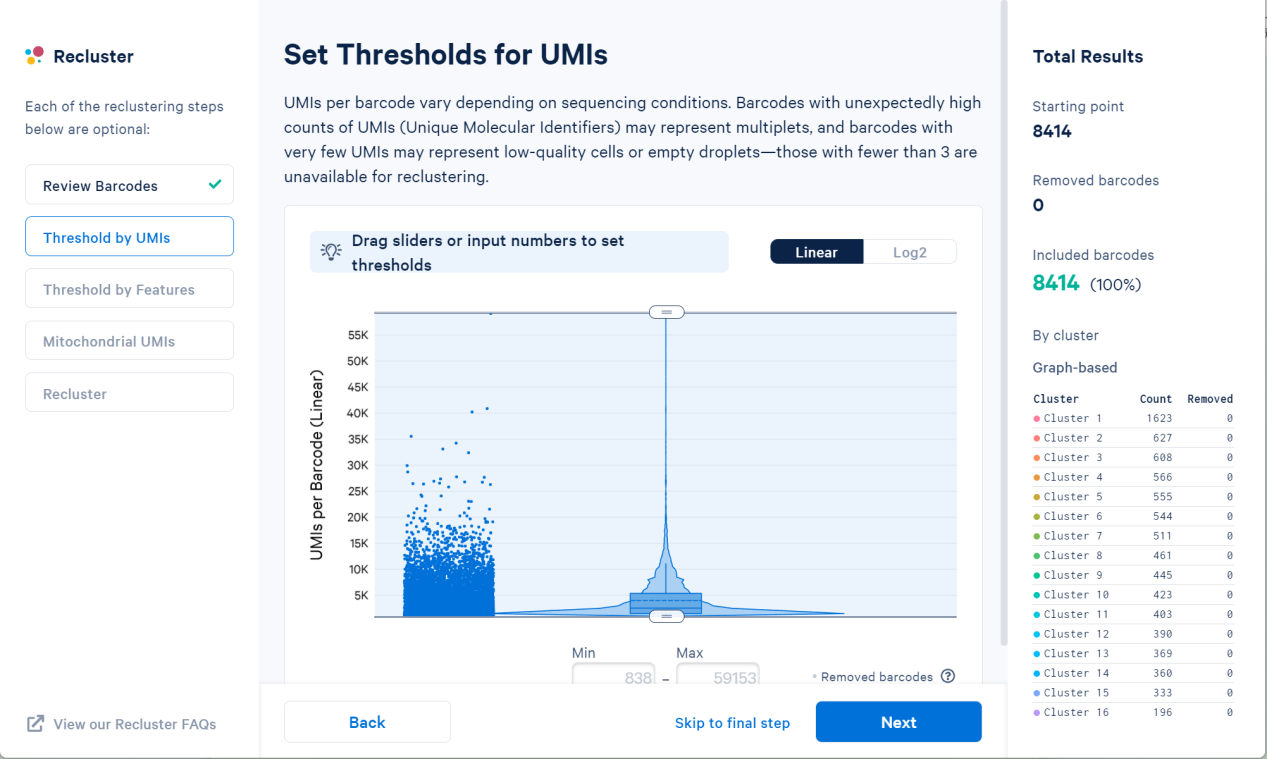
设置UMIs条数的阈值,因为会有空载的油滴(没有捕获单细胞)UMIs条数很少,我们可以选择UMIs阈值筛选掉这部分数据。
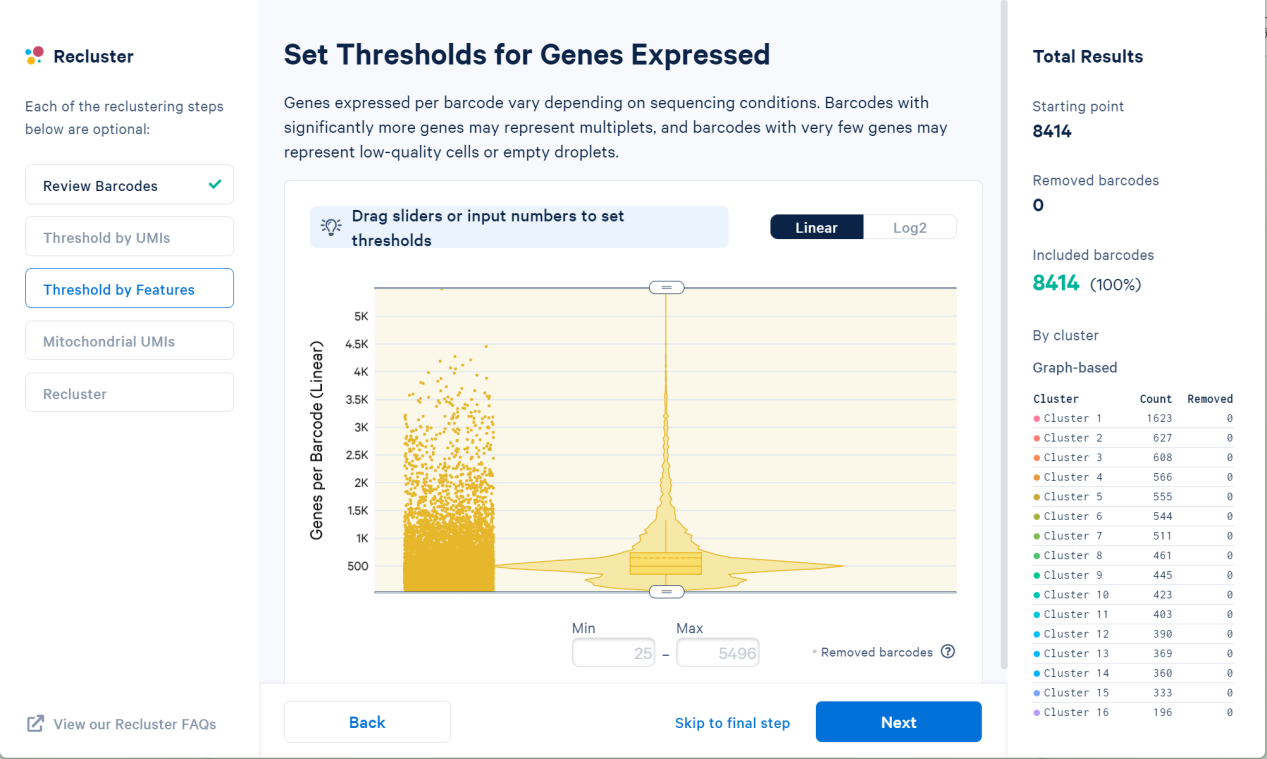
为表达的基因设置阈值。因为每个条码表达的基因因测序条件而异。具有明显更多基因的条形码可能代表多胞胎,而具有很少基因的条形码可能代表低质量细胞或空液滴。
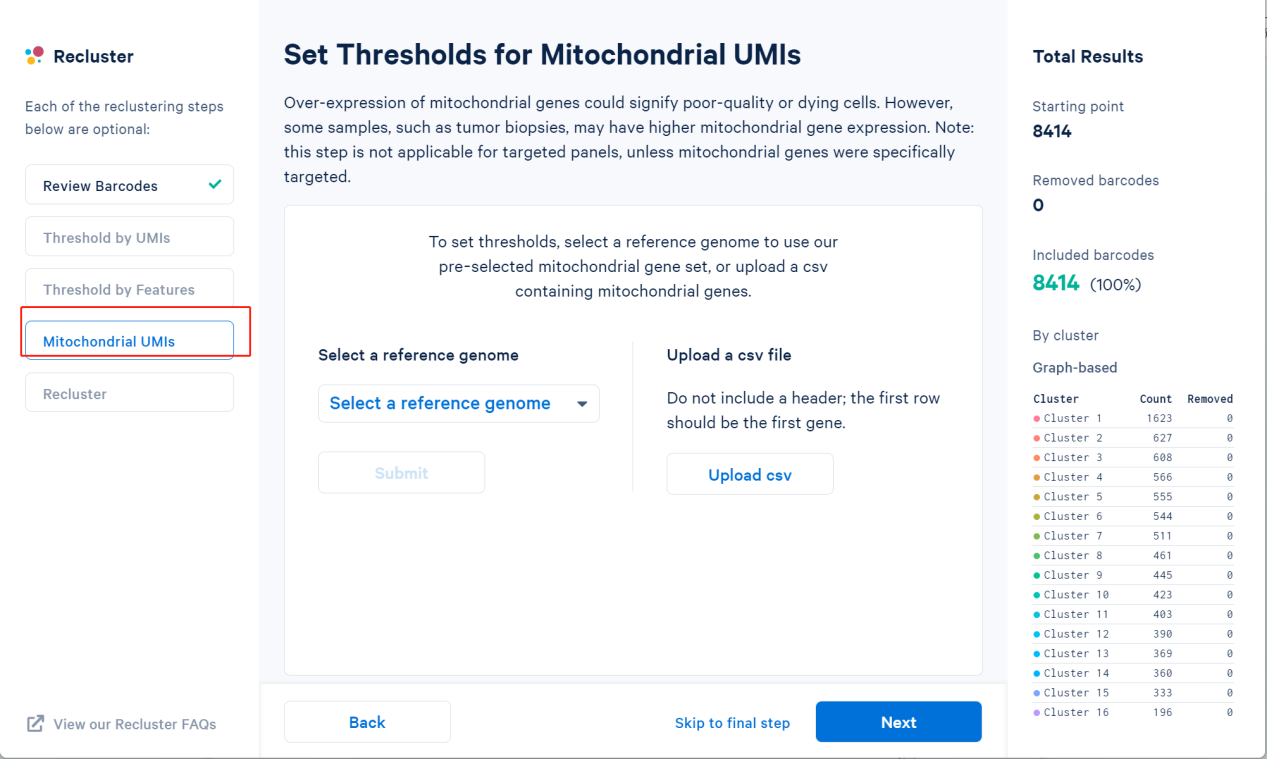
设置线粒体UMI阈值。线粒体基因的过度表达可能意味着细胞质量差或死亡。然而。一些样本,如肿瘤活检,可能有较高的线粒体基因表达。注:此步骤不适用于靶向面板,除非线粒体基因被明确靶向。
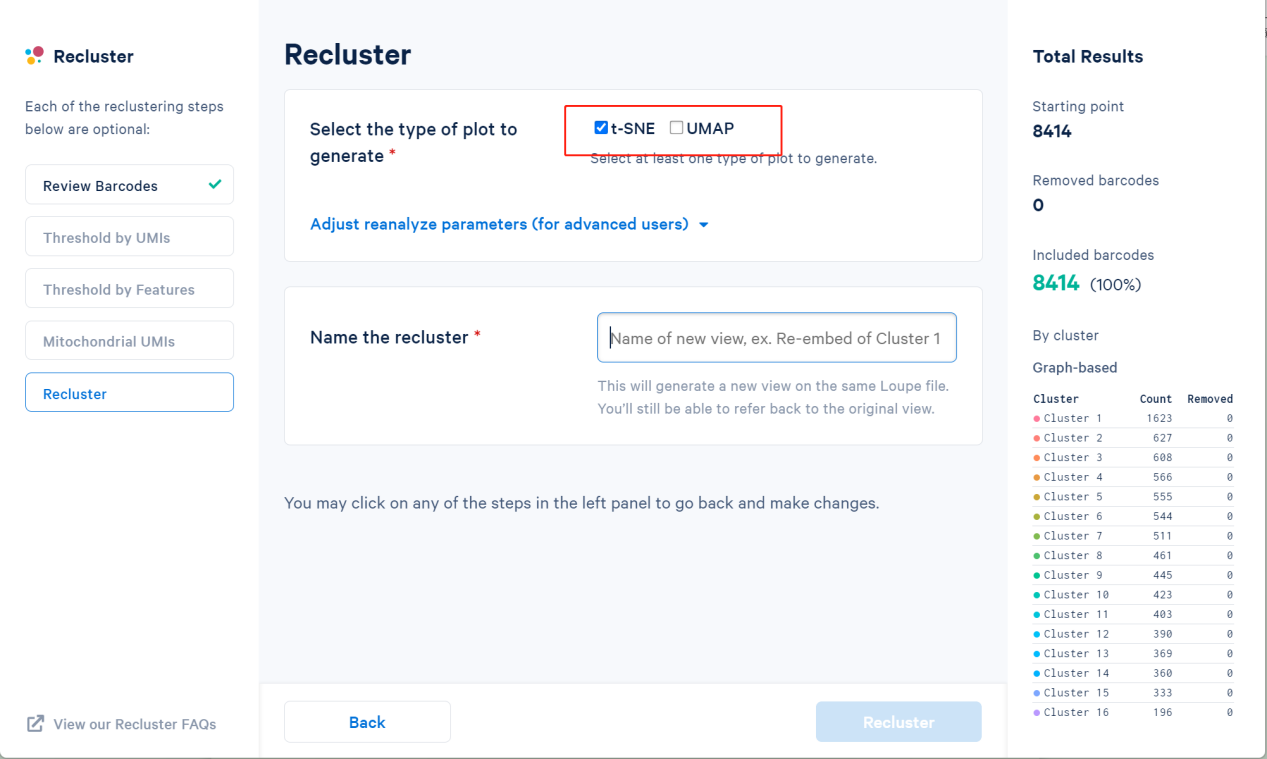
最后就可以聚类了,可以选择生成的数据图形,是t-SNE还是UMAP。选订好参数后,就可以重新聚类了。

这是重新聚类的UMAP图形。
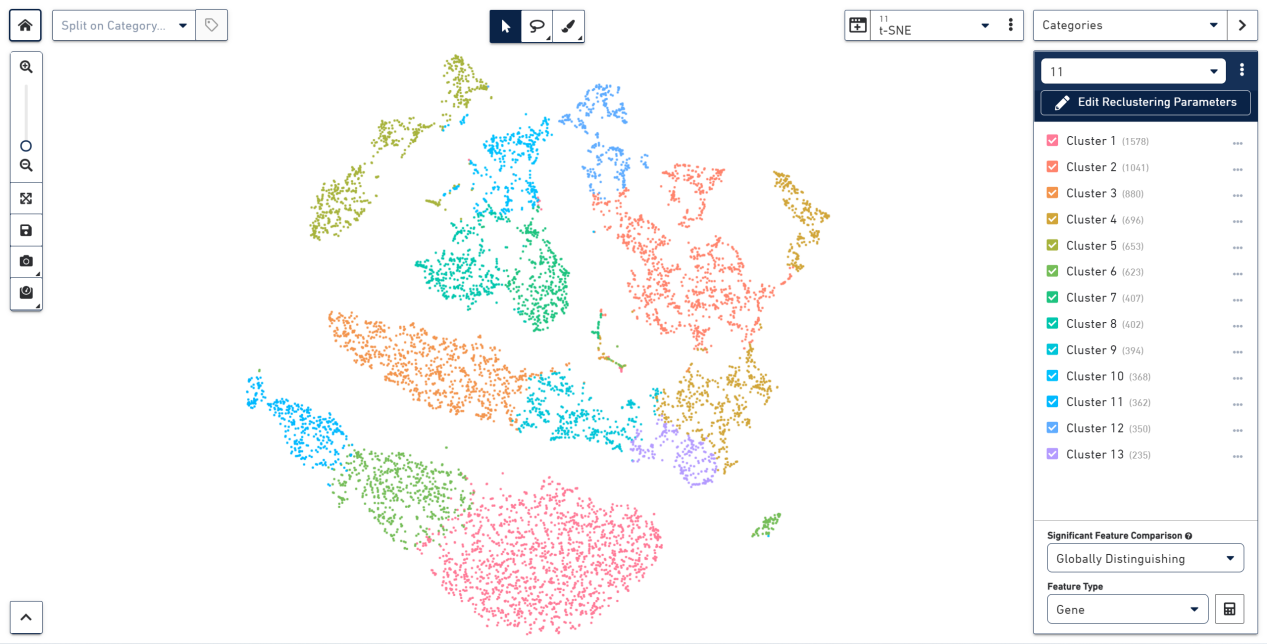 重新聚类的t-SNE图形。
重新聚类的t-SNE图形。
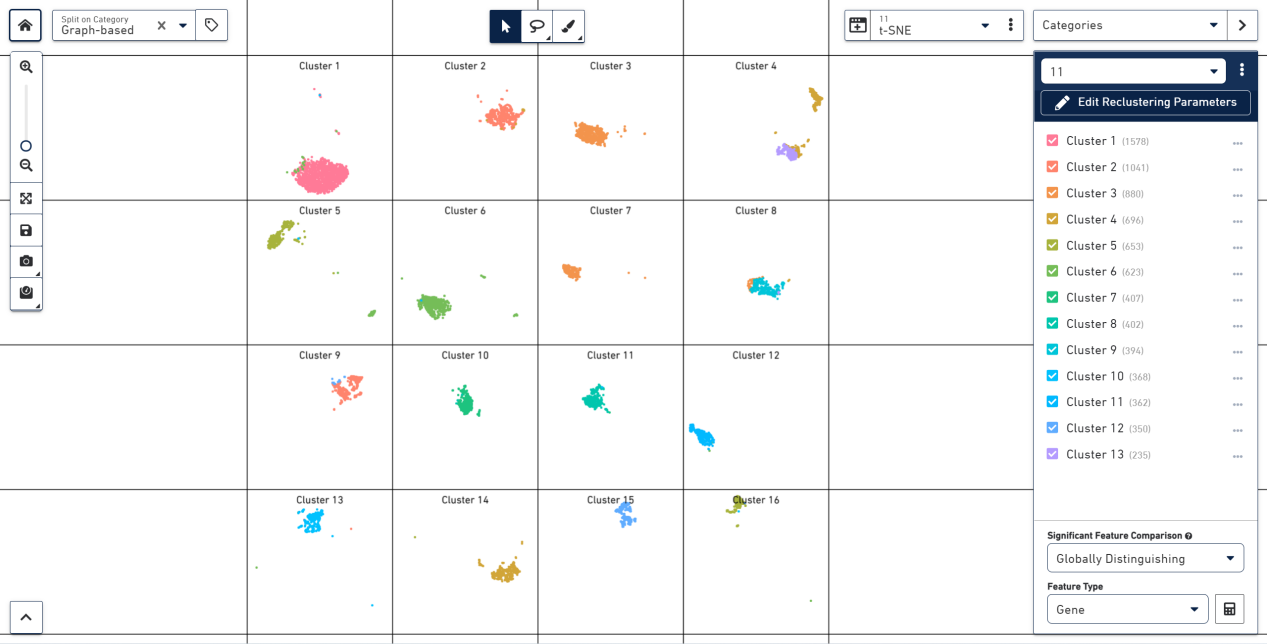
左上角可以将每类细胞单独放在一个格子中。
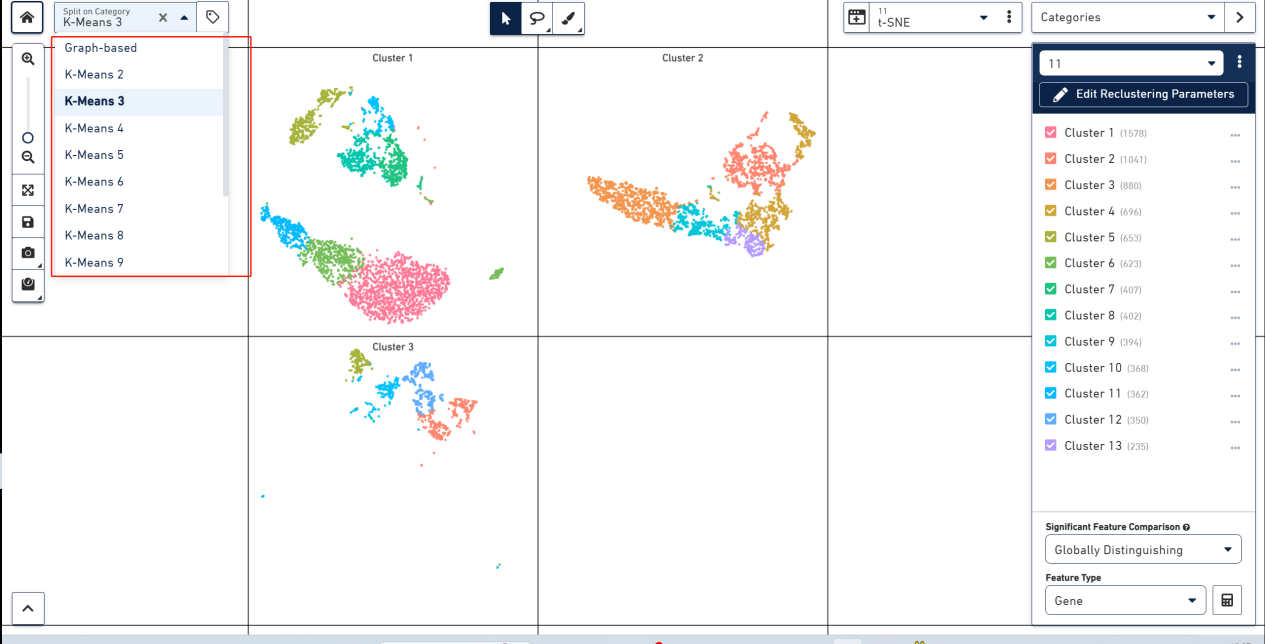
也可以选择分类的数量。
将分析结果传到windows系统下
将上期用Cell Ranger处理得到的cloupe文件传到桌面
sz cloupe.cloupe

加载我们的数据。

调节点的大小。

选择基因在细胞亚群中的分布,可以看出在右下角的细胞中是高表达,而在其他类型细胞中是低表达。

这个基因的小提琴图也说明了这个结果。
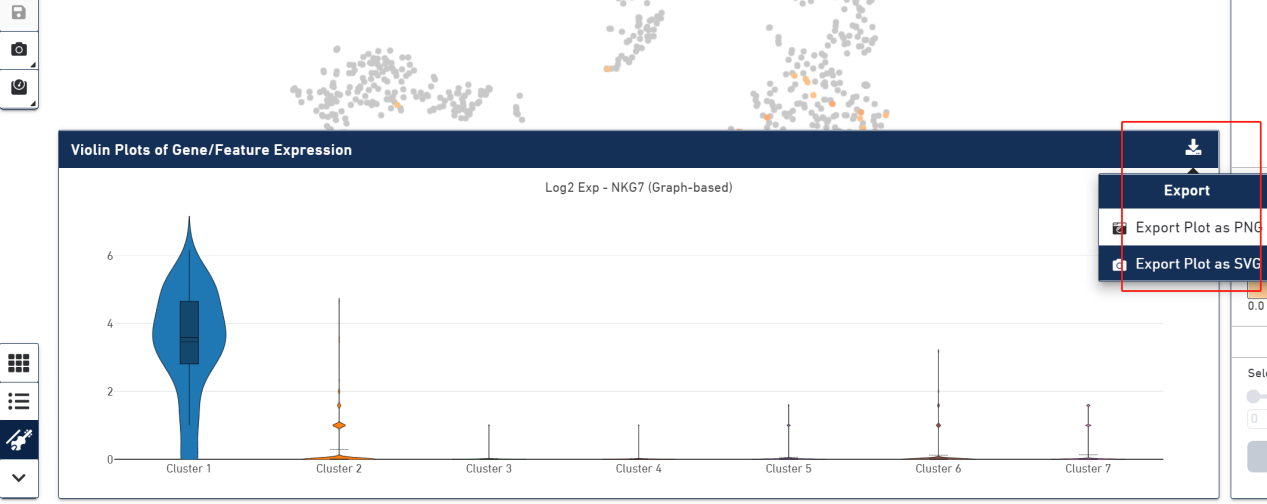
右上角支持对小提琴图进行导出,用于文章的插图。
好了你是否学会了呢?小果今天的分享就到这里了。下一期我们会用R语言的Seurat包对Cell Ranger的处理结果进行下游的分析。我们下期见。
10×genomics单细胞测序分析(三)Loupe Browser数据分析探索
上期小果带大家介绍了Cell Ranger处理分析后输出的文件,并解读了网页版的结果报告。虽然从网页版的报告中我们能够的到诸多的信息,例如:单细胞质控,测序,比对等信息,但是我们不能很好的进行样本的数据探索。这一期小果带大家使用10×genomics平台官方出的Loupe Browse来进行数据探索。
Loupe Browser下载

软件我们可以通过10×的官网下载到。下载安装好即可。

中间有自带的案例数据,有ATAC数据以及空间转录组数据。左边有详细的新手教程。并且可以下载其他的示例数据集。下方有反馈的途径。
Loupe browse教程
Loupe browse较网页板的分析报告,功能更多,更易于数据可视化与多维的数据探索。

选择加载教程数据集。

这是主界面。图中的每个点代表一个细胞,对其进行分类。示例的数据分了16组。
左边有探索数据的工具 。
上方有套索工具,可以对细胞群进行选择,进行进一步的操作。

右上角可以切换图形。

下方是基因的面板。

点击左侧网格图标,可以一键将基因显示成热图。

我们选择LCK基因,然后点击左侧小提琴图标。可以绘画出小提琴图,这个基因在各个细胞组中的基因表达情况,是如何分布的。

右侧框为切片器,我们可以选择或筛选感兴趣的基因。

我们可以通过选择Recluster对细胞重新进行聚类。

选择不同的算法。下方可以根据提供的barcodes列表,选择或者去除细胞。

设置UMIs条数的阈值,因为会有空载的油滴(没有捕获单细胞)UMIs条数很少,我们可以选择UMIs阈值筛选掉这部分数据。

为表达的基因设置阈值。因为每个条码表达的基因因测序条件而异。具有明显更多基因的条形码可能代表多胞胎,而具有很少基因的条形码可能代表低质量细胞或空液滴。

设置线粒体UMI阈值。线粒体基因的过度表达可能意味着细胞质量差或死亡。然而。一些样本,如肿瘤活检,可能有较高的线粒体基因表达。注:此步骤不适用于靶向面板,除非线粒体基因被明确靶向。

最后就可以聚类了,可以选择生成的数据图形,是t-SNE还是UMAP。选订好参数后,就可以重新聚类了。

这是重新聚类的UMAP图形。
 重新聚类的t-SNE图形。
重新聚类的t-SNE图形。

左上角可以将每类细胞单独放在一个格子中。

也可以选择分类的数量。
将分析结果传到windows系统下
将上期用Cell Ranger处理得到的cloupe文件传到桌面
sz cloupe.cloupe


加载我们的数据。

调节点的大小。

选择基因在细胞亚群中的分布,可以看出在右下角的细胞中是高表达,而在其他类型细胞中是低表达。

这个基因的小提琴图也说明了这个结果。

右上角支持对小提琴图进行导出,用于文章的插图。
好了你是否学会了呢?小果今天的分享就到这里了。下一期我们会用R语言的Seurat包对Cell Ranger的处理结果进行下游的分析。我们下期见。