GenomicRanges是R语言中一个强大而灵活的生物信息学包,主要用于处理基因组区间数据。在生物信息学领域,基因组区间数据是指具有位置信息的生物学实体,例如基因、染色体区域、变异位点等。这些区间数据在基因组学、转录组学和表观基因组学等研究中起着重要作用。GenomicRanges包提供了一系列函数和数据结构,用于存储、操作和可视化基因组区间数据,使生物信息学家和基础科研人员能够更加高效地进行基因组数据的分析和解释。
GenomicRanges包提供了广泛的函数和方法,用于对`GRanges`对象进行操作和分析。例如,可以使用`range()`函数来计算区间的长度,使用`overlaps()`函数来查找重叠的区间,使用`subset()`函数来选择特定染色体或位置的区间等等。这些函数使得用户能够方便地进行数据筛选、合并和转换,从而更好地理解基因组数据的特征和变化。
要使用GenomicRanges包,可以在R中使用以下命令进行安装和加载:
> install.packages(“GenomicRanges”) #安装语言包
> library(GenomicRanges) #加载语言包
一个重要的特性是,GenomicRanges包还支持对基因组区间进行集合运算。这意味着用户可以对多个`GRanges`对象进行并集、交集、差集等运算,从而进行更复杂的区间操作和分析。这对于整合多个数据源、比较实验条件或样本之间的差异非常有用,有助于发现特定生物学事件的共同模式或差异。
示例:
假设我们有两个基因组区间数据集,分别代表两个不同实验条件下的基因结合位点。我们将它们表示为两个GRanges对象:set1和set2。现在我们想了解这两个数据集之间的重叠和差异,以及在两个数据集中都存在的共同基因结合位点。
#加载语言包
> library(GenomicRanges)
# 创建示例数据集
> set1 <- GRanges(
+ seqnames = c(“chr1”, “chr1”, “chr2”),
+ ranges = IRanges(start = c(100, 200, 1500), end = c(300, 400, 1800))
+ )
> set2 <- GRanges(
+ seqnames = c(“chr1”, “chr2”),
+ ranges = IRanges(start = c(250, 1600), end = c(350, 1700))
+ )
# 进行集合运算
> union_set <- union(set1, set2) # 并集
> intersect_set <- intersect(set1, set2) # 交集
> setdiff_set1 <- setdiff(set1, set2) # 差集:在set1中而不在set2中的区间
> setdiff_set2 <- setdiff(set2, set1) # 差集:在set2中而不在set1中的区间
# 查看结果
> print(“并集:”)
[1] “并集:”
> print(union_set)
GRanges object with 2 ranges and 0 metadata columns:
seqnames ranges strand
<Rle> <IRanges> <Rle>
[1] chr1 100-400 *
[2] chr2 1500-1800 *
——-
seqinfo: 2 sequences from an unspecified genome; no seqlengths
> print(“交集:”)
[1] “交集:”
> print(intersect_set)
GRanges object with 2 ranges and 0 metadata columns:
seqnames ranges strand
<Rle> <IRanges> <Rle>
[1] chr1 250-350 *
[2] chr2 1600-1700 *
——-
seqinfo: 2 sequences from an unspecified genome; no seqlengths
> print(“在set1中而不在set2中的区间:”)
[1] “在set1中而不在set2中的区间:”
> print(setdiff_set1)
GRanges object with 4 ranges and 0 metadata columns:
seqnames ranges strand
<Rle> <IRanges> <Rle>
[1] chr1 100-249 *
[2] chr1 351-400 *
[3] chr2 1500-1599 *
[4] chr2 1701-1800 *
——-
seqinfo: 2 sequences from an unspecified genome; no seqlengths
> print(“在set2中而不在set1中的区间:”)
[1] “在set2中而不在set1中的区间:”
> print(setdiff_set2)
GRanges object with 0 ranges and 0 metadata columns:
seqnames ranges strand
<Rle> <IRanges> <Rle>
——-
seqinfo: 2 sequences from an unspecified genome; no seqlengths
以上示例中,我们首先创建了两个示例的GRanges对象 set1和 set2,分别代表两个不同实验条件下的基因结合位点。然后,我们使用union()函数计算了这两个数据集的并集,使用intersect()函数计算了交集,以及使用setdiff()函数分别计算了在set1中而不在set2中以及在set2中而不在set1中的基因结合位点。
这样,我们就能够分析和理解这两个数据集之间的重叠情况和差异,从而更好地了解基因结合位点在不同实验条件下的调控情况。这对于理解基因调控、研究转录因子结合等生物学问题非常有帮助。
除了基本的数据操作外,GenomicRanges包还具有强大的可视化功能。用户可以使用`plot()`函数直接绘制`GRanges`对象,将基因组区间在染色体上进行可视化,或者通过其他图表展示基因组数据的特征。这使得结果的解释更直观,有助于研究人员快速观察数据中的趋势和异常情况。
示例:
假设我们有一个GRanges对象,代表一组基因的起始和终止位置信息,以及它们所在的染色体名称。我们将使用plot()函数将这些基因在染色体上进行可视化。
# 导入GenomicRanges包
> library(GenomicRanges)
# 导入ggbio包
> install.packages(“BiocManager”)
> BiocManager::install(“ggbio”)
> library(ggbio)
# 创建示例数据集
> genes <- GRanges(
+ seqnames = c(“chr1”, “chr1”, “chr2”, “chr2”),
+ ranges = IRanges(start = c(100, 200, 1500, 1700), end = c(300, 400, 1800, 2000))
+ )
> # 将GRanges对象转换为data.frame
> genes_df <- as.data.frame(genes)
# 绘制基因组区间数据
> p <- ggplot() +
+ geom_rect(data = genes_df, aes(xmin = start, xmax = end, ymin = 0, ymax = 1), fill = “blue”) +
+ facet_wrap(~ seqnames, scales = “free_x”, ncol = 1) +
+ labs(x = “位置”, y = “染色体”) +
+ theme_bw()
> print(p)
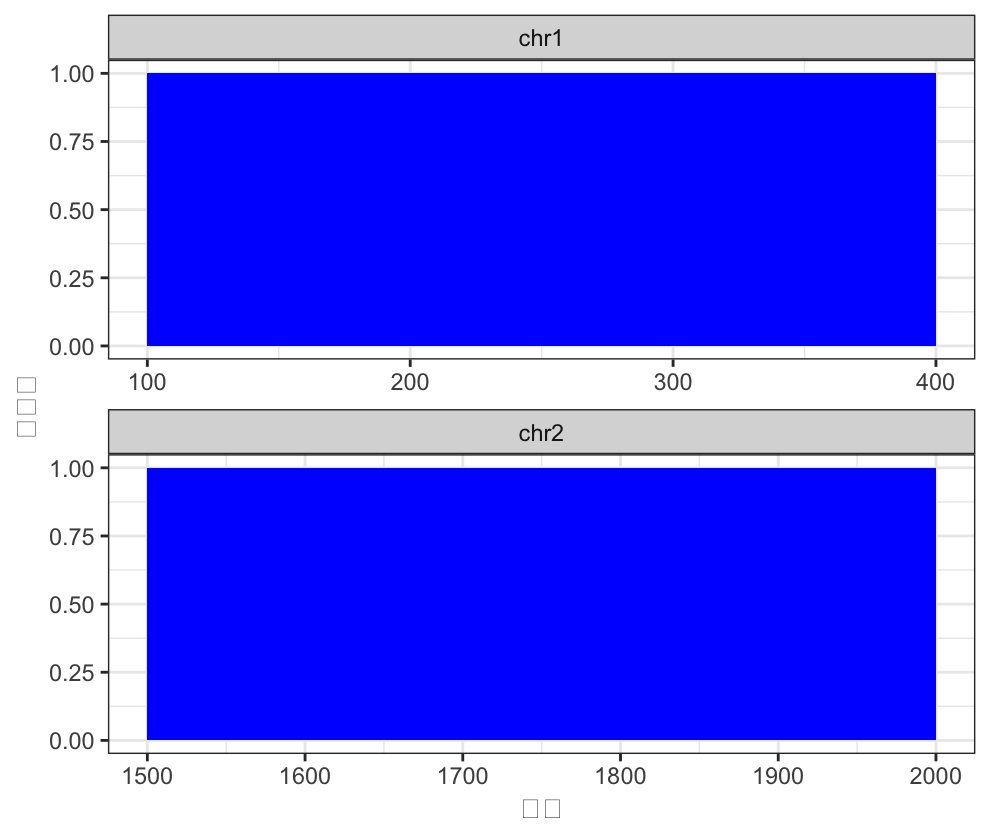
绘图结果会显示两条染色体,基因用蓝色矩形表示。每个矩形的横轴表示基因的起始和终止位置,纵轴表示染色体。由于我们使用了facet_wrap()函数,将图形按照染色体进行了分组,所以每个染色体的基因组区间都在一个单独的子图中展示。
在实际应用中,GenomicRanges包在许多生物信息学任务中发挥着重要作用。例如,它常用于处理转录组测序(RNA-seq)数据,通过将基因组区间与基因表达量关联,进行差异表达分析和功能注释。此外,基因组区间数据也经常用于寻找蛋白质结合位点、检测染色体变异、研究表观遗传学修饰等研究中。
以上就是对R语言包GenomicRanges的简单介绍啦,它为生物信息学领域的研究人员提供了高效、灵活的基因组区间数据处理工具。其丰富的函数和方法,以及易于使用的数据结构,使得基因组学数据的分析和解释变得更加简单和直观。不仅如此,GenomicRanges包还积极维护更新,与R生态系统中的其他包相互兼容,保证了其在生物信息学研究中的长期稳定性和广泛应用。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html
References: