在最近的这段时间里相信做科研的各位小伙伴都被GWAS和孟德尔随机化刷过屏,小果也有在做教学的推文哦~有兴趣的小伙伴可以关注我们的公众号生信果进行了解学习呀!
背景知识
现在,让小果简单介绍一下为何GWAS和孟德尔随机化具有如此紧密的联系吧!
孟德尔随机化(Mendelian Randomization)近来备受欢迎,其中部分原因在于它能够最大程度地减少其他观察性流行病学研究设计中的两个主要问题:混杂因素和逆向因果。通过将遗传变异视为一种“自然的随机化实验”,Mendelian Randomization能够提供关于可调控风险因素与疾病之间可能的因果关系的证据。此外,相较于随机临床试验,Mendelian Randomization所需的时间和成本较少,同时能够分析现有研究并解决随机临床试验无法解决的问题。
与此同时,全基因组关联研究(GWAS)也变得越来越受欢迎,因为它可以发现在Mendelian Randomization研究中调查的中间表型中对变异有贡献的多个遗传变异,从而增强了Mendelian Randomization研究的效力。
因此,GWAS和孟德尔随机化两者的结合成为了当前研究领域的新热点之一。
而小果今天想分享给大家的R包CMplot是一款专门绘制GWAS中非常重要的可视化工具–曼哈顿图的R包,与小果之前介绍过的qqman不同,CMplot包只有一个函数,但通过参数的设置,小伙伴们可以通过这一个函数绘制出SNP密度图,普通/环状的曼哈顿图,以及QQ图。
以下是曼哈顿图在GWAS中的重要性:
提供所有关联的概览:曼哈顿图简洁地总结了整个基因组中基因型(SNP、indels等)与表型之间的关联结果。这使得所有关联可以一目了然地查看。 确定显著区域:y轴显示关联的强度,使用-log10 p值。突破显著性阈值的峰值容易识别为潜在的表型风险位点。 显示空间模式:可以观察沿x轴排列的染色体上显著SNP的空间模式,如聚类。这有助于识别候选基因。 方便比较:曼哈顿图允许在不同的GWAS结果之间进行视觉比较,如病例与对照组之间。关联模式的相似性和差异性一目了然。 直观解释:曼哈顿图通过对强关联和弱关联的可视化分离,使其能够在没有统计专业知识的情况下直观解释。 有助于传播:作为简洁的汇总图,曼哈顿图对于向科学界和一般观众传达GWAS的关键发现非常有用。
结果简单的介绍过后,就由小果带领大家一起来入门CMplot这款R包吧!
# Step 0 下载R包
## CMplot可以通过最基本的下载方式进行下载哦
# install.packages(“CMplot”)
library(CMplot)
# Step 1 加载数据
## 在CMplot中,R包的作者为我们准备了两组数据,分别是cattle50k和pig60k,小果今天使用cattle50k为大家进行演示
data(“cattle50K”)
## 我们可以观察一下数据
head(cattle50K)
## SNP chr pos Somatic cell score Milk yield Fat percentage
## 1 SNP1 1 59082 0.000244361 0.000484255 0.001379210
## 2 SNP2 1 118164 0.000532272 0.000039800 0.000598951
## 3 SNP3 1 177246 0.001633058 0.000311645 0.000279427
## 4 SNP4 1 236328 0.001412865 0.000909370 0.001040161
## 5 SNP5 1 295410 0.000090700 0.002202973 0.000351394
## 6 SNP6 1 354493 0.000110681 0.000342628 0.000105792
## 数据最少有4列,分别是SNP,染色体,染色体位置和性状,p.s. 在这组数据中为我们展示了三个性状
加载数据过后就要开始学习今天CMplot的绘制学习啦! 小果为大家做了一个所有参数的汇总,参数有点多,各位小伙伴大概的浏览一下就可以哦~
@ 基础参数
Pmap:一个数据框,至少包含四列。第一列是SNP的名称,第二列是SNP的染色体,第三列是SNP的位置,剩余的列是每个特征的P值(每个特征一个列)。
band:染色体之间的距离,默认为1(如果band等于0,则染色体之间将没有间隔)。
H:每个圆的高度,每个圆代表一个特征,默认为1。
plot.type:一个字符或向量,只能使用”d”、”c”、”m”、”q”或”b”。如果plot.type=”d”,将绘制SNP密度图;如果plot.type=”c”,只绘制圆形曼哈顿图;如果plot.type=”m”,只绘制曼哈顿图;如果plot.type=”q”,只绘制Q-Q图;如果plot.type=”b”,将绘制圆形曼哈顿图、曼哈顿图和Q-Q图;如果plot.type=c(“m”,”q”),将绘制曼哈顿图和Q-Q图。
multracks:一个逻辑值,如果multracks=FALSE,则所有曼哈顿图将在分离的文件中绘制,如果为TRUE,则所有曼哈顿图将绘制在一个文件中。
points.alpha:一个数字,用于多特征曼哈顿图中点的透明度,范围从0(完全透明)到255(不透明)。
cex:一个数字或向量,用于点的大小,与<plot>中的”size”参数相同。如果是向量,则第一个数字控制圆形图中点的大小(默认为0.5),第二个数字控制曼哈顿图中点的大小(默认为1),第三个数字控制Q-Q图中点的大小(默认为1)。
r:一个数字,表示圆的半径(内半径),默认为1。
col:一个向量或矩阵。如果”col”是向量,则每个圆使用相同的颜色,表示相同染色体使用相同颜色。颜色不是固定的,可以使用一个、两个、三个或更多颜色。如果”col”的长度比染色体的长度短,则颜色会循环使用。如果”col”是矩阵,则行数是特征的数量,列数是用户想要为不同特征使用的颜色数,每个特征可以用不同数量的颜色绘制,缺失值可以用NA代替。
pch:一个数字,用于绘制点或多特征曼哈顿图的特征的形状。
type:一个字符,可以是”p”(点)、”l”(线)、”h”(垂直线)等,与<plot>中的”type”参数相同。
outward:一个逻辑值,如果outward=TRUE,则所有点将从内部向外绘制圆形曼哈顿图。
LOG10:逻辑值,是否将p-value转换为log10(p-value)。
@ 曼哈顿图参数
trait.legend.ncol:多特征曼哈顿图图例的列数。
trait.legend.cex:设置特征名称图例文本的大小。
trait.legend.pos:控制特征名称图例在图中的位置,有三个选项:”left”、”middle”、”right”可用。
@ QQ图参数
conf.int:逻辑值,是否在QQ图上绘制置信区间。
conf.int.col:字符或向量,QQ图置信区间的颜色。
@ 密度图参数
bin.size:SNP_density绘图的bin大小。
bin.range:一个向量,c(min, max),表示SNP_density绘图的最小/最大值。小于/大于’bin.range’的SNP数将使用相同的颜色。
bin.legend.num:SNP密度图图例的断点数量,默认为10。
@ 坐标轴
ylim:向量或列表,CMplot只会在此间隔内绘制点,ylim可以是向量或列表,如果是列表,不同特征可以分配不同的y轴范围。
cex.axis:一个数字,控制X/Y轴刻度标签的大小和圆形图的染色体标签的大小。
lwd.axis:一个数字,控制X/Y轴线的粗细。
cex.lab:一个数字,控制X/Y轴标签的大小。
ylab:一个字符,y轴的标签。
ylab.pos:ylab和y轴之间的距离。
xticks.pos:x轴刻度标签和x轴之间的距离。
@ 标题参数
main:字符或向量,曼哈顿图和QQ图的标题。
main.cex:标题的大小。
main.font:标题的字体。
@ 画布
box:逻辑值,此函数在当前曼哈顿图周围绘制一个框。
mar:绘图周围的白色间隔的大小,需要提供4个值,表示底部、左侧、上方和右侧的间隔方向。
@ 阈值
threshold:一个数字、向量或列表,表示显著性阈值。例如,Bonferroni校正方法:threshold=0.01/nrow(Pmap),可以在绘图中添加多条显著性线,如果是列表,则可以为每个特征分配不同的阈值,如果threshold=0或NULL,则不会添加阈值线。
threshold.col:一个字符或向量,表示阈值级别线的颜色,也可以控制Q-Q图的对角线的颜色。
threshold.lwd:一个数字或向量,表示阈值级别线的宽度,也可以控制Q-Q图的对角线的粗细。
threshold.lty:一个数字或向量,表示阈值级别线的类型,也可以控制Q-Q图的对角线的类型。
@ 显著点
amplify:逻辑值,CMplot可以放大显著性点。如果amplify=T,则大于最小显著水平的点将被放大,默认值:amplify=TRUE。
signal.cex:一个数字,如果amplify=TRUE,则可以设置显著性点的大小。
signal.pch:一个数字,如果amplify=TRUE,则可以设置显著性点的pch。
signal.col:一个字符,如果amplify=TRUE,则可以设置显著性点的颜色,如果signal.col=NULL,则显著性点的颜色不会改变。
signal.line:一个数字,表示显著性SNP的线的粗细。
highlight:一个向量或列表,表示需要突出显示的SNP的名称。向量用于单个特征,列表用于多个特征。
highlight.cex:一个向量或列表,表示需要突出显示的SNP的点的大小。
highlight.pch:一个向量或列表,表示需要突出显示的SNP的点的pch。
highlight.type:一个向量或列表,表示需要突出显示的SNP的点的类型。
highlight.col:一个向量或列表,表示需要突出显示的SNP的点的颜色。
highlight.text:一个向量或列表,表示需要在突出显示的SNP周围添加的文本。
highlight.text.col:一个向量或列表,表示添加的文本的颜色。
highlight.text.cex:一个值,表示添加的文本的大小。
highlight.text.xadj:对于单个特征的值或向量,对于多个特征的列表,控制文本在突出显示的SNP周围的位置,-1(左),0(中心),1(右)。
highlight.text.yadj:对于单个特征的值或向量,对于多个特征的列表,控制文本在突出显示的SNP周围的位置,-1(向下),0(中心),1(向上)。
highlight.text.font:突出显示的SNP的文本字体。
@ 染色体相关参数
chr.labels:一个向量,表示密度图和圆形曼哈顿图的染色体标签。
chr.border:逻辑值,是否在染色体之间绘制点线。
chr.labels.angle:一个值,调整曼哈顿图x轴刻度标签的角度(-90 < chr.labels.angle < 90)。
chr.pos.max:逻辑值,是否将每个染色体的物理位置包含在染色体的最大长度内。
chr.den.col:一个字符、向量或NULL,表示SNP密度的颜色。如果参数’chr.den.col’的长度大于1,则会在圆形图的底部绘制计算给定大小(’bin.size’)内的SNP数量的SNP密度。如果chr.den.col=NULL,则不会在曼哈顿图底部附加密度条。
cir.band:一个数字,表示圆之间的间距,默认为1。
cir.chr:逻辑值,是否在圆的周边绘制表示染色体的边界,默认为TRUE。
cir.chr.h:一个数字,表示边界的宽度,如果cir.chr=FALSE,则此参数无效。
cir.legend:逻辑值,是否添加每个圆的图例。
cir.legend.cex:一个数字,图例文本的大小。
cir.legend.col:一个字符,图例轴的颜色。
@ 文件输出参数
file.output:逻辑值,用户可以选择是否输出绘图结果。
file.name:字符或向量,输出文件的名称。
file:字符,用户可以选择绘图的不同输出格式,目前支持”jpg”、”pdf”、”tiff”。
dpi:一个数字,.jpg和.tiff文件的图片分辨率,默认为300。
height:输出文件的高度。
width:输出文件的宽度。
verbose:是否打印日志信息。
SNP密度图
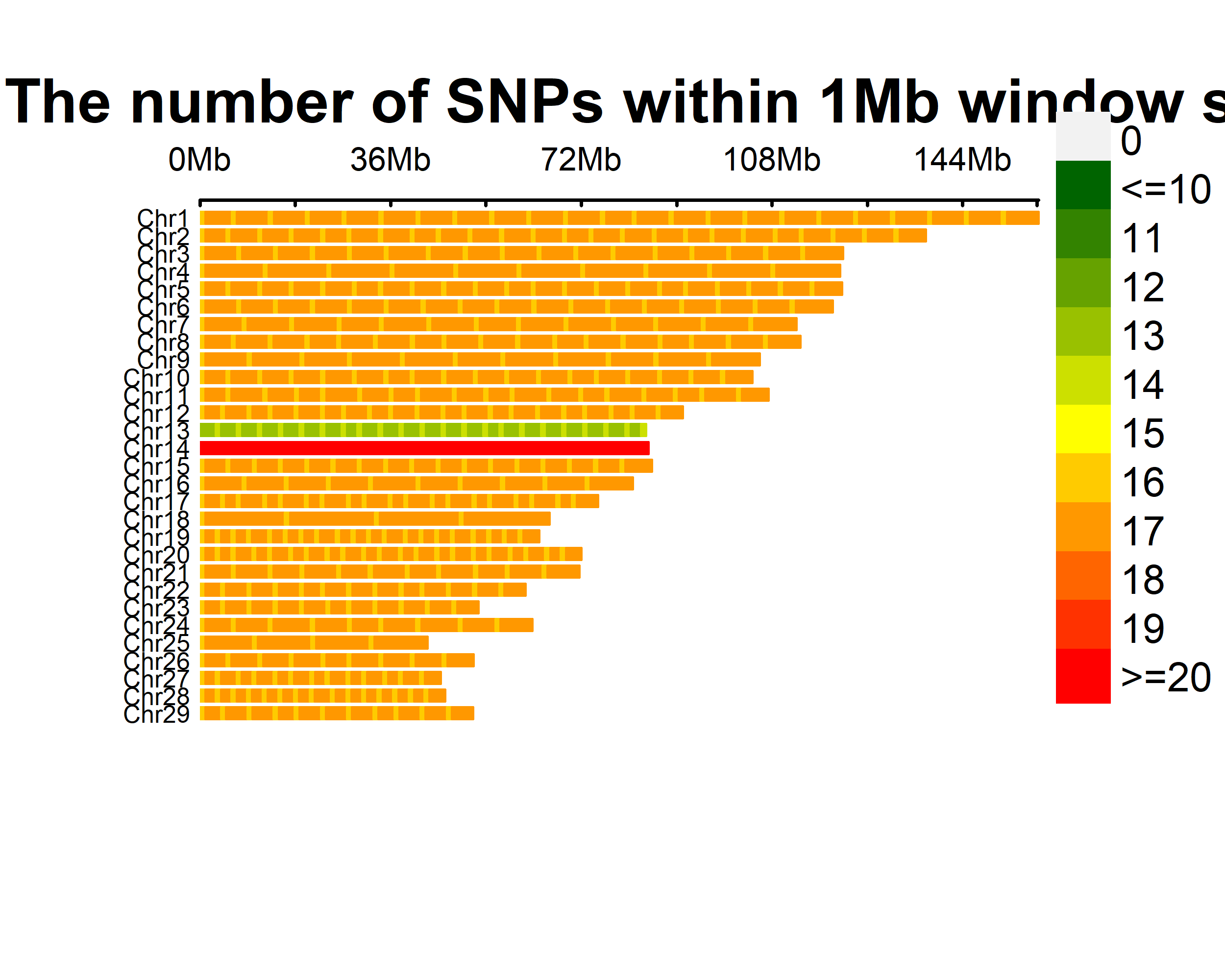
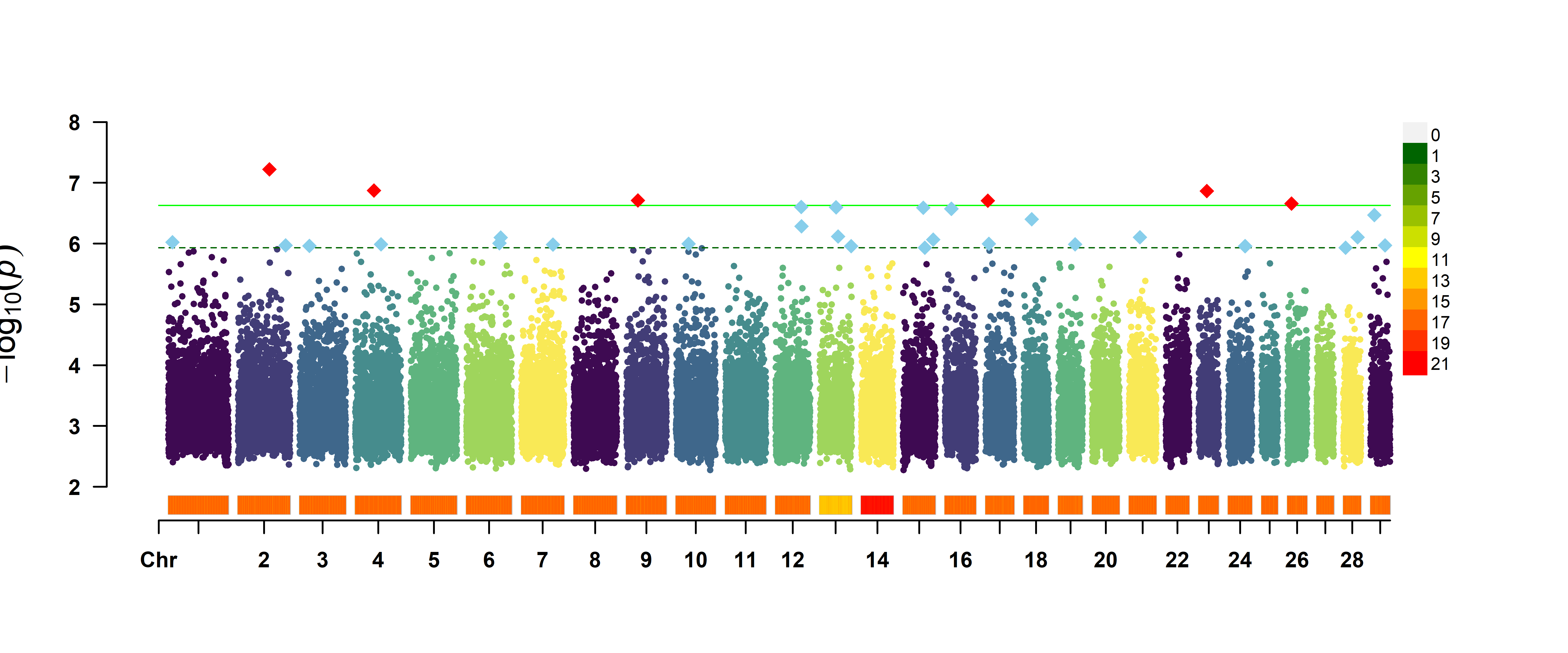
单性状曼哈顿图
CMplot(cattle50K, plot.type = “m”,
col= c(“#3E0A52”, “#423D77″,”#3F678B”,
“#468C8D”, “#5FB47F”, “#9FD55C”,”#F9E956″), cex = 0.5,
threshold = c(0.01,0.05)/nrow(cattle50K),
threshold.col = c(‘green’,’darkgreen’),threshold.lty = c(1,2),threshold.lwd = c(1,1),
amplify = T, signal.cex = c(1.5,1.5), signal.pch = c(18,18),signal.col = c(“red”,”skyblue”),
chr.den.col = c(“darkgreen”,”yellow”,”red”), # 在曼哈顿图中添加SNP密度图
file.output = F)
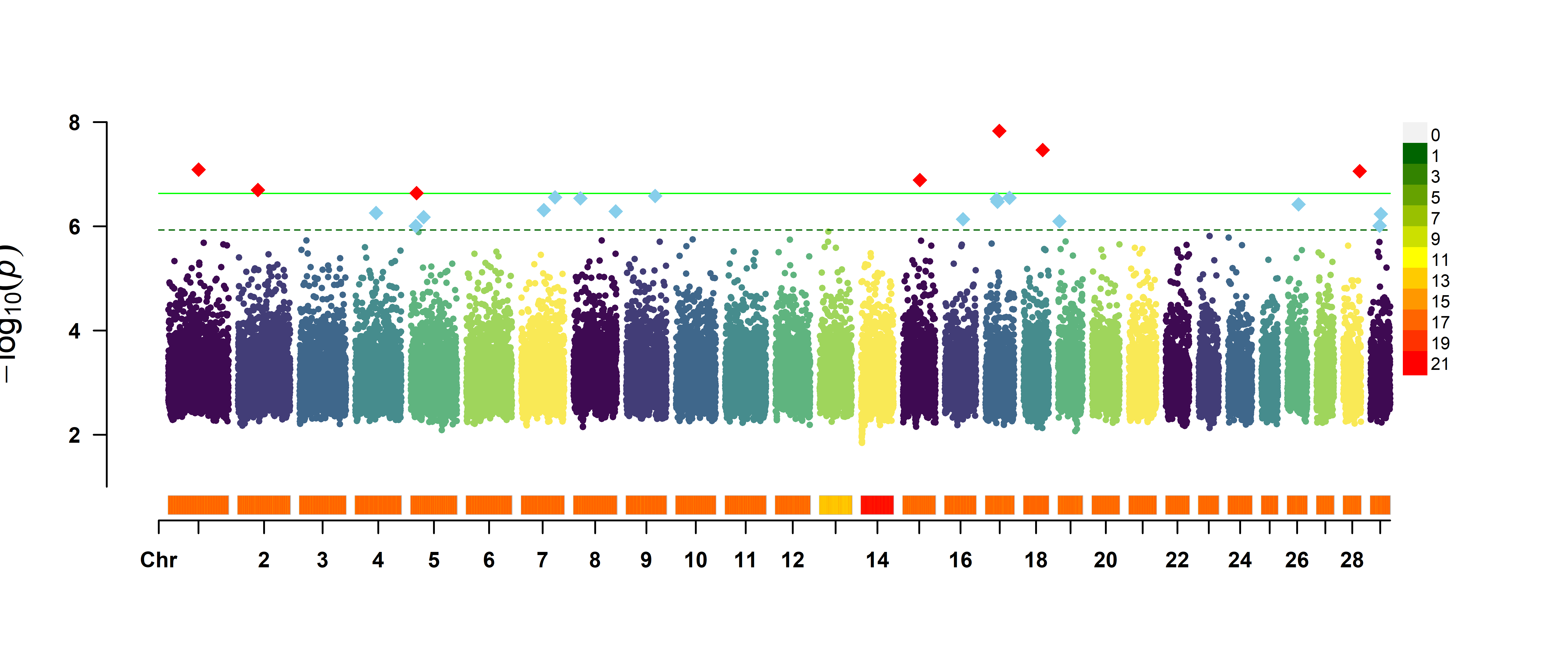
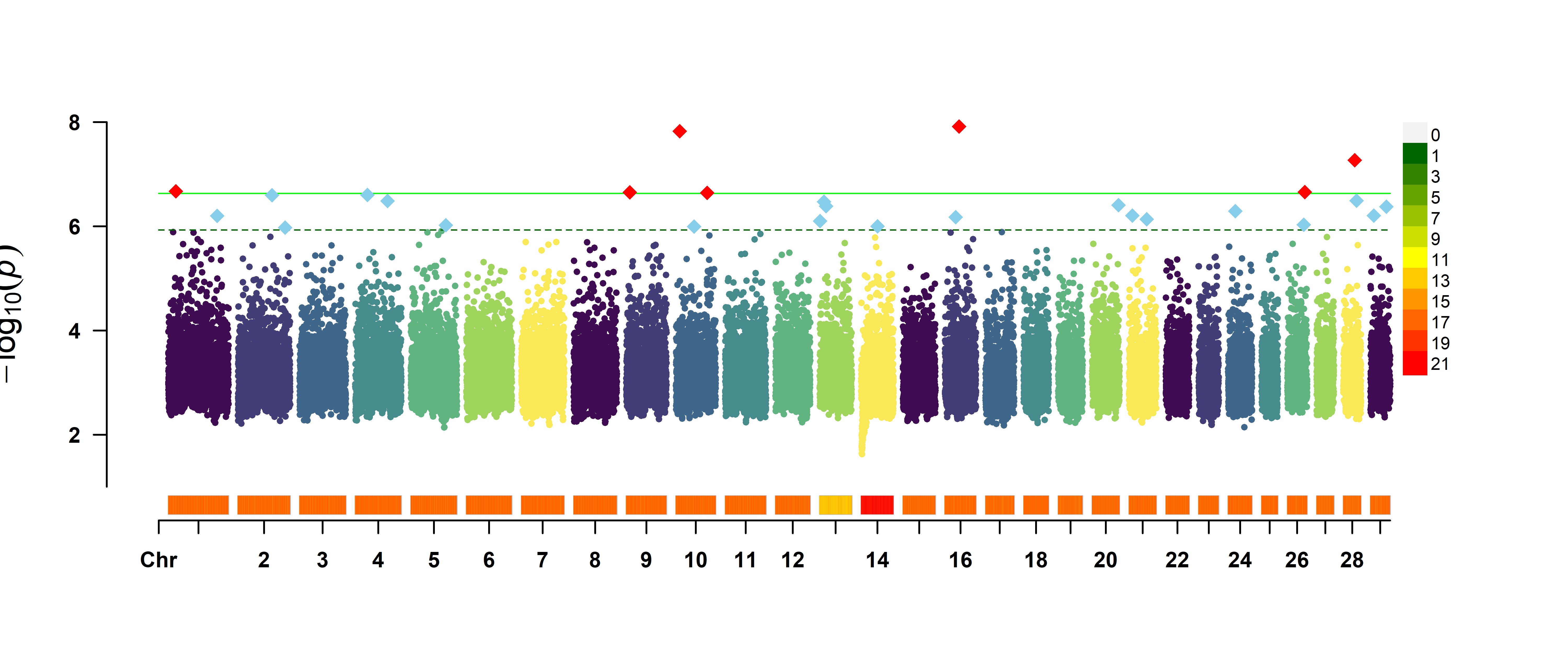
多性状曼哈顿图
CMplot(cattle50K, plot.type = “m”, cex = 0.5, r = 0.5, threshold = c(0.01,0.05)/nrow(cattle50K),
threshold.col = c(‘green’,’darkgreen’),threshold.lty = c(1,2),threshold.lwd = c(1,1),
amplify = T, multracks = T, signal.cex = c(1,1), signal.pch = c(18,18),signal.col = c(“red”,”skyblue”),
file.output = F)
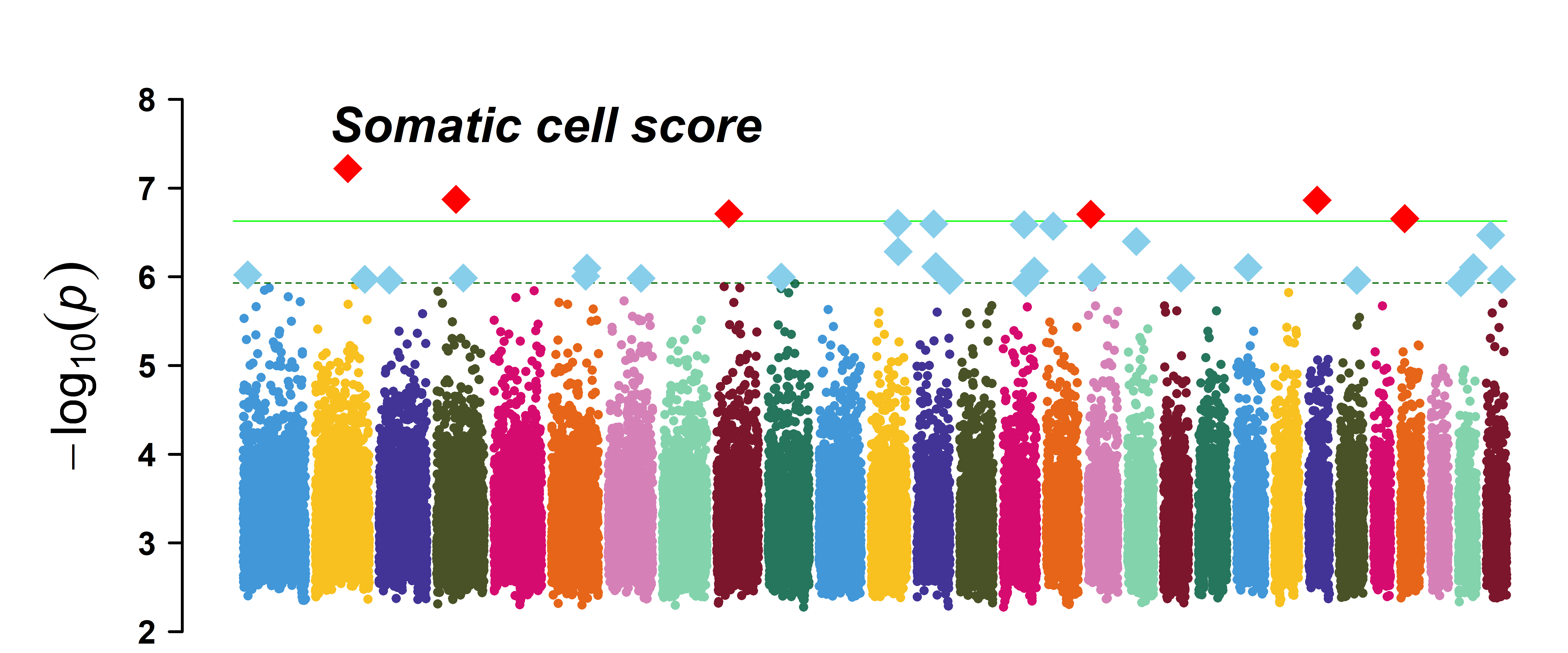

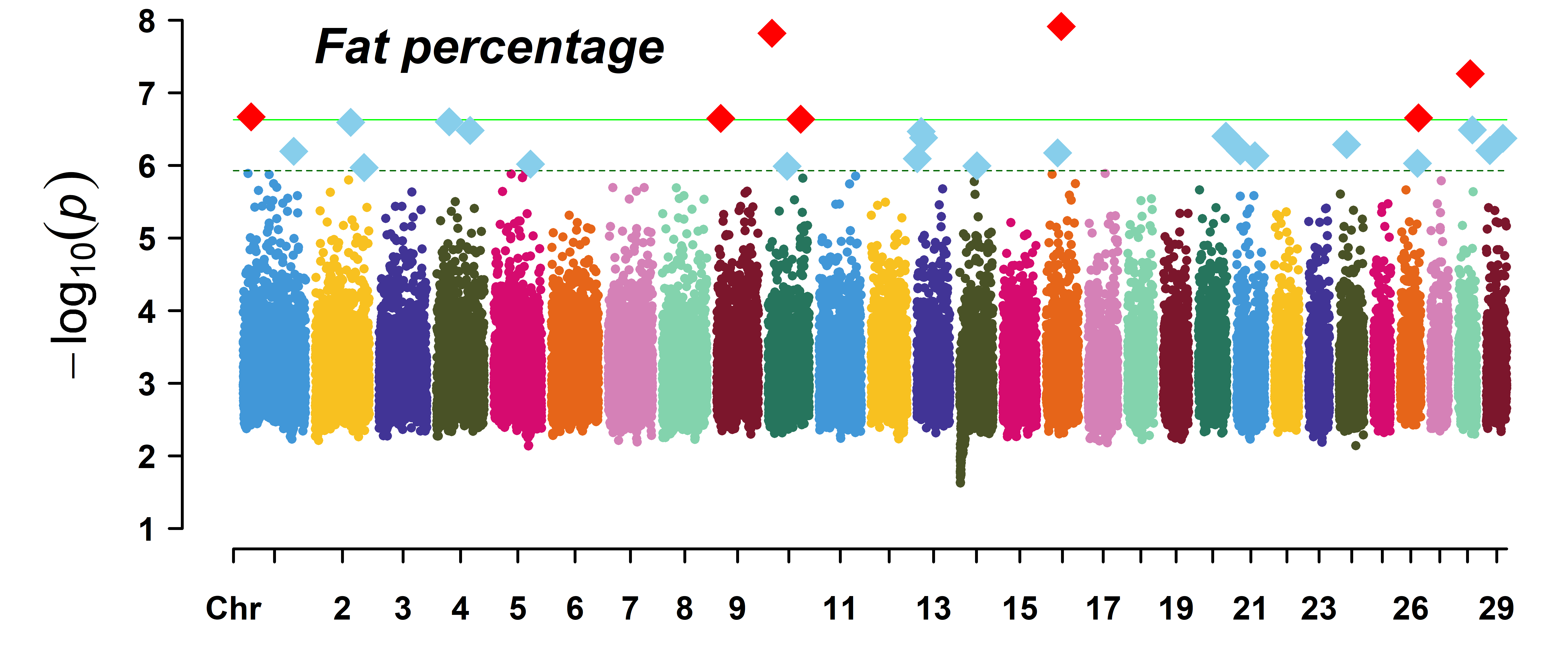
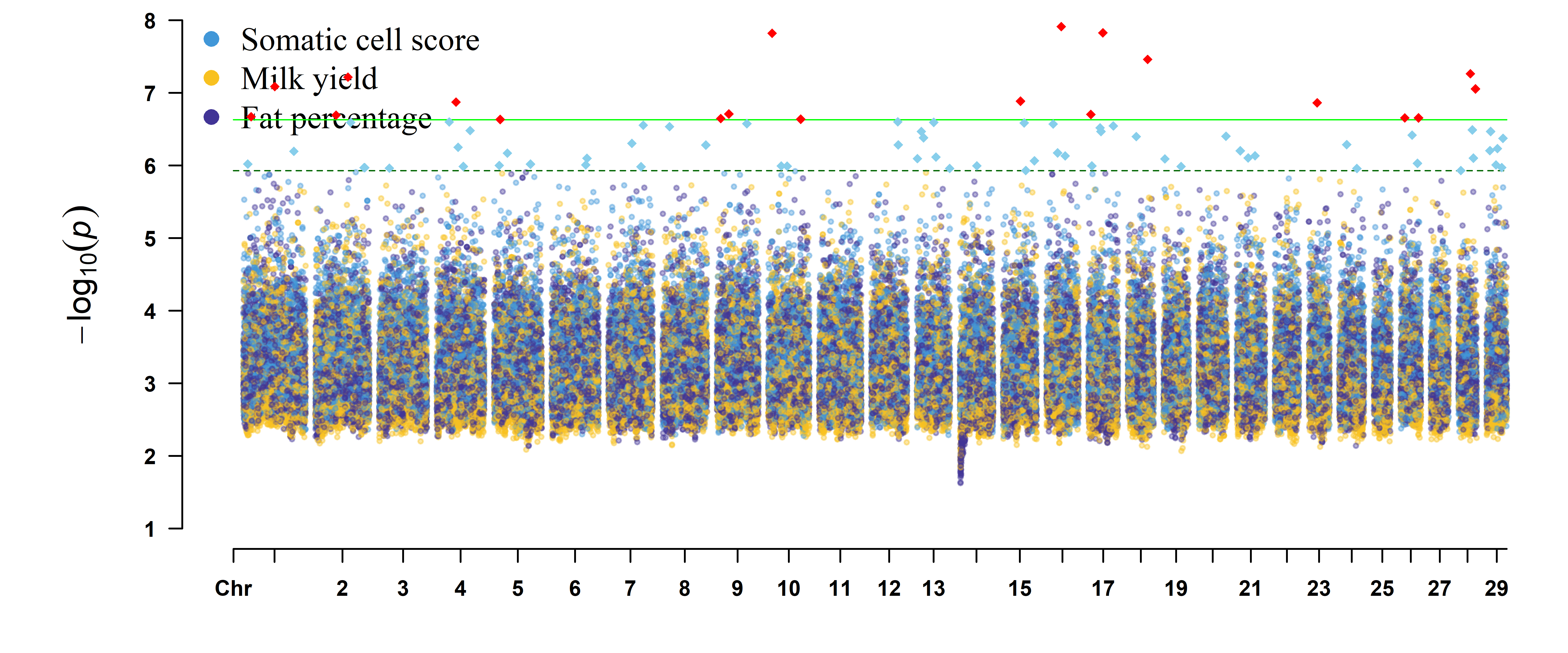
从这个图中我们可以看出多性状的漫画图比较杂乱,普通的曼哈顿图不适合展示多性状的GWAS结果。但我们可以使用环形曼哈顿图!
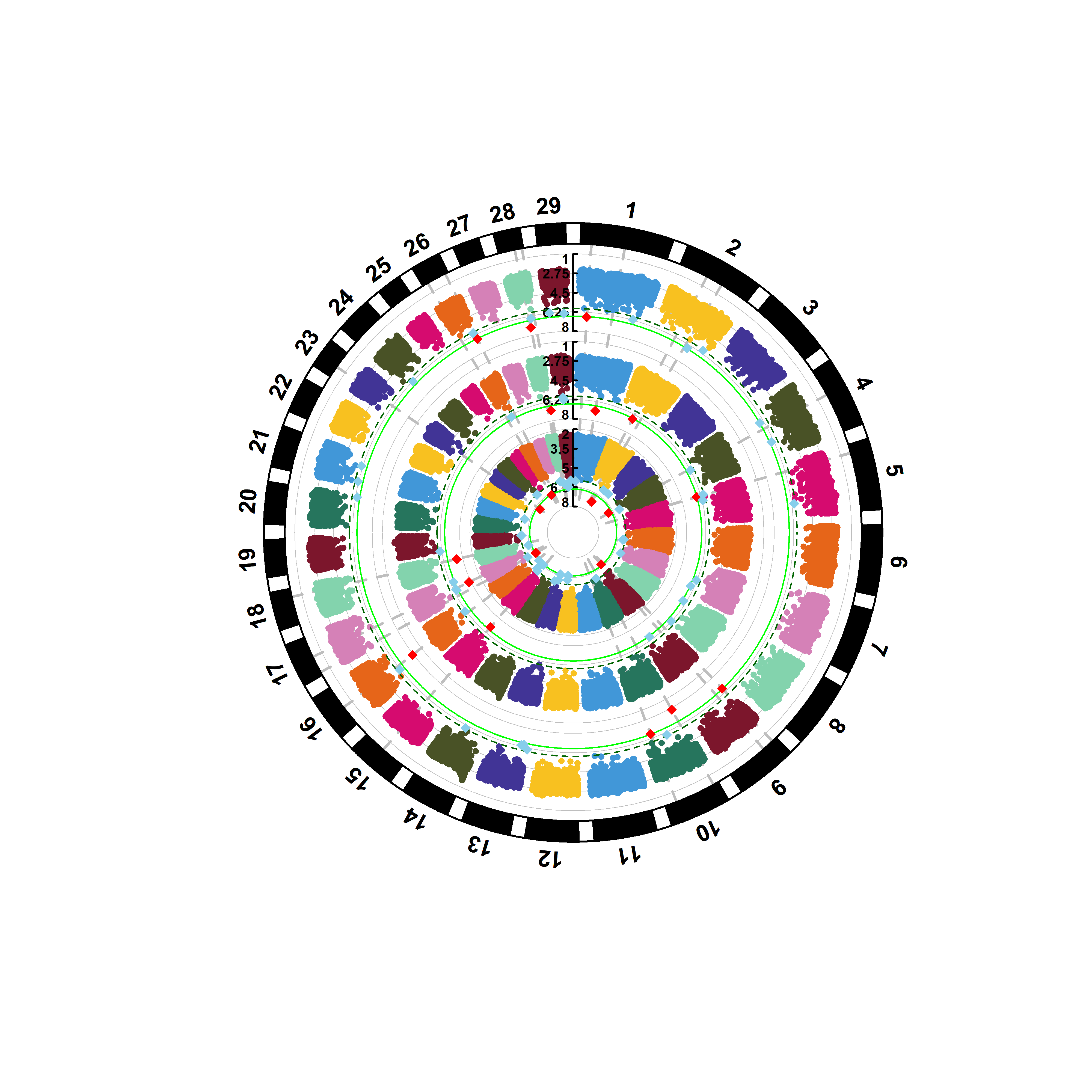
环形曼哈顿图
CMplot(cattle50K, plot.type=”c”, r=0.5, threshold=c(0.01,0.05)/nrow(cattle50K), cex = 0.5,
threshold.col = c(‘green’,’darkgreen’),threshold.lty = c(1,2),threshold.lwd = c(1,1),
amplify = T, cir.chr.h = 2,
signal.cex = c(1,1), signal.pch = c(18,18),signal.col = c(“red”,”skyblue”),file.output = F)
绘制QQ plot
CMplot(cattle50K,plot.type = “q”,multracks = T, threshold = 0.05, threshold.col = “red”,
amplify = T,signal.cex = 1.5, signal.pch = 20, signal.col = “skyblue”,
file.output = F)
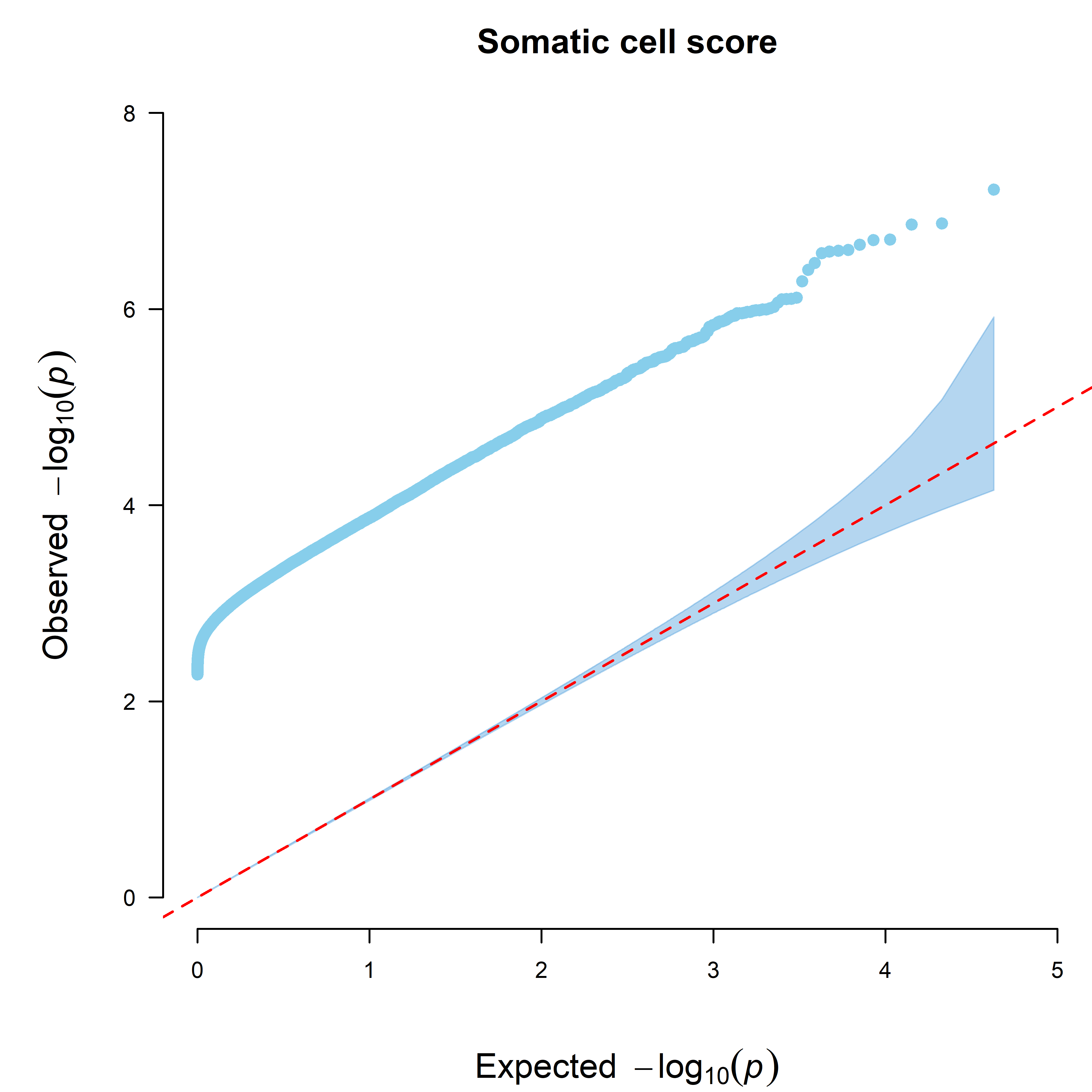
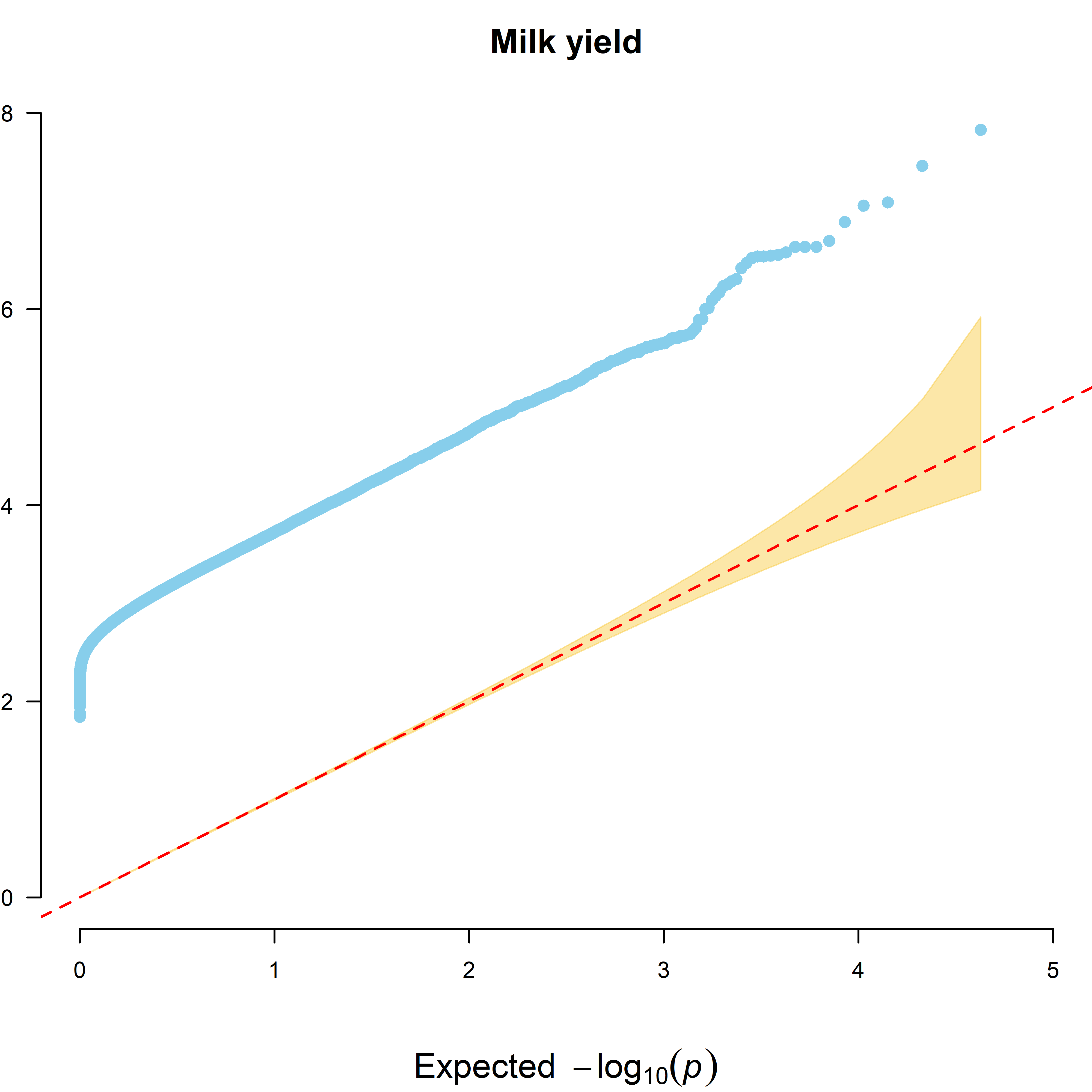
学会使用CMplot可以帮助我们简单快捷的绘制出精美的曼哈顿图,当我们遇到很多参数的时候不要害怕,小果在这里教大家一个小技巧–不停的去试,大胆的去修改参数,这样过后,自然而然地就知道每个参数的意义啦!
好啦今天小果的分享就到这里啦!希望今天小果的分享可以帮各位小伙伴及时的跟随研究的热点哦~如果小伙伴有其他数据分析需求,可以尝试使用本公司新开发的生信分析小工具云平台,零代码完成分析,非常方便奥,云平台网址为:(http://www.biocloudservice.com/home.html)