上一期小果使用了三种降维算法对单细胞数据进行了降维和聚类,将2700个细胞分成了9个细胞亚群。并比较了三种算法的分类效果,这期我们将在此基础上继续来探索这些细胞的奥秘。寻找每个细胞群的标志基因,最后根据标志基因识别细胞的类型。
下面就跟小果一起来做吧~
一、寻找标记基因
#寻找细胞群2的标记基因
cluster2.markers <- FindMarkers(pbmc, ident.1 = 2, min.pct = 0.25)
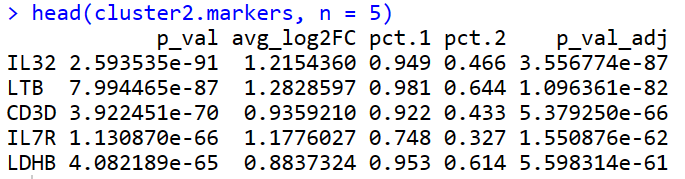
这里输出得行名是基因名,每一列分别是p值,细胞群2与其他细胞群相比平均得差异表达倍数。最后一列是校正后得p值。
使用FindMarkers函数即可寻找指定细胞群的标记基因。原理是计算和其他细胞群的表达差异倍率最大的基因,即为该细胞群的标记基因。
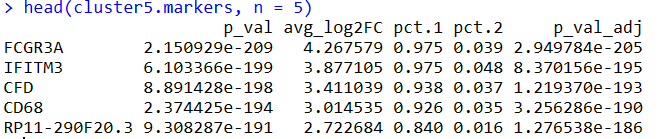
#查找区分细胞群5与细胞群0和3的所有标记
cluster5.markers <- FindMarkers(pbmc, ident.1 = 5, ident.2 = c(0, 3), min.pct = 0.25)
head(cluster5.markers, n = 5)
#一次寻找每个细胞群标记基因
pbmc.markers <- FindAllMarkers(pbmc, only.pos = TRUE, min.pct = 0.25, logfc.threshold = 0.25)
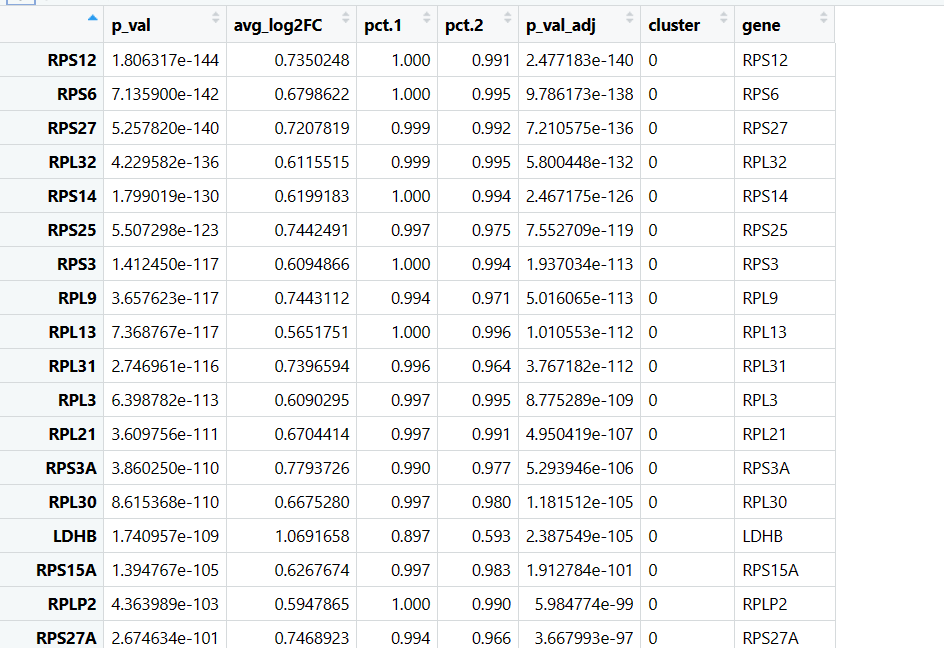
使用FindAllMarkers函数就能一次找出每一细胞群的标记基因。函数名非常直白,是不是非常好记呢。~
观察数据可以看到,后面增加了基因来自哪个细胞群,以及增加了一列gene的名称,便于数据操作。
top3 <- pbmc.markers %>% group_by(cluster) %>% top_n(n=3,wt=avg_log2FC)
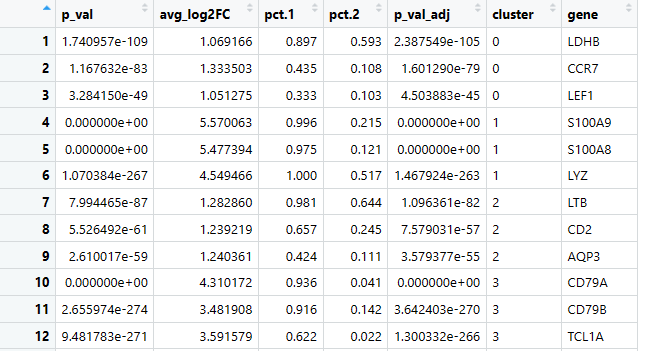
使用这样一行命令,通过管道操作,group_by将基因按细胞群分类,然后列出差异表达前三的基因。即为标记基因。
Seurat有几个检验算法,可以使用test.use参数进行设置。例如,这里使用的ROC测试返回任何单个标记的“分类能力”(范围从0-1)。还有wilcox、bimod、t、LR等检验算法,详情请使用?FindMarkers查看。
cluster0.markers <- FindMarkers(pbmc, ident.1 = 0, logfc.threshold = 0.25, test.use = “roc”, only.pos = TRUE)
二、标记基因可视化
Seurat提供了几个常用的可视化细胞群标记基因的工具,非常适合是数据探索与挖掘。以及论文插图绘制。
小提琴图
VlnPlot(pbmc, features = c(“MS4A1”, “CD79A”))
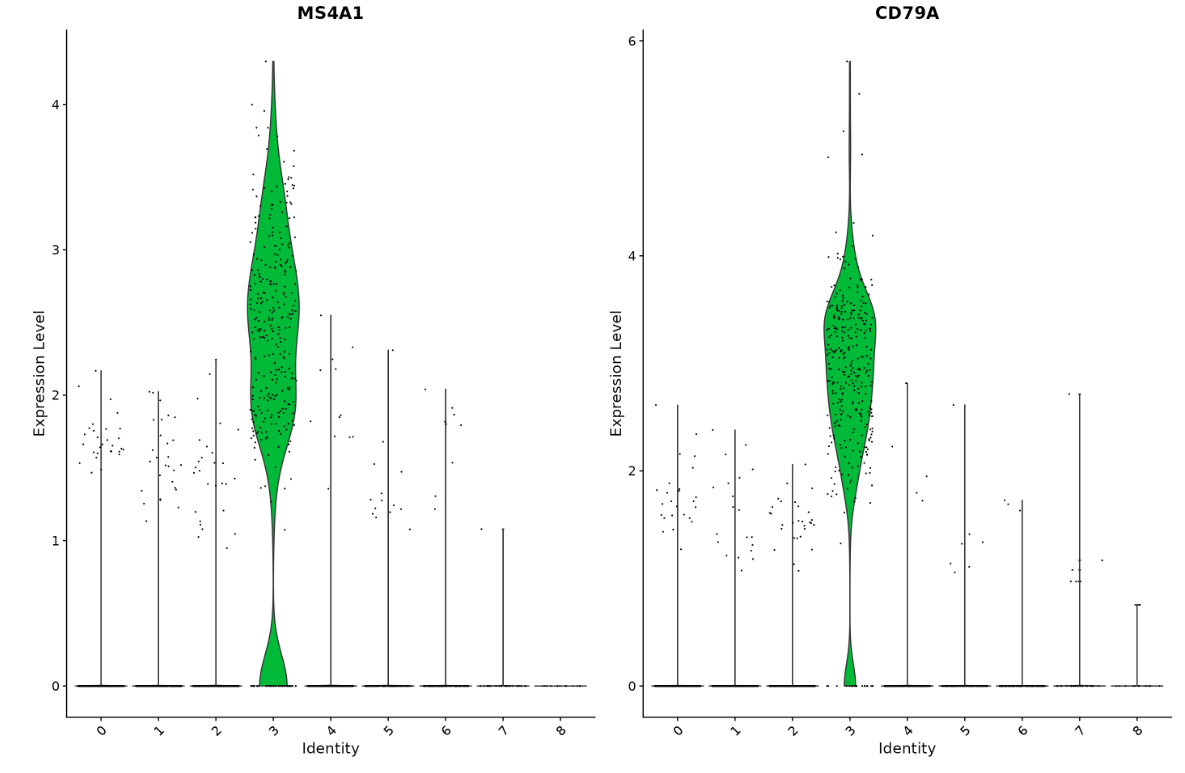
通过小提琴图可以明显看出”MS4A1″, “CD79A”是细胞群3的标记基因。
标记基因图
FeaturePlot(pbmc, features = c(“MS4A1”, “GNLY”, “CD3E”, “CD14”, “FCER1A”, “FCGR3A”, “LYZ”, “PPBP”,”CD8A”))
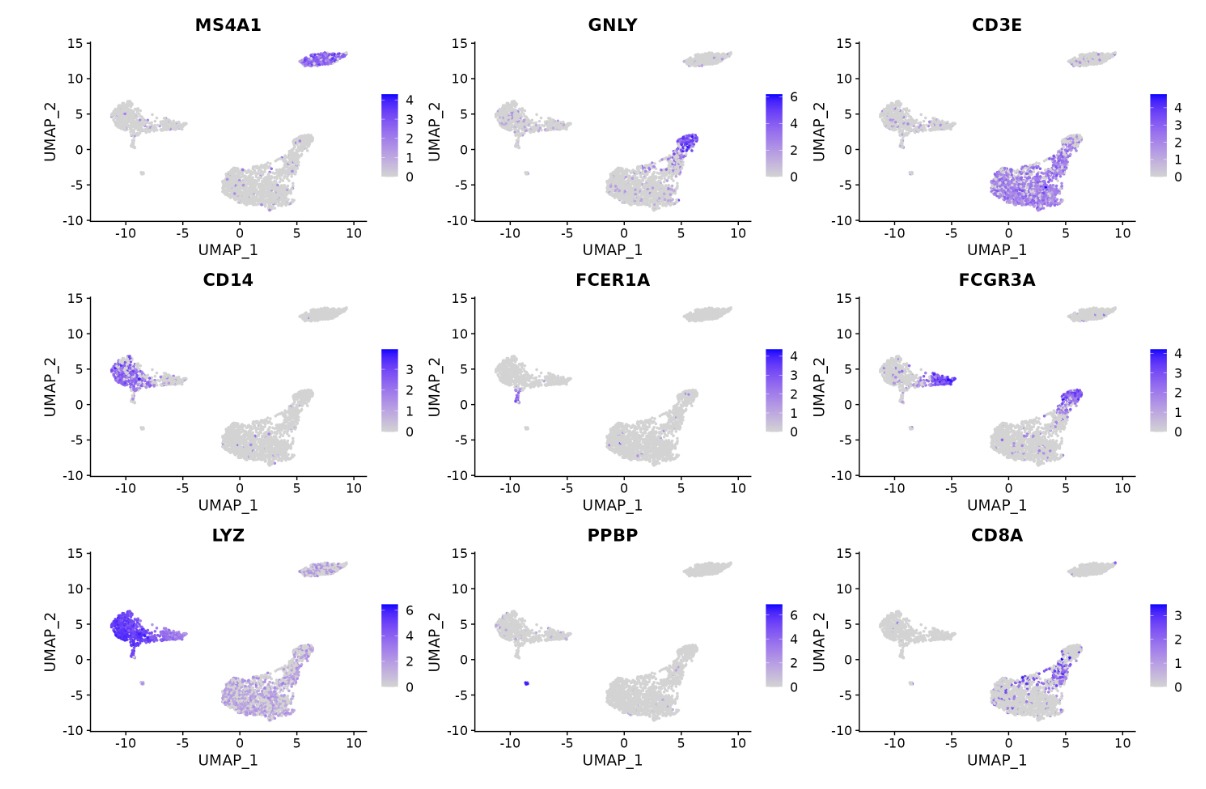
这个图是最常用的图之一。 features是提前挖掘的每个细胞群的标记基因。清楚显示了每个细胞群的标记基因。
热图
pbmc.markers %>%
group_by(cluster) %>%
top_n(n = 10, wt = avg_log2FC) -> top10
DoHeatmap(pbmc, features = top10$gene) + NoLegend()
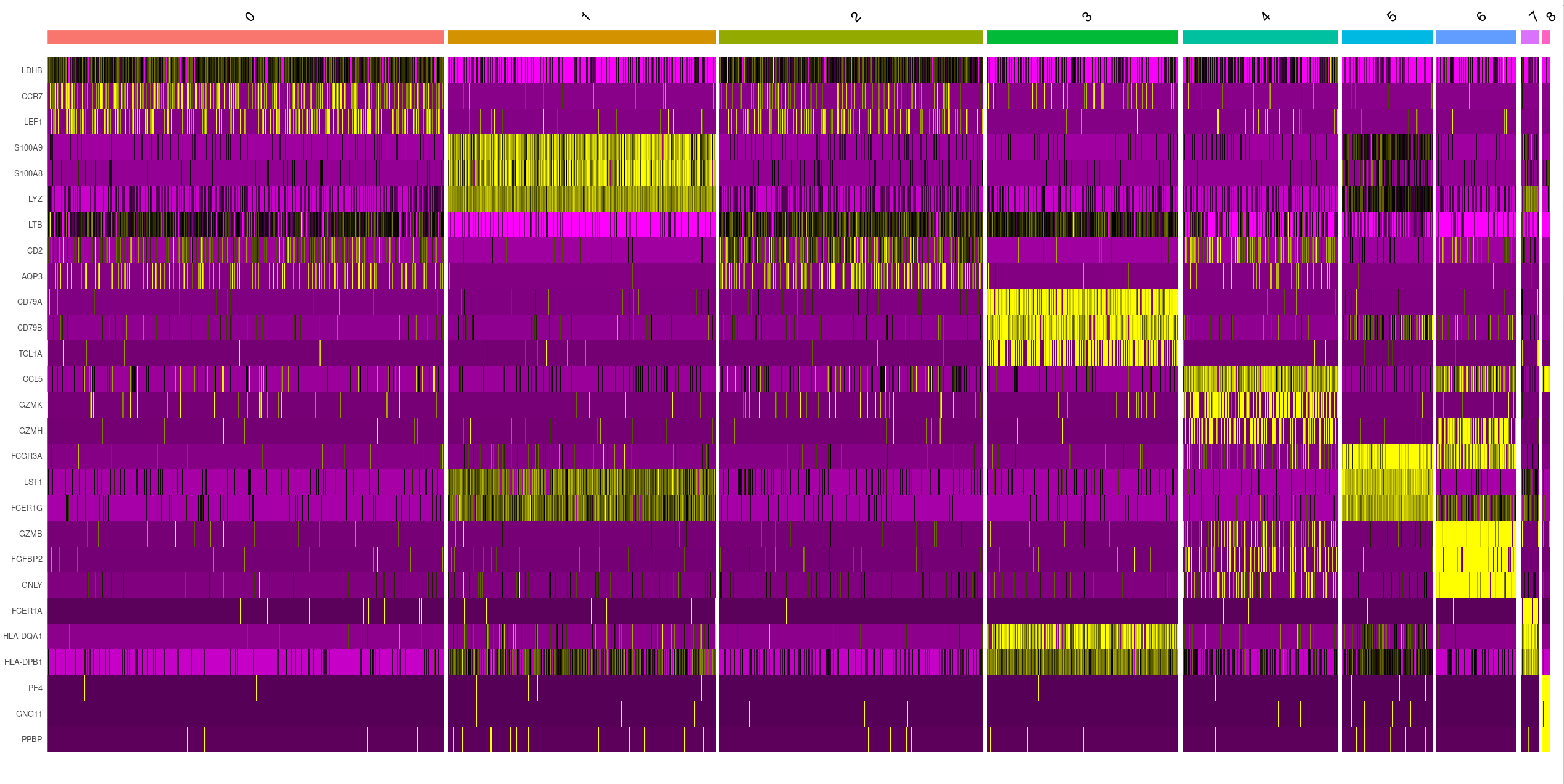
可以清晰的看出区分细胞群的标记基因(黄色部分)。
三、细胞鉴定
我们可以根据细胞的标志基因查阅其确切的细胞名称。在这个数据集的情况下,我们可以使用规范名称来轻松地将无偏聚类的细胞群与已知细胞类型相匹配:
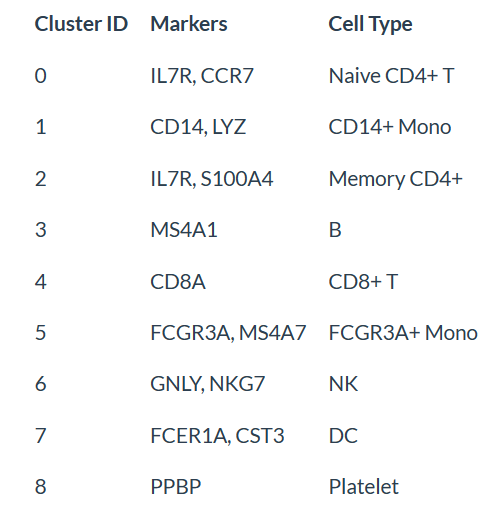
new.cluster.ids <- c(“Naive CD4 T”, “CD14+ Mono”, “Memory CD4 T”, “B”, “CD8 T”, “FCGR3A+ Mono”,”NK”, “DC”, “Platelet”)
names(new.cluster.ids) <- levels(pbmc)
#细胞群类重命名
pbmc <- RenameIdents(pbmc, new.cluster.ids)
#绘制UMAP图
DimPlot(pbmc, reduction = “umap”, label = TRUE, pt.size = 0.5) + NoLegend()
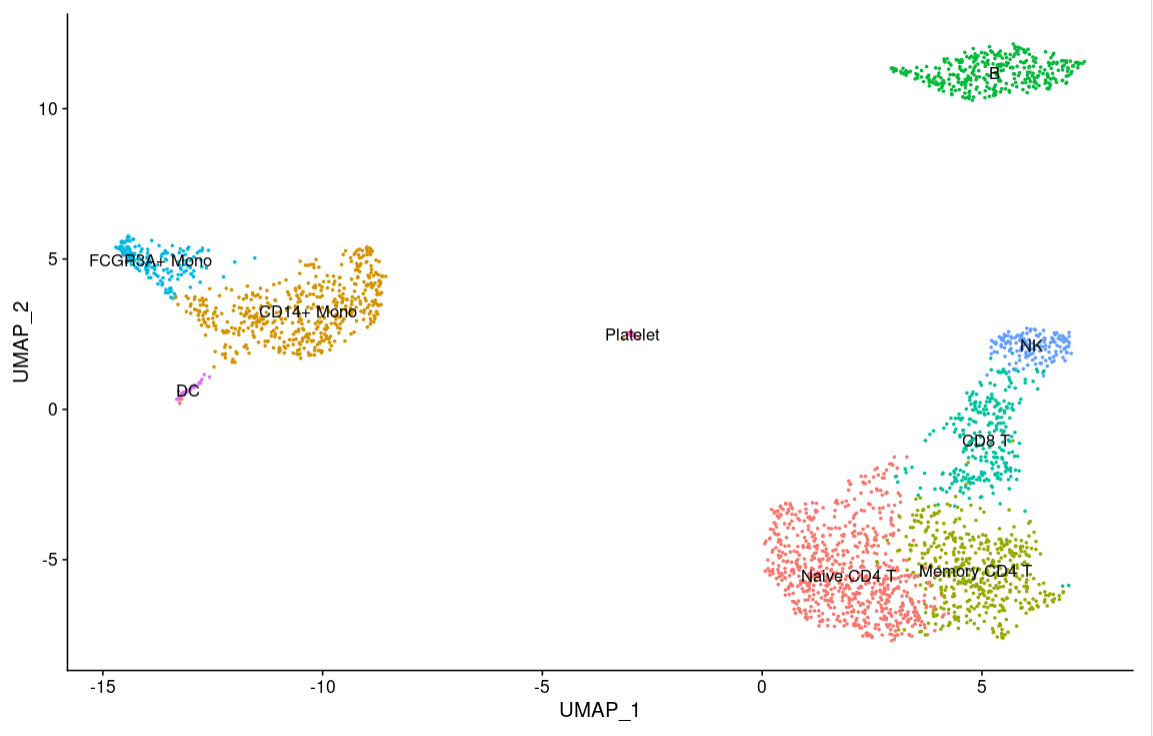
这样就识别出每个细胞群的类型了。
p1 <- DimPlot(pbmc, reduction = “umap”, label = TRUE, pt.size = 1.5) + NoLegend()
p2 <- DimPlot(pbmc, reduction = “tsne”, label = TRUE, pt.size = 1.5) + NoLegend()
p3 <- DimPlot(pbmc, reduction = “pca”, label = TRUE, pt.size = 1.5)
p1+p2+p3
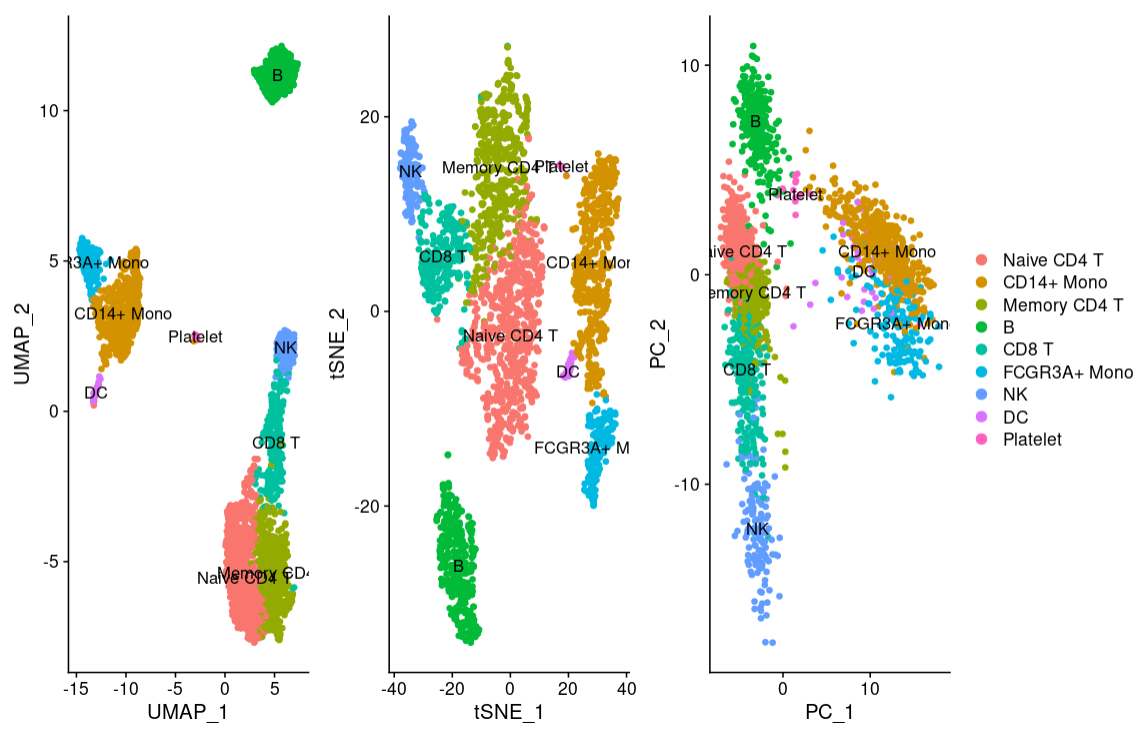
我们再对三种降维算法分类的细胞亚群进行比较,重新输出结果图。
四、输出软件版本信息
sessionInfo()
最后可以用sessionInfo()来输出软件版本信息,用于论文的补充材料当中。
小果的Seurat单细胞分析就到这里了。如果您跟着做了一次的话一定能对单细胞分析理解得更加深入。小果还会为大家带来更多更优质的学习资源。我们下次见啦~