小果在最近发现了一个处理序列时候发现了一个好用的生信小工具。今天小果把它分享给你们~
简介
Seqkit 是一个快速、高效、功能丰富的生物信息学工具,用于处理DNA、RNA和蛋白质序列。它提供了一系列命令行工具和API接口,可以进行序列文件格式转换、序列处理和分析、序列统计等操作。
首先,Seqkit 支持常见的序列文件格式,如FASTA、FASTQ、GTF、BED等,可以方便地实现不同文件格式之间的转换。
其次,除了基本的文件格式转换外,Seqkit 还有很多强大的功能。例如,它可以对序列进行修剪、截取、过滤,并支持对序列长度、GC含量、碱基比例等进行统计分析。此外,还可以根据参考序列进行比对与比对结果处理以及进行序列互补、反向互补、反向互补互补等常用操作。
下载安装
1、页面下载
最新版发布页面:https://github.com/shenwei356/seqkit/releases
可以复制上述的连接中适配自己电脑系统的连接
这里以Linux上为例子
wget https://github.com/shenwei356/seqkit/releases/download/v2.5.0/seqkit_linux_amd64.tar.gz
tar -zxvf seqkit_linux_amd64.tar.gz
![]()
上述图片中绿色的就是我们解压后的小工具了,可以直接使用啦
2、conda安装
conda install -c bioconda seqkit
命令解析
seqkit 一共有37个可用的命令
amplicon 通过引物检索扩增子(或其周围的特定区域)
bam 检查和在线绘制BAM记录文件的直方图
common 通过id/名称/序列查找多个文件的公共序列
concat 连接多个文件中具有相同ID的序列
convert 转换FASTQ质量编码格式:支持格式包括:桑格,Solexa和Illumina
duplicate 重复序列N次
faidx 创建FASTA索引文件并提取子序列
fish 使用局部比对在较大的序列中寻找短序列
fq2fa 转换FASTQ到FASTA
fx2tab 将FASTA/Q转换为表格格式(包含长度/GC含量/GC偏好)
genautocomplete 生成shell自动完成脚本
grep 通过ID/name/sequence/sequence motif搜索序列,允许错配
head 打印第一条序列
help 打印帮助信息
locate 定位序列,或者motifs,允许错配
mutate 编辑序列(点突变、插入、删除)
pair 匹配双端序列文件
range 打印一个范围内的序列
rename 重命名重复序列ID
replace 使用正则表达式修改名称或者序列
restart 重置环状基因组的起始位置
rmdup 通过id/名称/序列删除重复的序列
sample 按数量或比例对序列进行抽样
sana 清理损坏的单行fastq文件
scat real time recursive concatenation and streaming of fastx files
seq 转换序列(反向,补充,提取ID…)
shuffle 随机序列
sliding 序列滑窗提取,支持环形基因组
sort 按id/名称/序列/长度排序序列
split 按id/seq区域/大小/部件将序列拆分为文件(主要用于FASTA)
split2 按序列数量/文件数将序列拆分为多个文件(FASTA, PE/SE FASTQ)
stats FASTA/Q文件的简单统计
subseq 通过region/gtf/bed得到子序列,包括侧翼序列
tab2fx 转换表格格式为FASTA/Q格式
translate 翻译DNA/RNA到蛋白质序列(支持歧义碱基)
version 打印版本信息并检查是否更新
watch 序列特征的监测和在线直方图
实践操作
因为sepkit的工具太强大了,这里列举一些我们常用常见的操作
下载两个序列做为示例操作,一个fastq文件,一个fasta文件
wget http://data.biostarhandbook.com/reads/duplicated-reads.fq.gz
wget ftp://ftp.ncbi.nih.gov/refseq/release/viral/viral.1.1.genomic.fna.gz
stats 简单统计
#统计序列格式fasta(fa)/fastq(fq)、内容类型DNA/RNA/Protein,序列数量、总长度,最小、平均和最大长度
# FASTA DNA
seqkit stats viral.1.1.genomic.fna.gz

# FASTQ DNA
seqkit stats duplicate.fastq
seq 序列操作
“-n”: 提取序列ID,包括“>”后面的全部内容
seqkit seq -n viral.1.1.genomic.fna.gz |head
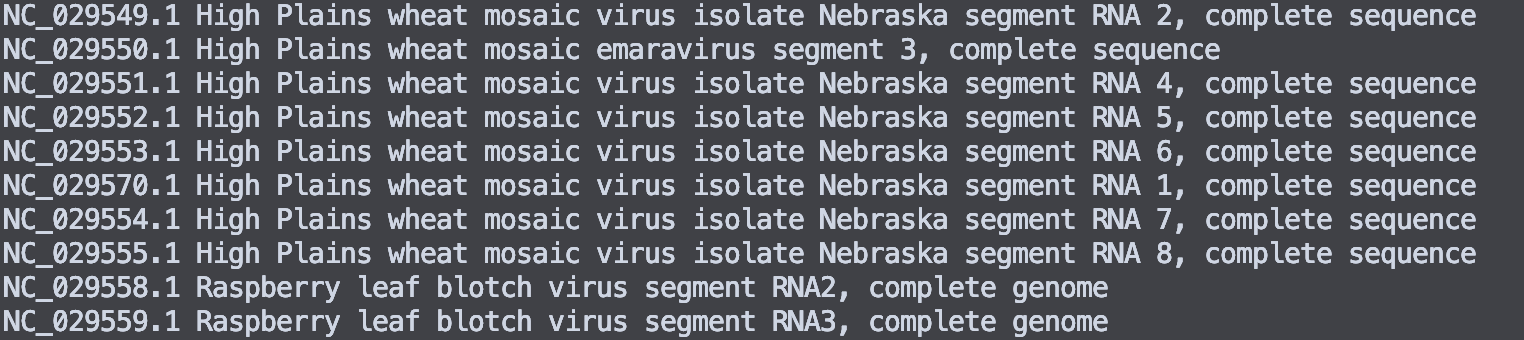
“-n -i”: 仅提取第一个空格前的ID
seqkit seq -n -i viral.1.1.genomic.fna.gz |head
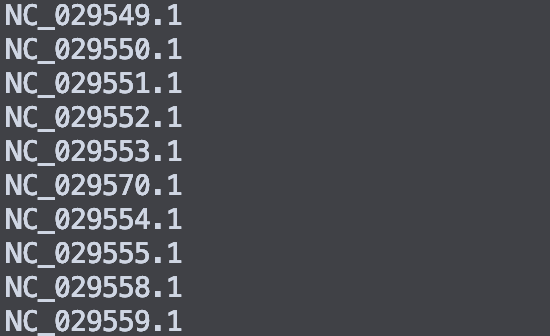
提取序列长度大于50的并统计长度信息
seqkit seq -m 50 viral.1.1.genomic.fna.gz |seqkit stats

设置最小序列长度和最大序列长度,用于过滤序列,并统计
seqkit seq -m 100 -M 1000 viral.1.1.genomic.fna.gz
- 保存>100且<1000长度的序列
seqkit seq -m 100 -M 1000 viral.1.1.genomic.fna.gz >a_100_1000.fa
单行/多行转换
-s提取并展示序列
-w 代表每行的碱基数量,0代表不换行
seqkit seq viral.1.1.genomic.fna.gz -s -w 0 |head
-r 序列反向 -p序列互补
# 序列反向互补,-r反向,-p互补
echo -e “>123\nATCG”|seqkit seq -r -p

删除gap/大小写转换
-g 去除序列中的间隔,将中间的横杠去掉
-u转化序列为大写字母展示
echo -e “>seq\nACGT-ACTGC-acc” | seqkit seq -g -u

—rna2dna 将RNA序列转化为DNA序列
echo -e “>seq\nUCAUAUGCUUGUCUCAAAGAUUA” | seqkit seq –rna2dna
subseq指定区域
-r 通过区域来截取序列2:10提取2到10个碱基,-12:-1提取序列结尾12个碱基;
seqkit subseq -r 2:10 viral.1.1.genomic.fna.gz |head -2

fq2fa 将fq转为fa格式
seqkit fq2fa duplicate.fastq -o duplicate.fa
head duplicate.fa

fx2tab & tab2fx 序列转化表格格式
fx2tab
这一转化非常的有用,往往用于表格/矩阵处理的时候
seqkit fx2tab viral.1.1.genomic.fna.gz | less -S
# 打印序列长度、GC含量
seqkit fx2tab viral.1.1.genomic.fna.gz -l -g -n -i -H | head
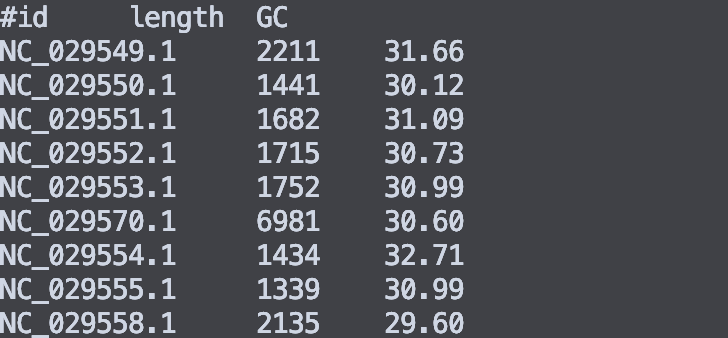
#通过矩阵格式的序列文件统计序列长度和质量值
-l 统计序列长度
-g 统计平均GC含量
-i 只打印名称(不打印序列)
-H 打印标题行
tab2fx
seqkit fx2tab viral.1.1.genomic.fna.gz |seqkit tab2fx |head

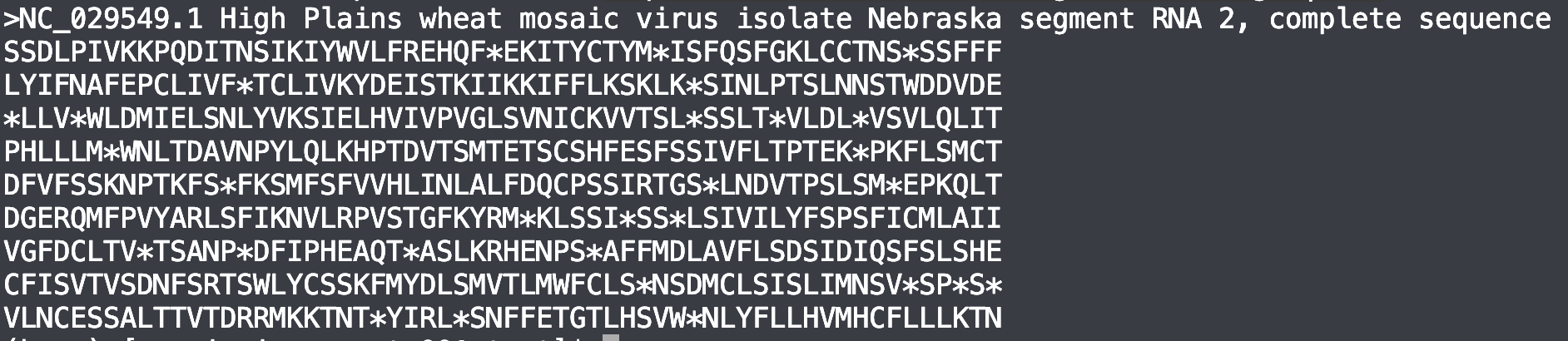
translate 翻译DNA/RNA为蛋白质序列
seqkit translate viral.1.1.genomic.fna.gz |head
faidx 建立索引文件、提取子序列
seqkit faidx viral.1.1.genomic.fna
seqkit faidx viral.1.1.genomic.fna NC_030200.1 #提取ID信息
seqkit faidx viral.1.1.genomic.fna NC_030200.1 # -f 标题全部输出
seqkit faidx viral.1.1.genomic.fna NC_030200.1:1-10 #提取子序列第1个到第10个碱基

grep 匹配
seqkit grep -f id.txt viral.1.1.genomic.fna -o result.fq.gz#按 ID 列表文件搜索(不包含空格)
![]()
seqkit grep -n -f name.txt viral.1.1.genomic.fna -o result.fa.gz #使用序列名称列表进行搜索
cat viral.1.1.genomic.fna | seqkit grep -s -i -p aggcg #提取包含 AGGCG 的序列
zcat viral.1.1.genomic.fna | seqkit grep -s -r -i -p ^aggcg #提取以 AGGCG 开头的序列
#-i参数忽略大小写
spilt 拆分
seqkit split viral.1.1.genomic.fna -s 10000 #将序列拆分为按照10000个序列的部分
seqkit split viral.1.1.genomic.fna -p 4 #将序列拆分为4部分
seqkit split viral.1.1.genomic.fna -p 4 -2#加上-2减少内存使用
seqkit split viral.1.1.genomic.fna -i –id-regexp “^([\w]+)\-” -2 #按id拆分序列
seqkit split viral.1.1.genomic.fna -r 1:3 -2 #按前三个序列碱基来区分

seqkit小工具隐藏很多好用的功能,需要我们不断尝试,小果把软件的github连接给你们
https://github.com/shenwei356/seqkit,希望你遇到有关处理数据的问题,可以尝试用这个万能小工具来解决~
如果遇到不懂的也可以借助线上的云平台哦~http://www.biocloudservice.com/home.html
好了今天的seqkit小软件就讲到这里,欢迎大家有问题与小果一起讨论哦~

