MatrixGenerics是一款强大且与生物学相关的R语言包,它是由Martin Morgan在R语言社区中开发的。Martin Morgan是一位资深的生物信息学家和R语言程序员,他在R语言生态系统中贡献了许多有用的包和工具。该包的设计强调了代码的模块化和可扩展性,这使得用户能够更轻松地编写可读性强、可维护性好的代码。
生物学领域的数据通常以矩阵或数组的形式存储,例如基因表达数据、DNA测序数据、蛋白质互作网络等。MatrixGenerics包的主要目标是为这些复杂的数据结构提供通用的函数,简化生物学家们对数据的处理和分析。其核心功能是在不同类型的矩阵和数组上执行通用操作,例如计算均值、求和、标准差以及行列变换等。
要使用MatrixGenerics包,可以在R中使用以下命令进行安装和加载:
> install.packages(“MatrixGenerics”) #安装MatrixGenerics语言包
> library(MatrixGenerics) #加载语言包
MatrixGenerics的优势之一是其对广泛的矩阵类对象的支持,包括Matrix包中定义的各种特殊矩阵类型,如dgCMatrix、dgTMatrix、dsCMatrix等。这使得生物学家可以在不同的数据类型之间无缝切换,并且不必为每种数据类型单独编写函数。
此外,MatrixGenerics还支持用户自定义矩阵类的扩展,使其能够适应特定的生物学数据需求。用户可以根据自己的需求定义新的矩阵类,并在其上应用MatrixGenerics的函数,从而更好地处理生物学数据。
示例:
# 安装和加载MatrixGenerics包
> install.packages(“MatrixGenerics”)
> library(MatrixGenerics)
# 加载其他必要的包
> install.packages(“ggplot2”)
> library(ggplot2)
接下来,我们创建一个模拟的基因表达数据集,并将其转换为Matrix对象,我们可以使用MatrixGenerics包中的函数来对基因表达数据进行处理和可视化。
# 创建模拟基因表达数据
> gene_names <- c(“Gene1”, “Gene2”, “Gene3”, “Gene4”, “Gene5”)
> sample_names <- c(“Sample1”, “Sample2”, “Sample3”)
> expr_data <- matrix(data = rnorm(15, mean = 10, sd = 2), nrow = 5, ncol = 3, dimnames = list(gene_names, sample_names))
# 将数据转换为Matrix对象
> expr_data_matrix <- as(expr_data, “matrix”)
> # 计算每个基因的平均表达值
> gene_means <- rowMeans(expr_data_matrix)
# 可视化每个基因的平均表达值
> gene_means_plot <- ggplot(data.frame(gene = gene_names, mean_expr = gene_means), aes(x = gene, y = mean_expr)) +
+ geom_bar(stat = “identity”) +
+ labs(title = “Average Gene Expression”,
+ x = “Gene”,
+ y = “Mean Expression”)
> print(gene_means_plot)
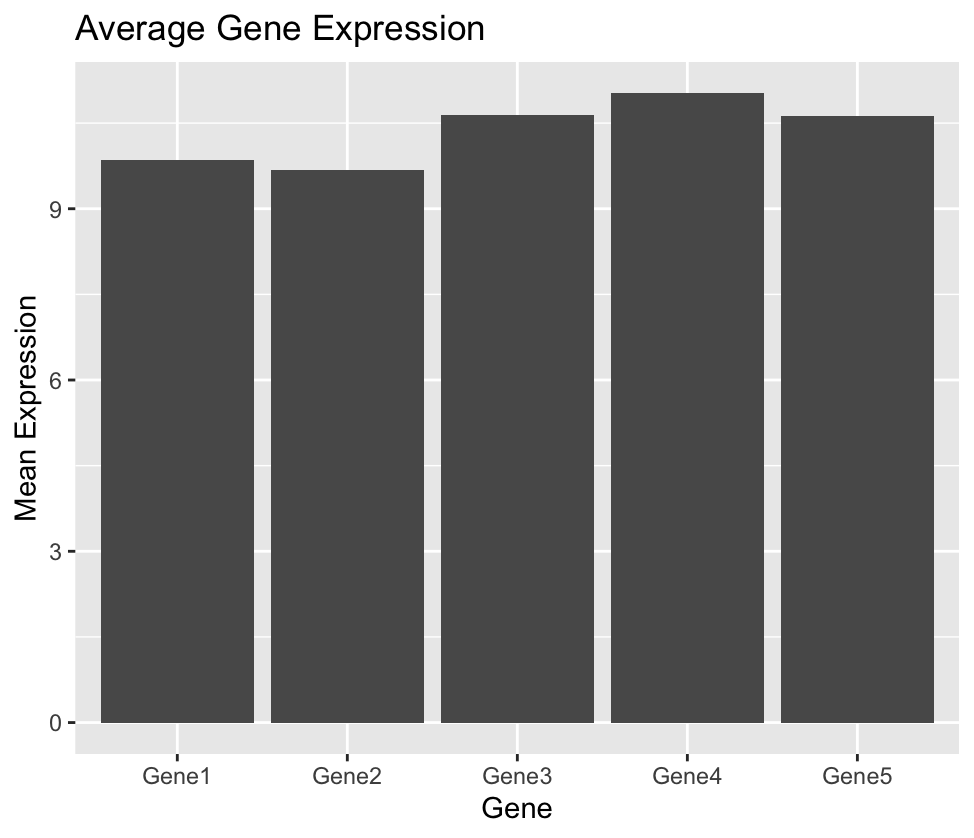
接下来,我们计算每个样本的总体表达量,并进行可视化。
# 计算每个样本的总体表达量
> sample_totals <- colSums(expr_data_matrix)
# 可视化每个样本的总体表达量
> sample_totals_plot <- ggplot(data.frame(sample = sample_names, total_expr = sample_totals), aes(x = sample, y = total_expr)) +
+ geom_bar(stat = “identity”) +
+ labs(title = “Total Expression per Sample”,
+ x = “Sample”,
+ y = “Total Expression”)
> print(sample_totals_plot)
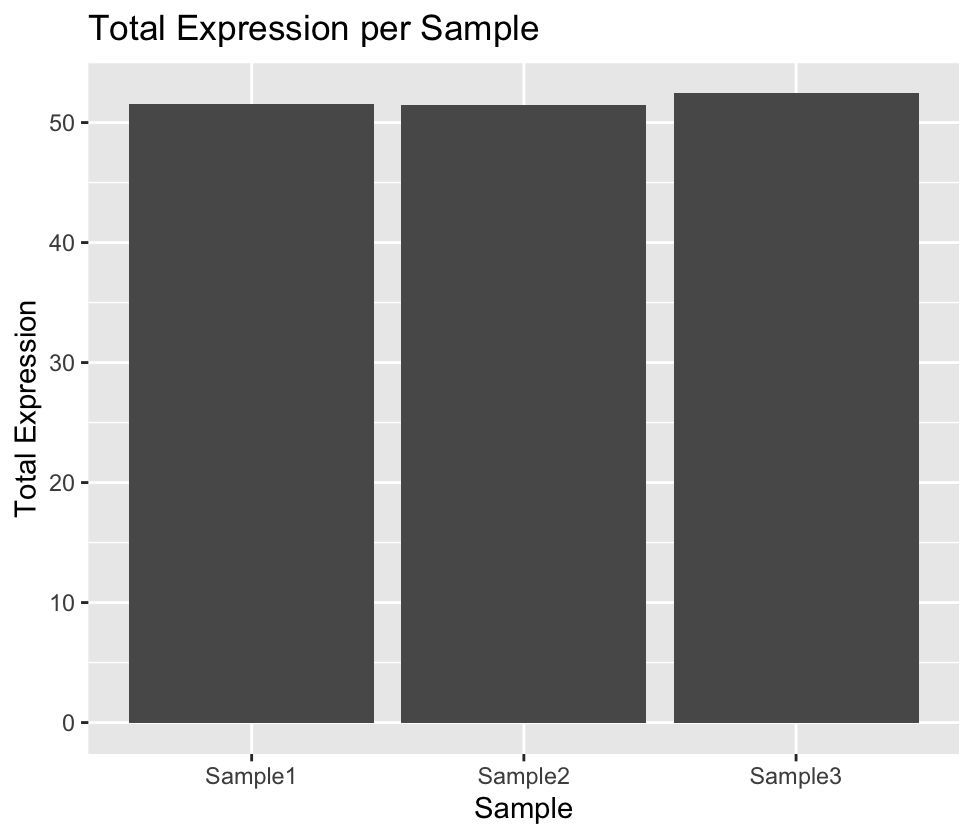
在这个示例中,我们使用MatrixGenerics包的rowMeans和colSums函数计算了每个基因的平均表达值和每个样本的总体表达量。然后,我们使用ggplot2包进行了简单的可视化,分别展示了每个基因的平均表达值和每个样本的总体表达量。
在生物学数据分析中,特别是基因组学和蛋白质组学研究中,数据集往往非常庞大,因此对性能的要求非常高。MatrixGenerics通过利用高效的底层算法和优化技术,提供了出色的性能和计算效率。这为生物学家处理大规模数据提供了便利,使得他们可以更快地进行探索性研究、数据可视化和统计分析。

以上就是对R语言包MatrixGenerics的简单介绍啦,它的通用框架使得处理不同类型的矩阵和数组数据变得更加简单和高效。对于处理庞大的生物学数据集,它的优化算法和高性能确保了快速的数据处理能力。如果你是一个生物学家或生物信息学家,MatrixGenerics无疑会是你数据分析工作中不可或缺的助手。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html
References: