在上一次的分享中,小果给大家演示了如何使用Discovery Studio设计及优化小分子配体结构,那么这一次,小果就来给大家分享一下如何使用Discovery Studio来优化蛋白质结构,使其可以在后续与我们设计好的小分子配体进行对接等一系列操作。废话不多说,跟上小果直接开始吧!
为什么要优化蛋白质结构?
如果想要真正理解药物设计的流程,不能只知道该如何操作,还要理解每一步的意义何在。可能有些小伙伴还不知道为什么要优化蛋白质结构以及优化后的蛋白质有什么用处,下面小果就给大家详细地介绍一下:
处理后的蛋白质用途
当处理后的蛋白质结构应用于药物分子设计领域时,它可以发挥以下作用:
- 药物模式识别:即确定蛋白质与药物结合相关的结构模式。通过计算和模拟分析,可以识别出与药物分子相互作用的关键氨基酸残基或结构域,这有助于指导药物分子的设计,使其能够在特定的蛋白质位点上实现最佳结合,并发挥治疗效果。
- 药物偶联物设计:如设计抗体药物或载体。通过了解蛋白质结构的特定区域和位点,可以将药物分子连接到适当的载体上,实现药物精确输送、靶向释放等功能。这有助于提高药物的生物利用度和针对性,减轻剂量和副作用。
- 药物代谢预测:通过模拟药物与代谢酶的结合,可以预测药物的代谢途径、代谢产物以及代谢速率。这对于识别潜在的代谢问题,优化药物的代谢稳定性和代谢特性具有重要意义。
- 药物副作用评估:通过分析药物与非靶蛋白质的交互作用,可以发现可能引起副作用的结构特征或亲和性。这有助于提前识别潜在的不良反应,并为药物优化和副作用管理提供指导。
- 药物抗性机制研究:通过分析蛋白质结构中的变异位点和突变模式,可以揭示药物抗性所涉及的结构和功能变化。这有助于理解药物失效的原因,并为克服药物抗性提供策略和指导。
总之,通过分析预测处理后的蛋白质与药物的各种作用效果能够为药物的设计和优化提供关键信息,确保药物的高效性、选择性和安全性,因此处理蛋白是药物分子设计中不可缺少的一环。
为什么一定要处理?
通过上面的分析,小果相信小伙伴们已经理解了蛋白质在药物分子设计中的重要作用,那么可能会有小伙伴问到,为什么一定要对蛋白质结构进行处理呢?可不可以不处理?如果不处理会怎么样呢?善于思考是一项非常好的优点,下面小果就给大家解答疑惑:
需要对蛋白质结构进行优化的原因如下:
- 提高药物的亲和力:通过优化蛋白质结构,药物与靶蛋白之间的相互作用可以被改善。这有助于增加药物对目标蛋白的亲和力,提高药物与靶标的结合效率和稳定性。
- 增加药物的选择性:蛋白质结构的优化可以帮助设计具有更高选择性的药物。通过了解靶蛋白的结构细节,可以为药物设计提供指导,使其能够更精确地与目标蛋白相互作用,减少与其他非靶蛋白的相互作用,从而降低非特异性副作用。
- 改进药物代谢和稳定性:通过优化蛋白质结构,可以改变药物与代谢酶的相互作用,影响药物的代谢途径、代谢速率和半衰期。这有助于改善药物的代谢稳定性,延长药物在体内的停留时间,提高治疗效果。
- 减轻药物副作用:优化蛋白质结构可以帮助预测和评估药物的副作用。通过分析药物与非靶蛋白质的相互作用,可以发现可能引起副作用的结构特征或亲和性。从而可以提前识别潜在的不良反应,并为药物设计和副作用管理提供指导。
- 克服药物抗性:蛋白质结构的优化有助于研究和克服药物抗性的机制。通过分析蛋白质结构中的变异位点和突变模式,可以揭示药物抗性所涉及的结构和功能变化。这有助于寻找对抗药物抗性的策略,重新设计药物分子以恢复治疗效果。
综上所述,通过优化蛋白质结构可以提高药物的亲和力和选择性,改善药物代谢和稳定性,减轻药物副作用,并克服药物抗性。这些优化措施有助于提高药物疗效、降低不良反应,因此是至关重要、不可省略的步骤。
如何获得蛋白质结构?
小伙伴们看到这里一定已经理解了优化蛋白质的重要性,现在是不是已经迫不及待地想看一看如何操作了呢?但是不要忘记,在我们处理之前,需要先获得蛋白质结构才可以,不然我们处理什么呢?获取蛋白质结构,小果一般会选择在RCSB PDB中进行搜索和下载。下面就让小果给大家介绍一下这个实用的数据库吧:
PDB(Protein Data Bank)是一个国际知名的蛋白质结构数据库,致力于收集、存储和分发生物大分子(尤其是蛋白质)的三维结构数据。作为全球科学界的重要资源,它为研究者们提供了一个广泛和可靠的平台,以便进行生物大分子结构的研究、分析和解释。PDB在全球有多个分站,其中RCSB PDB(Research Collaboratory for Structural Bioinformatics Protein Data Bank)是美国分站,使用最为广泛。
RCSB PDB的数据来源非常多样,其中包括了通过X射线晶体学、核磁共振等实验技术获得的蛋白质结构数据,也就是说这些数据均通过实际实验得来而非仅仅模拟预测。这些数据以PDB文件格式进行存储,其中包含了关于原子坐标、结构拓扑、分子间相互作用等重要信息,能够描述蛋白质分子在空间中的立体构象。
RCSB PDB提供了多种工具和资源,帮助研究者们进行蛋白质结构的可视化、模拟计算和功能注释。研究者们可以使用PDB ID或关键词进行搜索,获取感兴趣的蛋白质结构数据,并可通过在线工具进行结构可视化、结构比对和结构分析等操作。此外,RCSB PDB还提供了高质量的教育资源,用于培训和教学,以推广蛋白质科学的学习和应用。
非常值得推崇的一点是,PDB数据库中的数据均为共用数据,使用起来不需要担心著作权的问题,因此小果非常推荐大家使用。
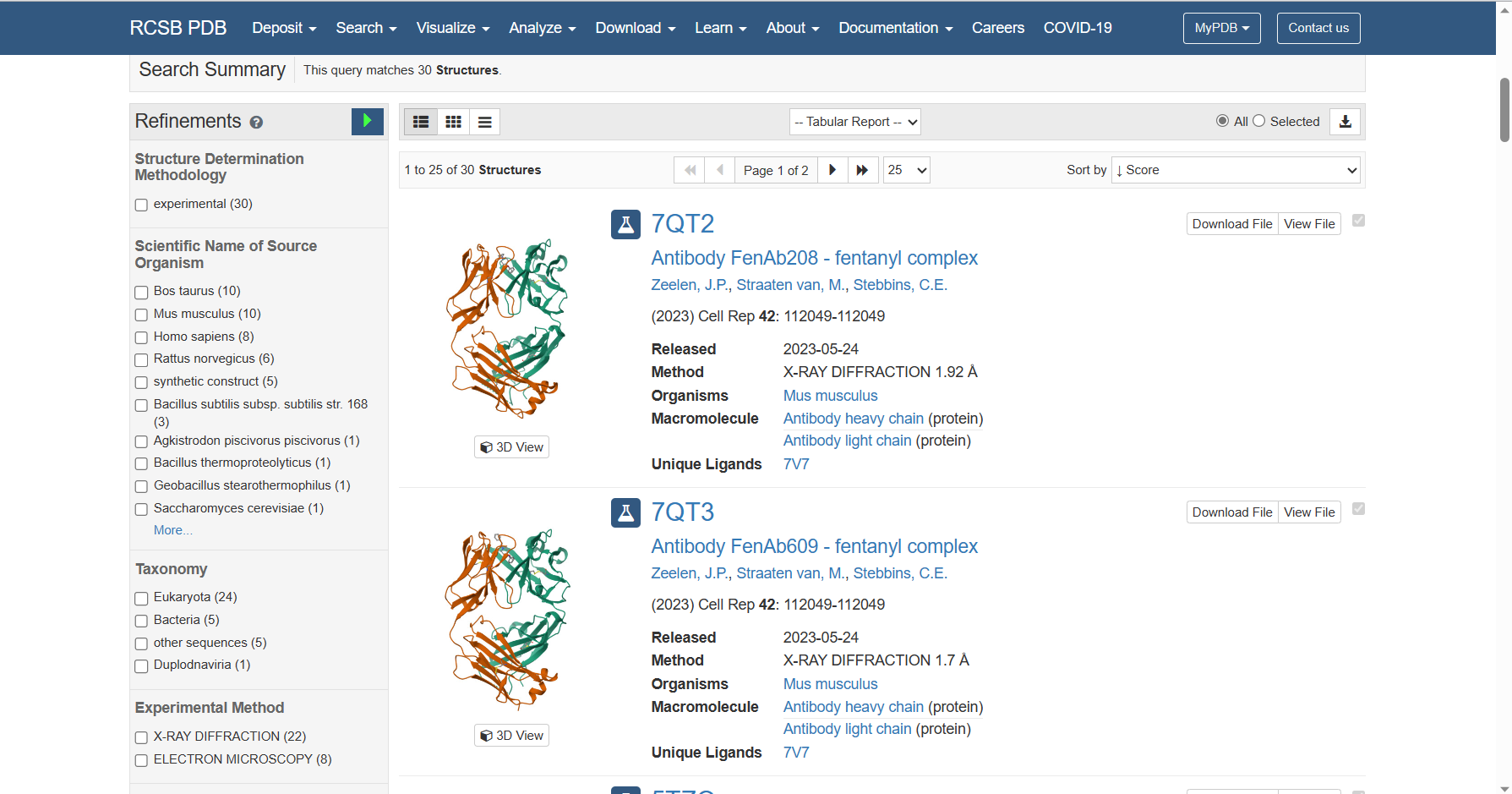
看到这里相信大家已经理解了RCSB PDB的厉害之处,那么我们该如何使用它呢?在右上角我们可以直接输入感兴趣的蛋白质名称、PDB ID或关键词来搜索,甚至我们可以直接输入配体的名称来检索结构中包含有该配体的蛋白质。
除了直接搜索以外还有一个非常实用的功能就是高级搜索(advanced search)可以帮助我们进一步缩小搜索得到的范围:
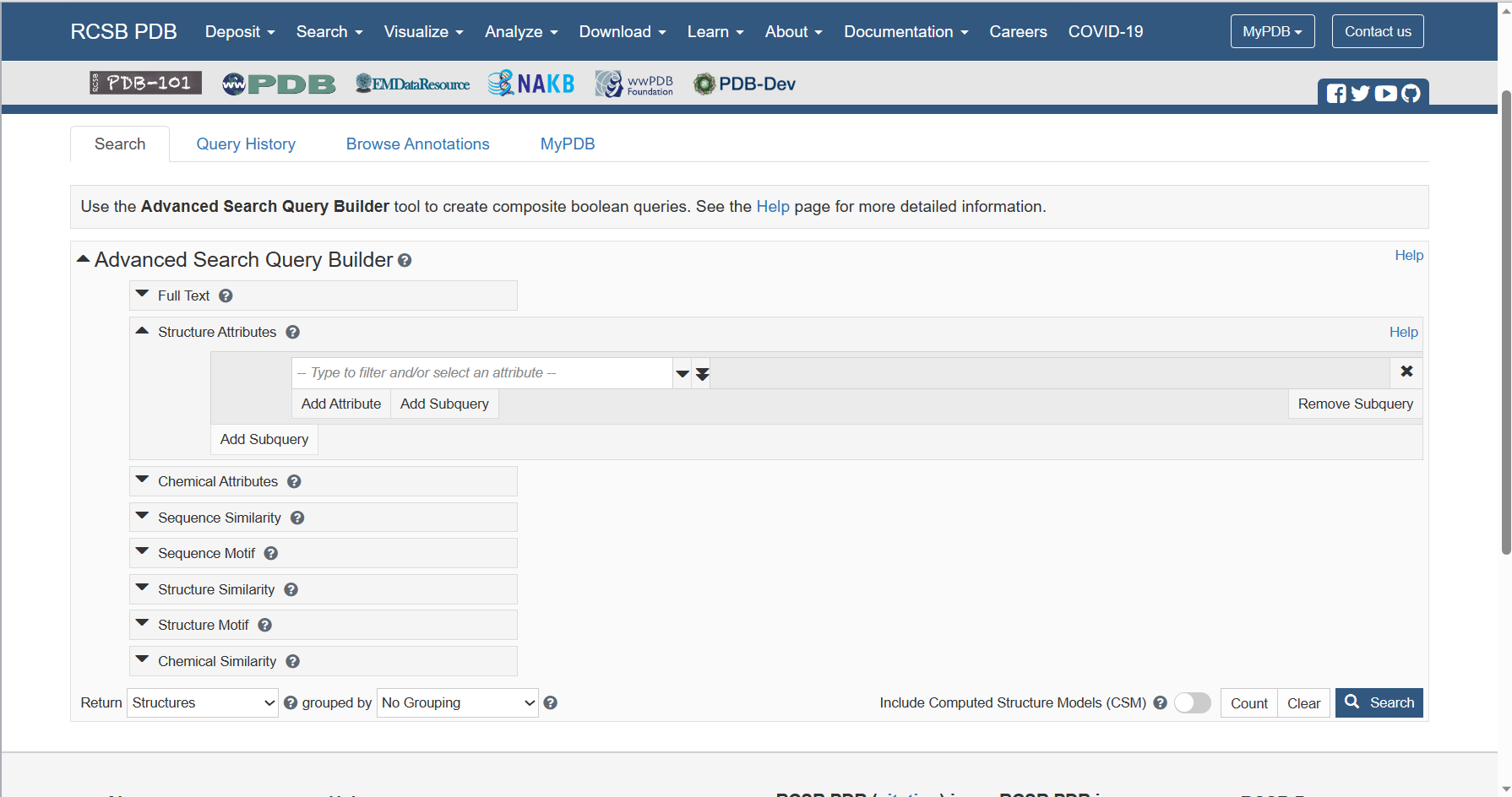
关于高级搜索如何使用以及其他用法如如何查看结果页,我们可以看到在页面中给出了Help按钮,感兴趣的小伙伴们可以点进去了解一下,官方给的使用方法还是非常详细的,小果在这里就不赘述啦。
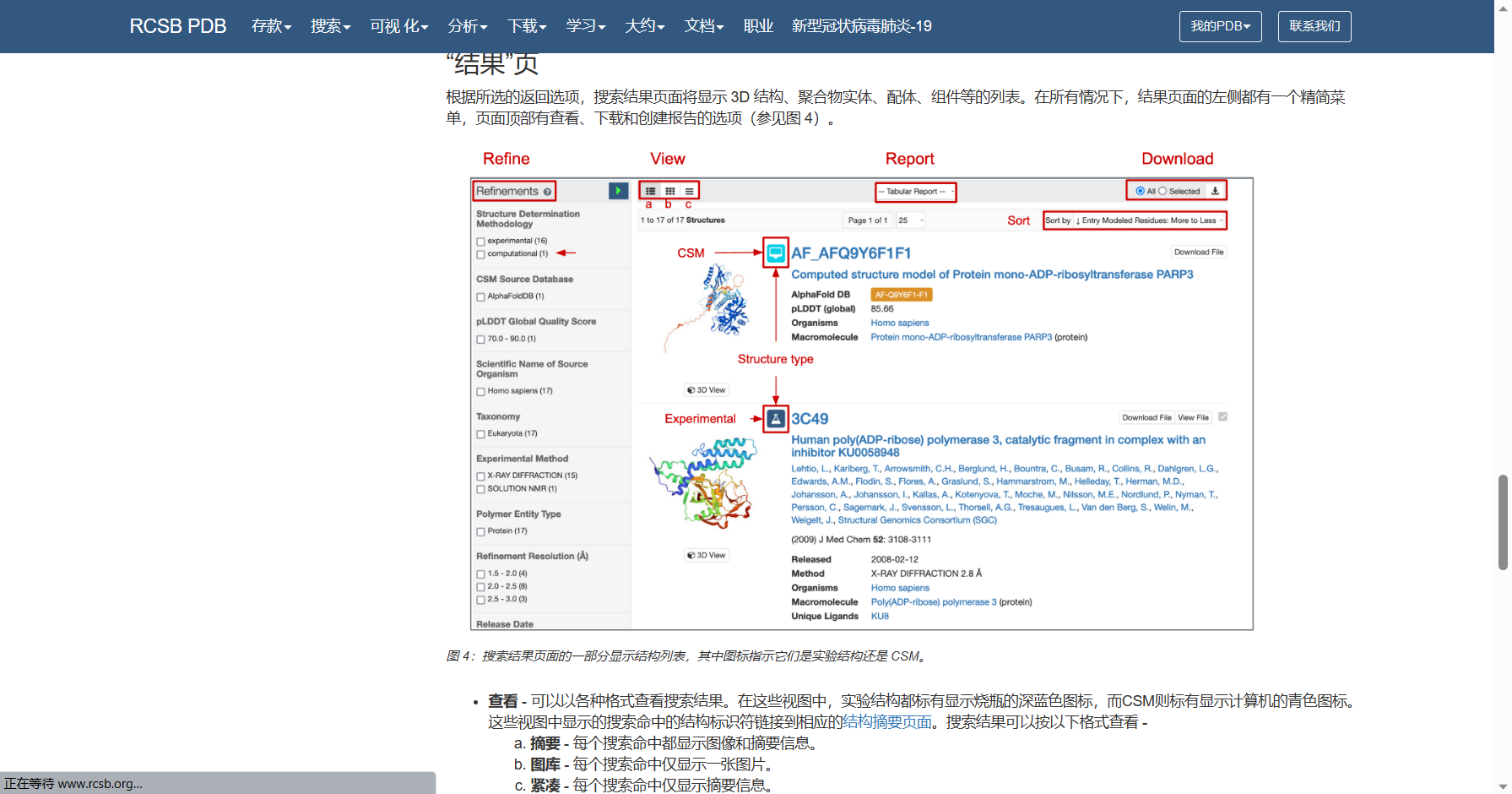
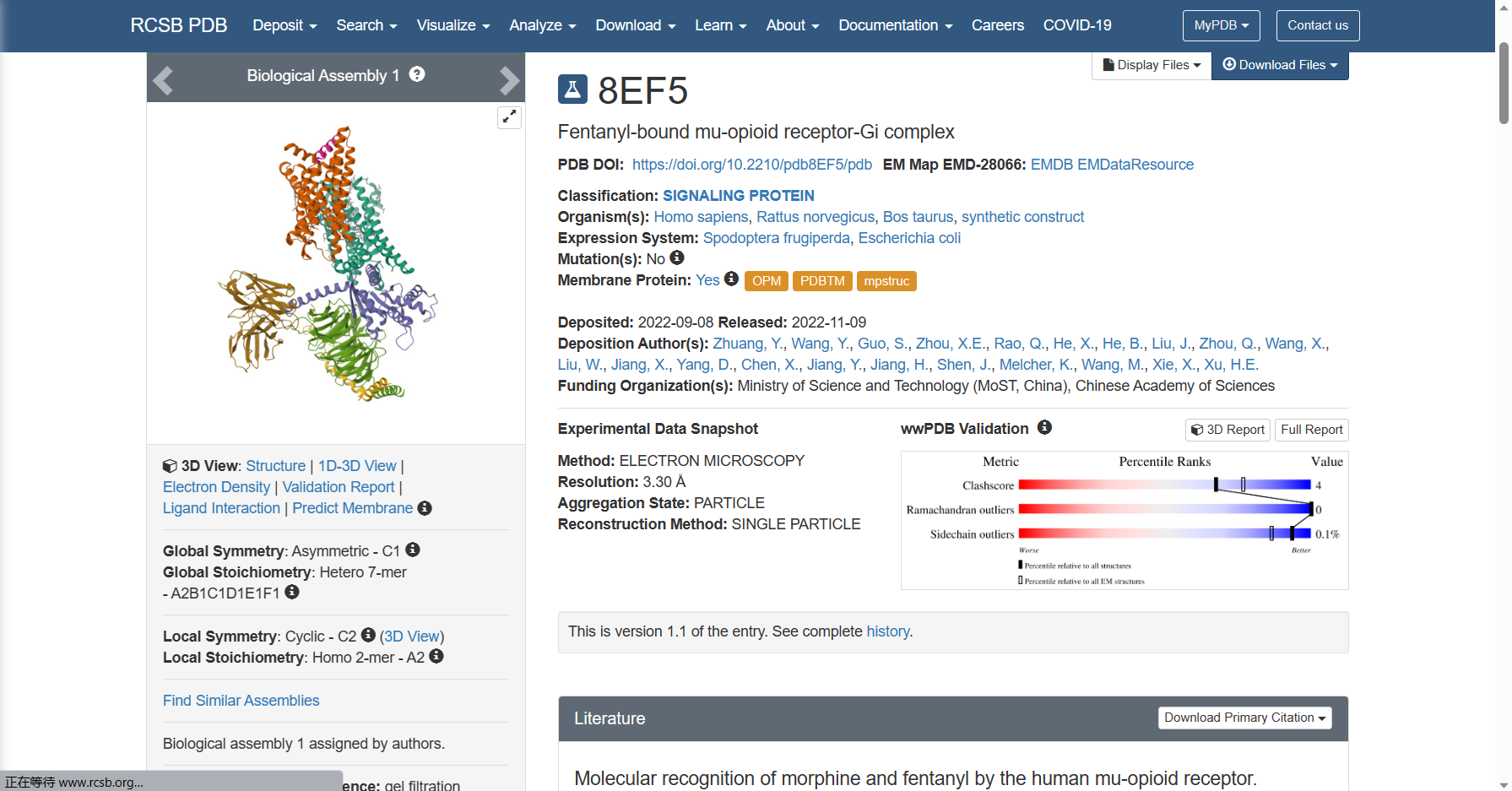
当我们正确搜索并从结果页左侧筛选出我们想查询的物质时,我们可以直接点进去查看它的各种信息并下载它的结构信息。需要注意的是,如果想导入到Discovery Studio中进行分析的话一般下载的是PDB 格式。(PDB gz为压缩后的PDB格式)
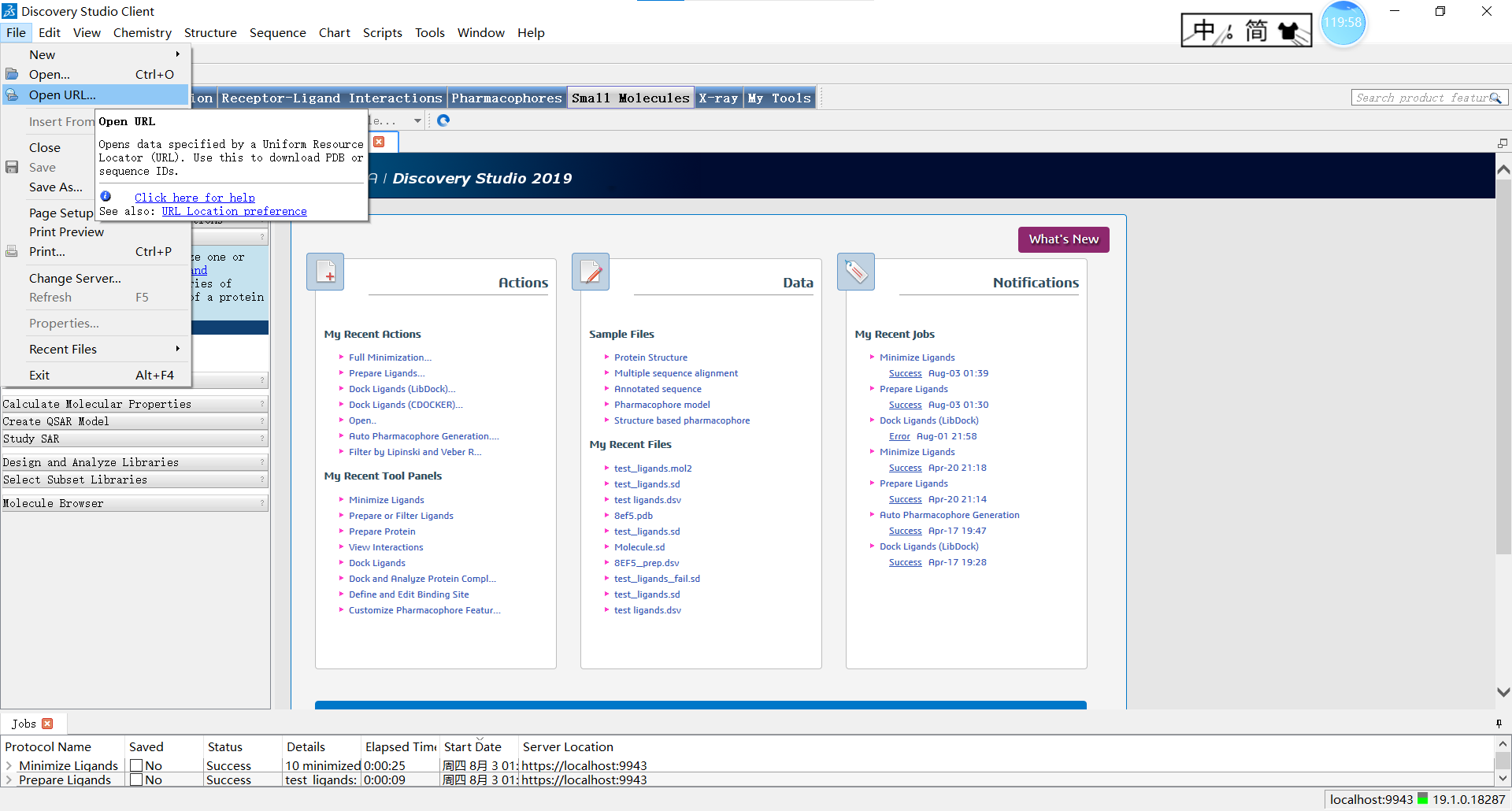
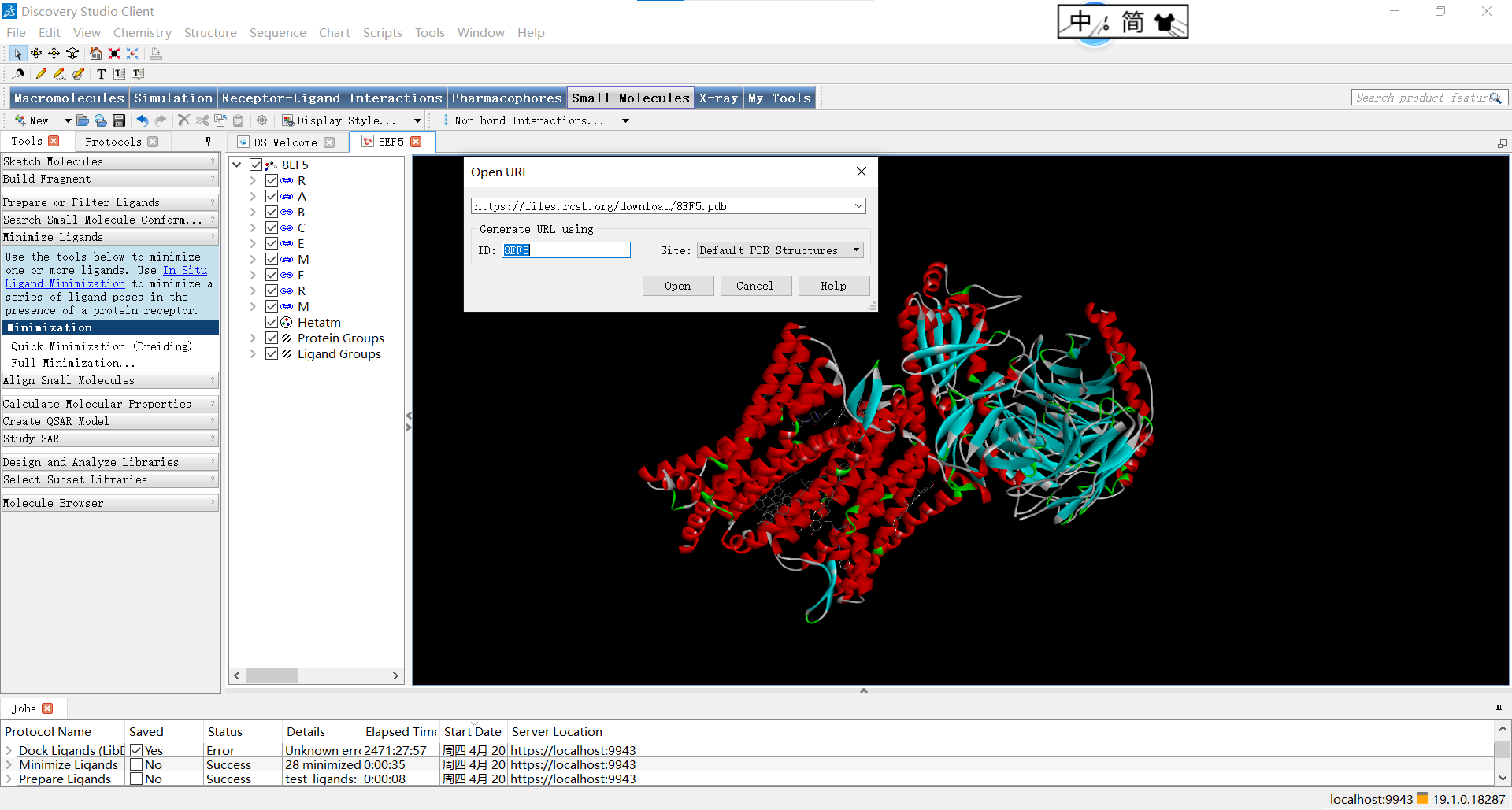
除了下载后从Discovery Studio打开文件这种方法,Discovery Studio还提供了通过PDB编号直接载入该结构的方法:点击菜单栏File->Open URL,随后输入编号等待一会即可打开。
如何对蛋白质结构优化?
优化蛋白质结构可能会有什么步骤呢,下面小果就给大家介绍一下:
- 构建蛋白质模型:针对目标蛋白质,可能需要根据已有的蛋白质结构数据、序列信息或相关模板进行蛋白质模型的构建。这有助于理解蛋白质的三维结构,并提供一个基础框架用于后续的计算和模拟。
- 蛋白质结构修复:对于蛋白质结构中存在的缺失、错误或不完整的部分,需要进行修复和优化。这包括修复缺失的原子、修正不合理的构象、修复结构错误等,以确保蛋白质的结构是准确和可靠的。
- 活性位点识别:活性位点是蛋白质上与配体结合并发挥生物活性的区域。通过对蛋白质结构进行分析和预测,可以识别出潜在的活性位点,这有助于在药物设计中选择和优化相互作用的区域。
- 水合剂处理:蛋白质在生物体内通常是水合状态的。在对蛋白质进行计算和模拟时,需要对水分子进行处理,包括添加、删除或优化水合剂的位置和构象。这有助于更准确地模拟蛋白质与其他分子的相互作用过程。
- 晶体结构处理:如果目标蛋白质的结构是通过X射线晶体学得到的,可能需要进行一些额外步骤来处理晶体结构,如去除溶剂分子、修复结晶中的原子错误等。
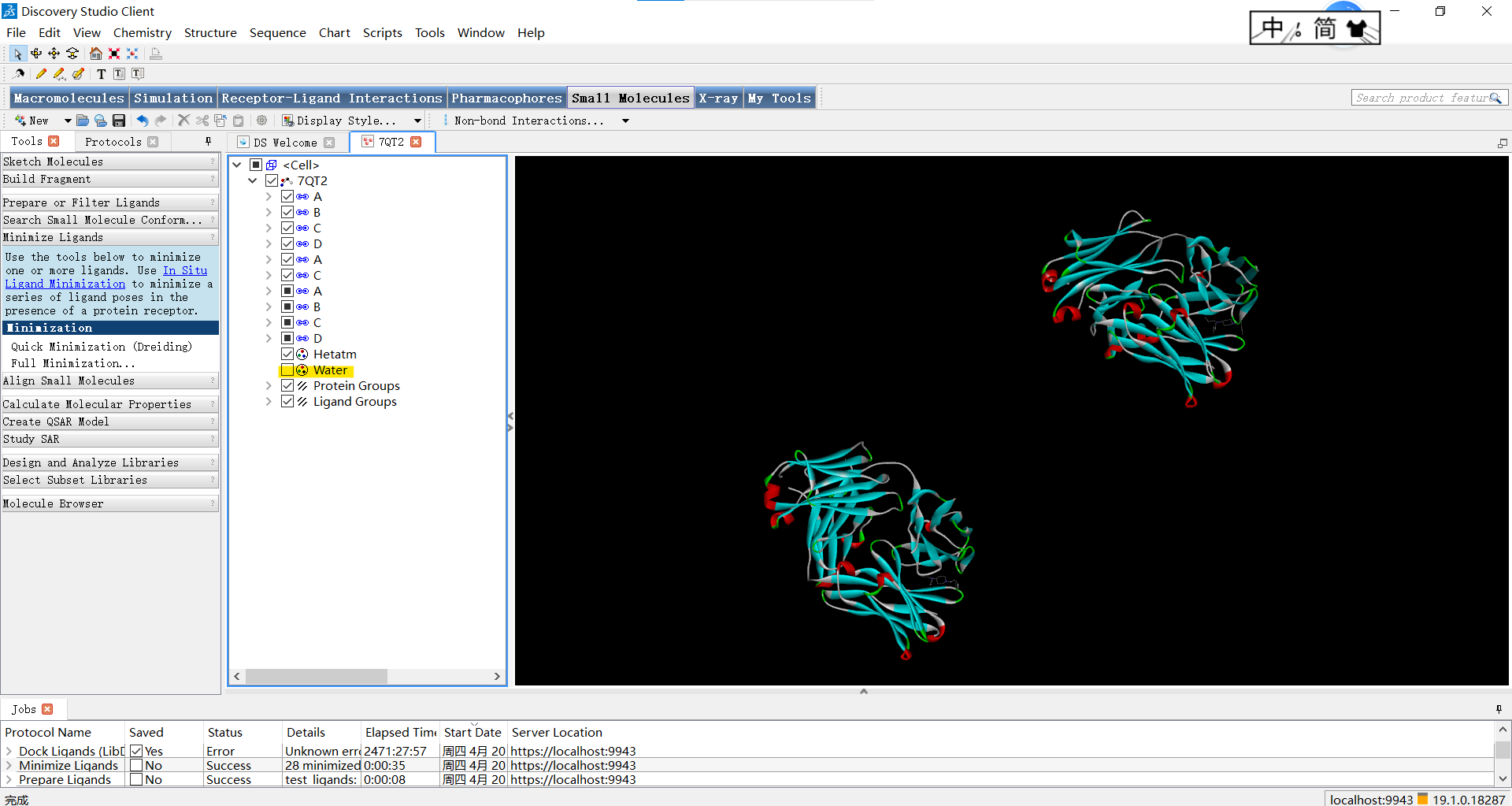
将蛋白结构导入Discovery Studio后,除非有特殊需求,一般首先将水分子去除(想去除哪一部分直接将勾选取消即可),因为8EF5中没有水分子,因此小果打开另一个分子给大家展示如何去除水分子(标黄部分)。(使用Ctrl H和Ctrl T可直接打开系统视图及表格视图)
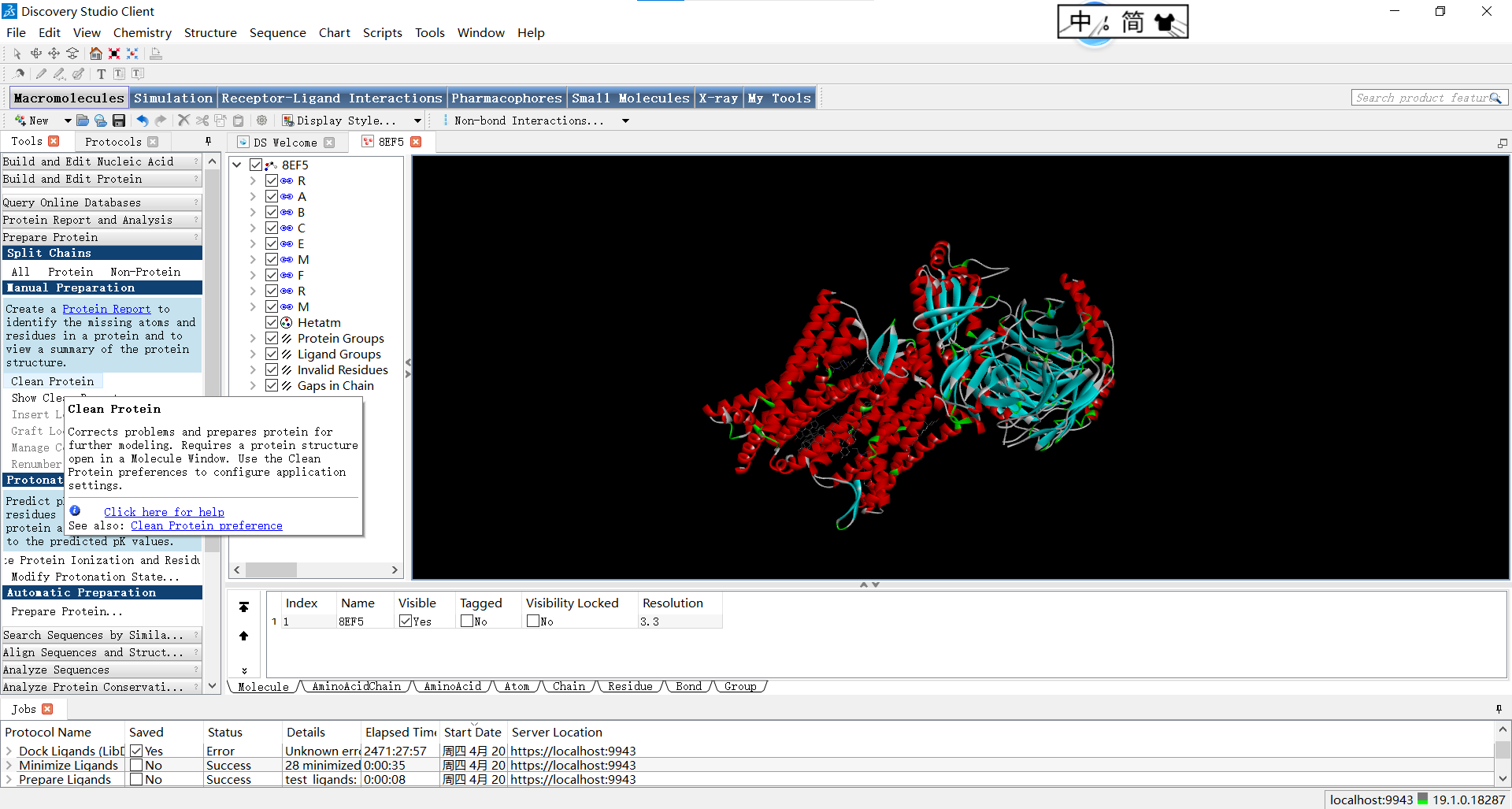
随后,我们一般使用Discovery Studio中快捷的Macromolecules>Prepare Protein>Clean Protein功能对蛋白质结构进行处理,即修复和优化蛋白质的原子坐标,以确保其符合合理的几何学约束,并提高后续计算和模拟的准确性。它执行以下主要功能:
- 去除非标准残基:例如水分子、离子和小分子配体。
- 校正氨基酸末端:修复氨基酸末端残基的缺失或不完整,确保所有的氨基酸残基都具有正确的连接和原子坐标。
- 修复剪切连接:剪切连接是由于实验或计算误差而导致的蛋白质链断开的情况,该功能能够自动检测和修复这些断开的连接。
- 优化原子坐标:该工具使用分子力学力场对蛋白质的原子坐标进行最小化或优化,以提高其几何学质量。这可以通过克服原子间的过度重叠、修正扭曲构象和优化氢键等方式来实现。
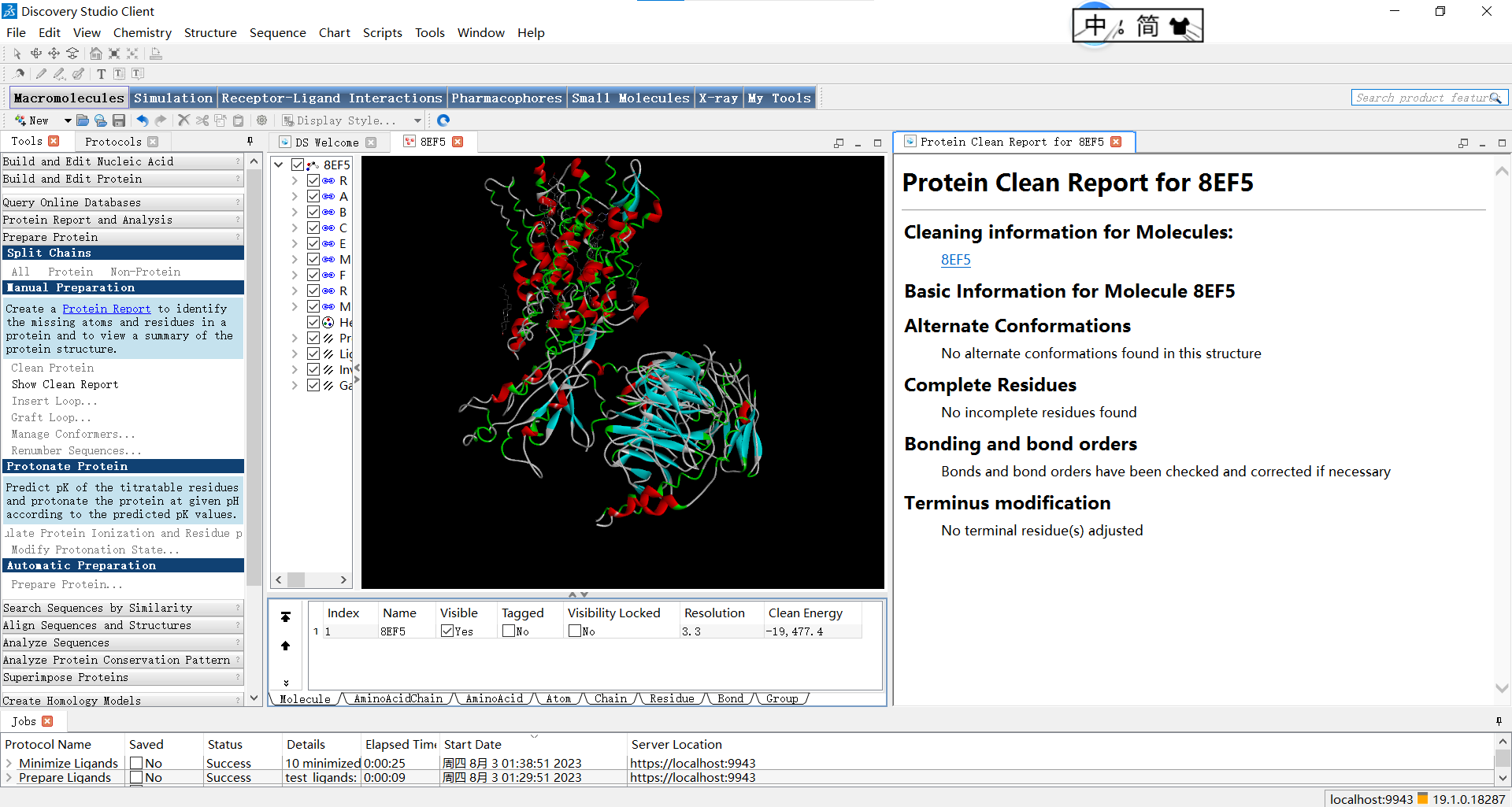
在Clean Protein按钮下面有显示相应报告的按钮:
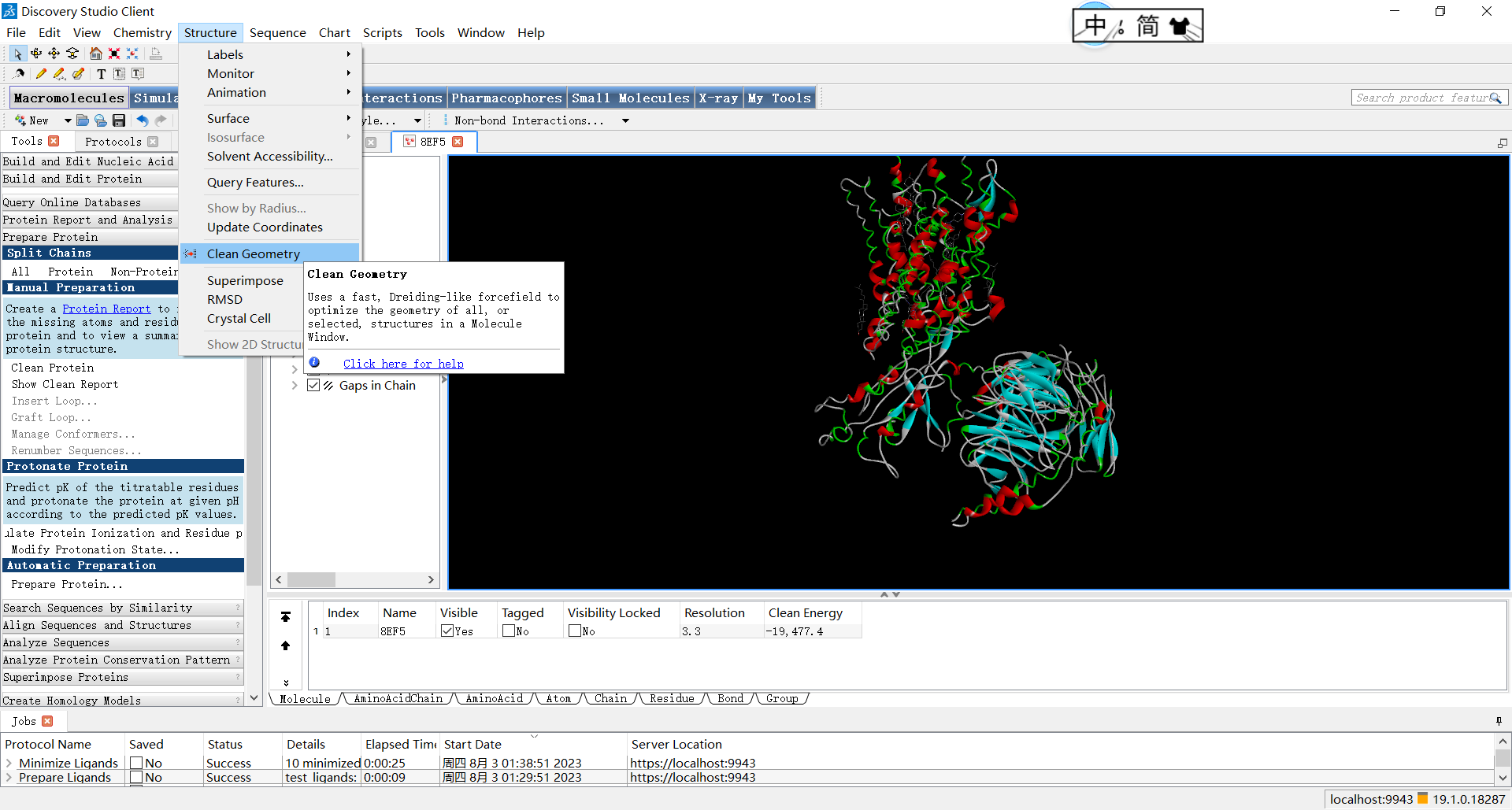
除了最便捷最常用的Clean Protein功能外,Discovery Studio还提供了非常多的其他功能来优化蛋白结构,例如主菜单栏structure->clean geometry,它可以清除蛋白质结构中的几何错误或不良几何特征,修复蛋白质结构中的构象问题,例如不正常的键长、键角或扭转角。
通过右键Macromolecules的tools时弹出的菜单我们可以很直观地感受到Discovery Studio的功能之强大和丰富:
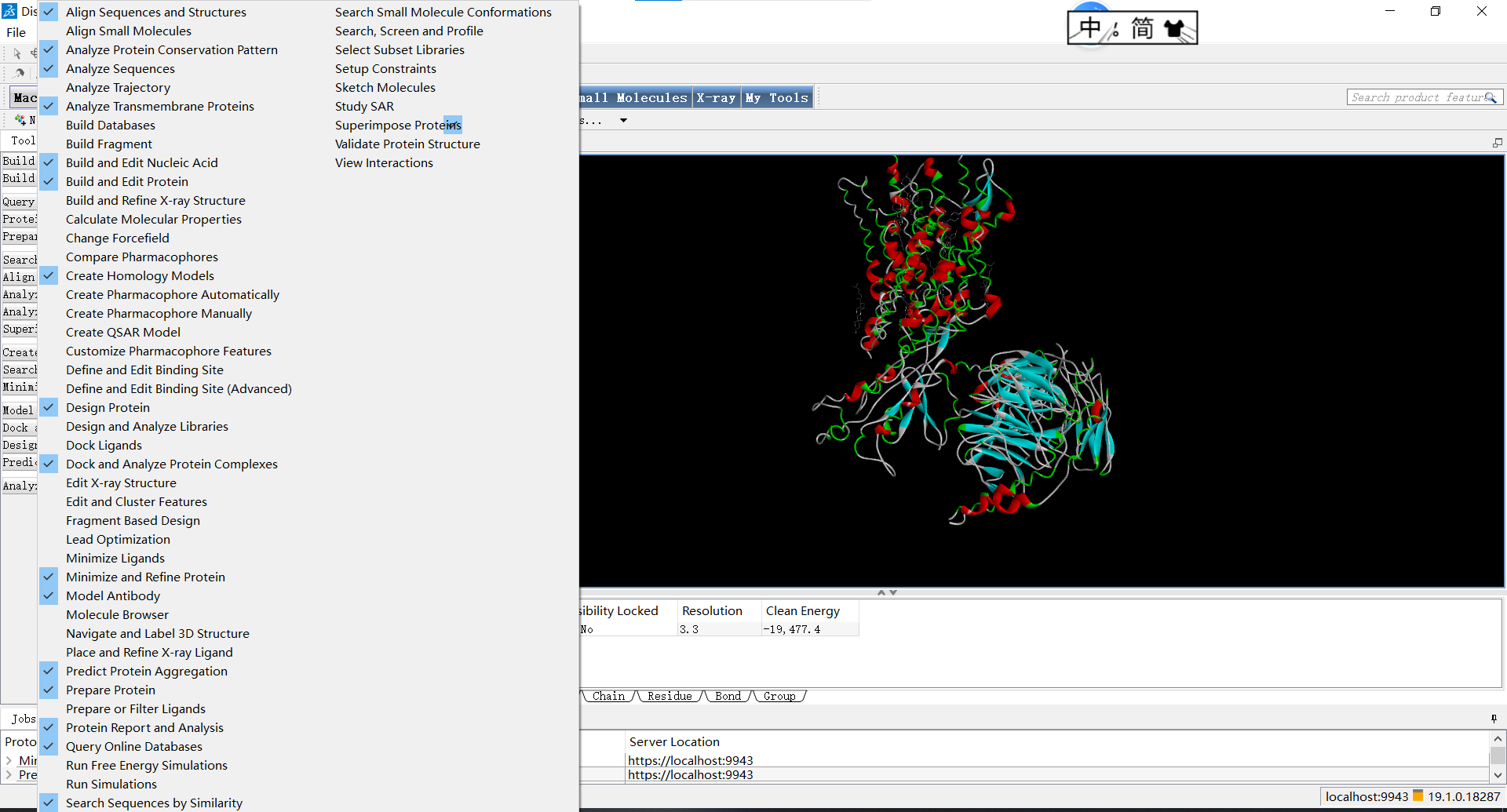
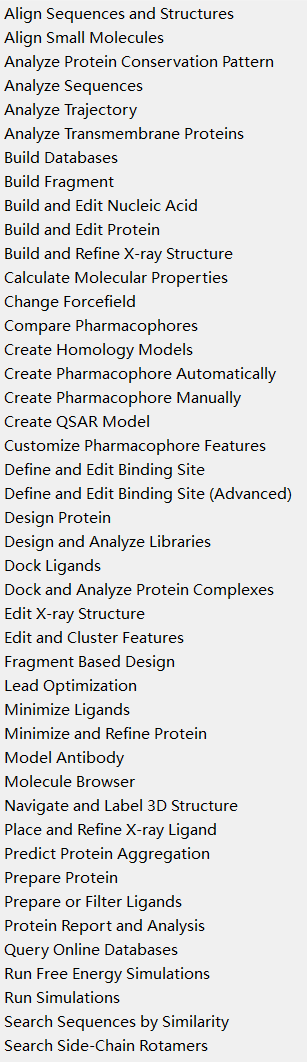
小果在这里给大家翻译成中文,让大家看着更加方便:
对小分子进行对齐;
分析蛋白质保守性模式;
分析序列;
分析轨迹;
分析跨膜蛋白;
构建数据库;
构建片段;
构建和编辑核酸;
构建和编辑蛋白质;
构建和改进X射线结构;
计算分子性质;
更改力场;
比较药效团;
创建同源模型;
自动生成药效团;
手动创建药效团;
创建QSAR模型;
自定义药效团特征;
定义和编辑结合位点;
定义和编辑结合位点(高级);
设计蛋白质;
设计和分析库;
对配体进行对接;
对接和分析蛋白质复合物;
编辑X射线结构;
编辑和聚类特征;
基于片段的设计;
引导优化;
最小化配体;
最小化和改进蛋白质;
建模抗体;
分子浏览器;
浏览和标记3D结构;
放置和改进X射线配体;
预测蛋白质聚集;
准备蛋白质;
准备或筛选配体;
蛋白质报告和分析;
查询在线数据库;
运行自由能模拟;
运行模拟;
通过相似性搜索序列;
搜索侧链Rotamer。
上面有一些名词小伙伴可能不太熟悉,小果在这里给大家稍微解释一下,比如:
- QSAR模型代表定量构效关系(Quantitative Structure-Activity Relationship),是一种用于预测化合物活性和性质的计算模型。它建立了化合物结构与其生物活性之间的定量关系,通过分析和量化分子结构的化学特征,预测其活性或其他相关性质。
- 药效团(Pharmacophore)是指在药物分子中对特定生物活性起关键作用的功能基团或结构模式。它是通过分析和总结一系列具有相似生物活性的化合物,确定出对活性贡献最显著的共同特征。
- 侧链Rotamer是指在蛋白质结构中,氨基酸侧链的不同构象或旋转状态。每个氨基酸侧链都可以采取多种不同的构象,这些构象之间的旋转和空间排布存在差异。侧链Rotamer描述了侧链的不同构象,并通过命名和分类不同的旋转状态来表示。通过搜索侧链Rotamer,可以找到合适的蛋白质侧链构象,用于蛋白质的模拟、设计和分析等应用。
总而言之,Discovery Studio是一款功能强大且丰富的软件,小果在这里是跟大家介绍不完的,想要深入学习的小伙伴们可以自己查看help文档并亲自多尝试尝试,毕竟实践才能出真知。本次的分享就到这里啦,如果小伙伴们平时在生信分析的操作过程中遇到困难,欢迎大家使用小果开发的生信工具平台http://www.biocloudservice.com/home.html哦。
那么我们下次再见啦,期待与小伙伴们继续共同成长哦,拜拜┏(^0^)┛~