ggpairs()简介
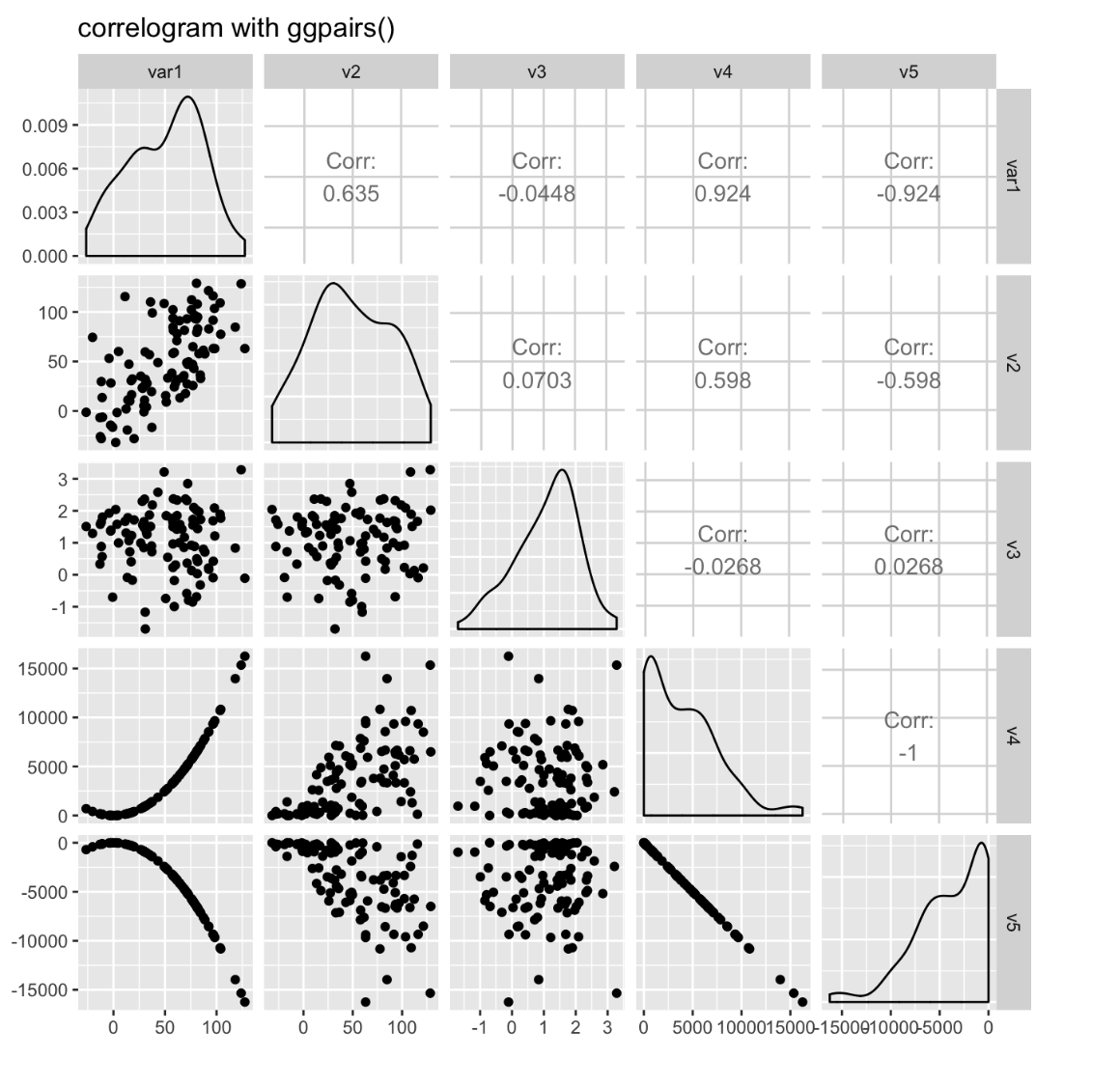
在学习ggpairs之前我们可以先来欣赏一下这个包绘制的图形如下,
每对数值变量的散点图绘制在图的左侧。Pearson相关性显示在右侧。对角线上有可变分布。
小试一下
# 设置工作路径
setwd(“”)
# 加载R包
install.packages(“GGally”)
library(GGally)
# 构建数据集
data <- data.frame( var1 = 1:100 + rnorm(100,sd=20), v2 = 1:100 + rnorm(100,sd=27), v3 = rep(1, 100) + rnorm(100, sd = 1))
data$v4 = data$var1 ** 2
data$v5 = -(data$var1 ** 2)
#这里小果为了让数据具有相关性,分别生成了5列具有不同类型的相关,这里可以导入自己数据,l例如以下代码为读入相关文件的操作。
data <read.csv(“test.xls”,header = TRUE,row.names = 1)
# 用包绘制基础的相关性(如散点图),分布状态和打印相关系数
ggpairs(data, title=”correlogram with ggpairs()”)
#这里的
函数详解
在ggpairs()函数中,还可以使用许多其他参数来自定义生成的图形,例如:
data: 要分析的数据集,通常是一个数据框或数据框的子集。必需参数。
columns: 一个字符向量,指定要在矩阵图中考虑的变量。默认情况下,将考虑所有的变量。例如,columns = c(“var1”, “var2”, “var3”)。
mapping: 一个aes()映射,用于指定额外的映射参数,例如颜色或形状。例如,mapping = aes(color = species)。
lower: 指定下三角矩阵中要显示的图表类型。可以是NULL(无图表)、points(散点图)、smooth(散点图和平滑曲线)、density(核密度图)或自定义函数。默认为points。
upper: 指定上三角矩阵中要显示的图表类型。可以是NULL(无图表)、cor(相关性系数)或自定义函数。默认为cor。
diag: 指定对角线上的图表类型。可以是NULL(无图表)、density(核密度图)、barDiag(条形图)或自定义函数。默认为density。
legend: 一个逻辑值,用于指定是否显示图例。默认为TRUE。
ggplot2_args: 一个列表,包含传递给底层ggplot2图形的其他参数。
可视化结果:ggpairs()函数将生成一个矩阵图,显示数据集中变量之间的散点图和相关性图。该图具有以下特征:
对角线上的图表:默认情况下,对角线上的图表显示了各个变量的直方图。这使您可以了解每个变量的分布情况。
下三角矩阵图表:下三角矩阵中的每个图表显示了两个变量之间的散点图和一些相关性信息,例如相关系数和回归线。
上三角矩阵图表:上三角矩阵中的每个图表显示了两个变量之间的相关性矩阵。
高级绘图
#更改相应的变量名称
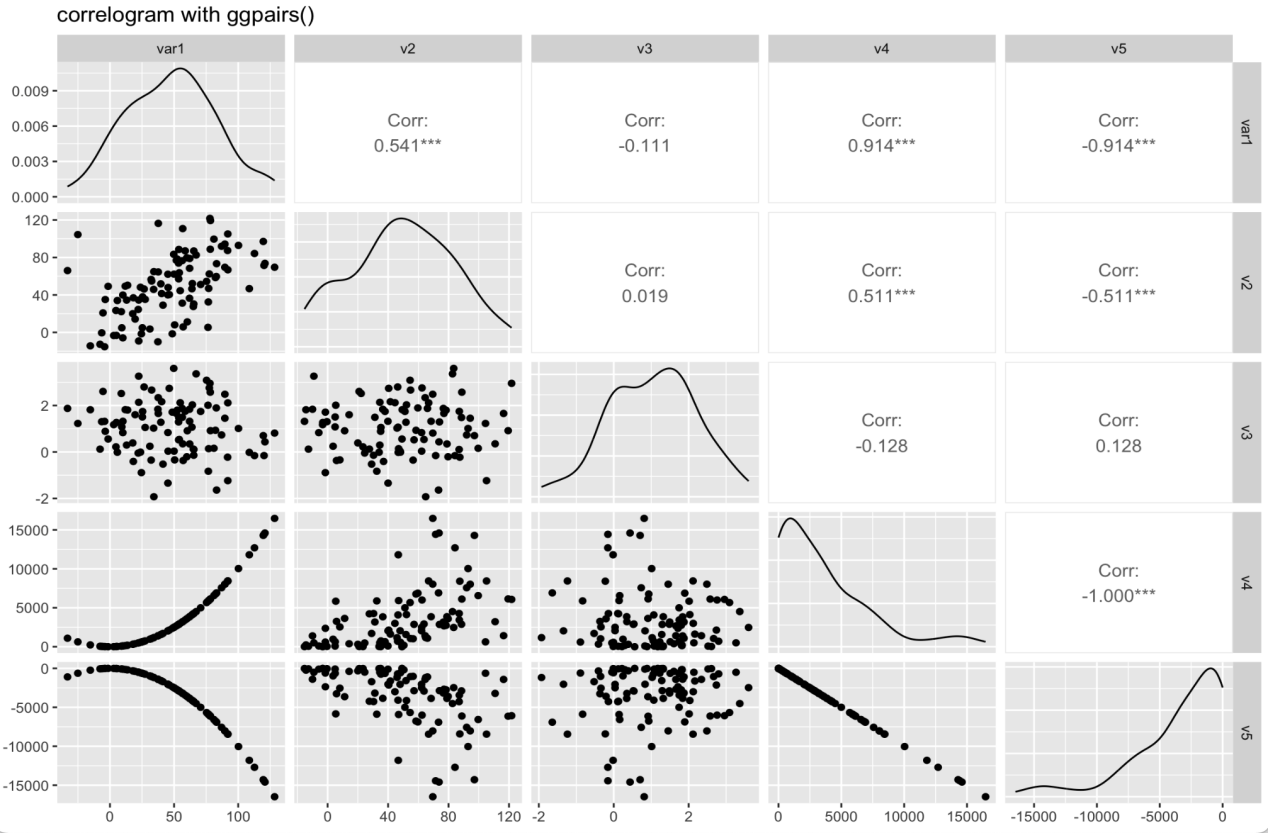
ggpairs(data, columnLabels = c(“var1″,”var2″,”var3″,”var4″,”var5”))
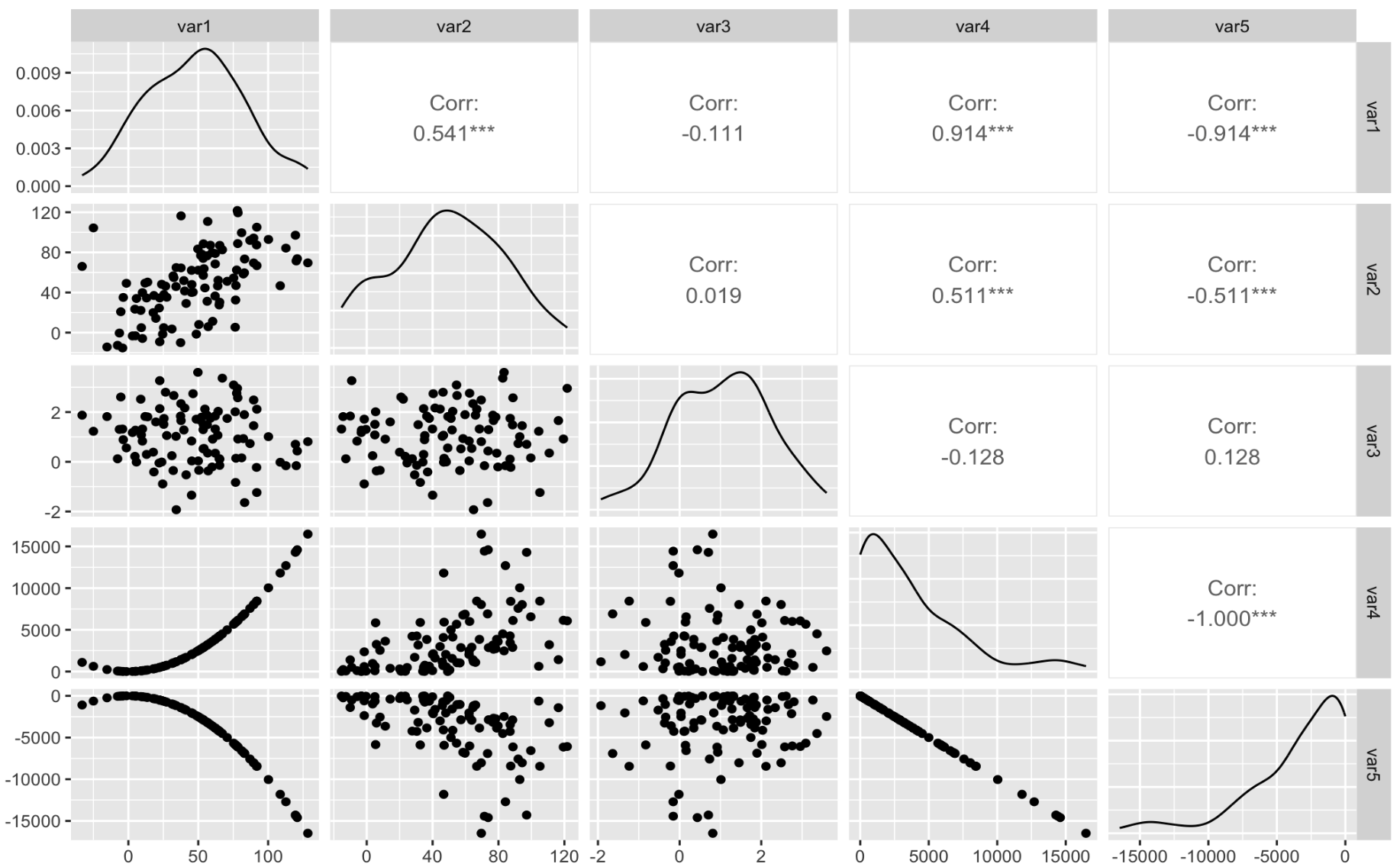
#指定对角线、上三角区和下三角区图形的类型
ggpairs(data,columns = 1:5, # 选择需要绘图的变量所在列号
columnLabels = c(“var1″,”var2″,”var3″,”var4″,”var5”),
#upper = “black” # 不显示upper部分
upper = list(continuous = “cor”, # 为各种数据类型设置显示形式,ggally中功能函数的后缀(ggally_cor())。
#combo = “box_no_facet”, # 设置combo数据类型的绘图参数
#discrete = “count”,# 设置discrete数据类型的绘图参数。
na =”na”),# 设置参数名称与成对数据类型名称一致。
lower = list(continuous = “smooth”,#ggally_smooth()
#combo = “facethist”,
#discrete = “facetbar”,
na =”na”),
diag = list(continuous = “densityDiag”, # ‘densityDiag’, ‘barDiag’, ‘blankDiag’可选
#discrete = “barDiag”, # ‘barDiag’, ‘blankDiag’可选
#diag =NULL # 不显示diag部分
na = “naDiag”))+
theme_bw()# 设置主题参数
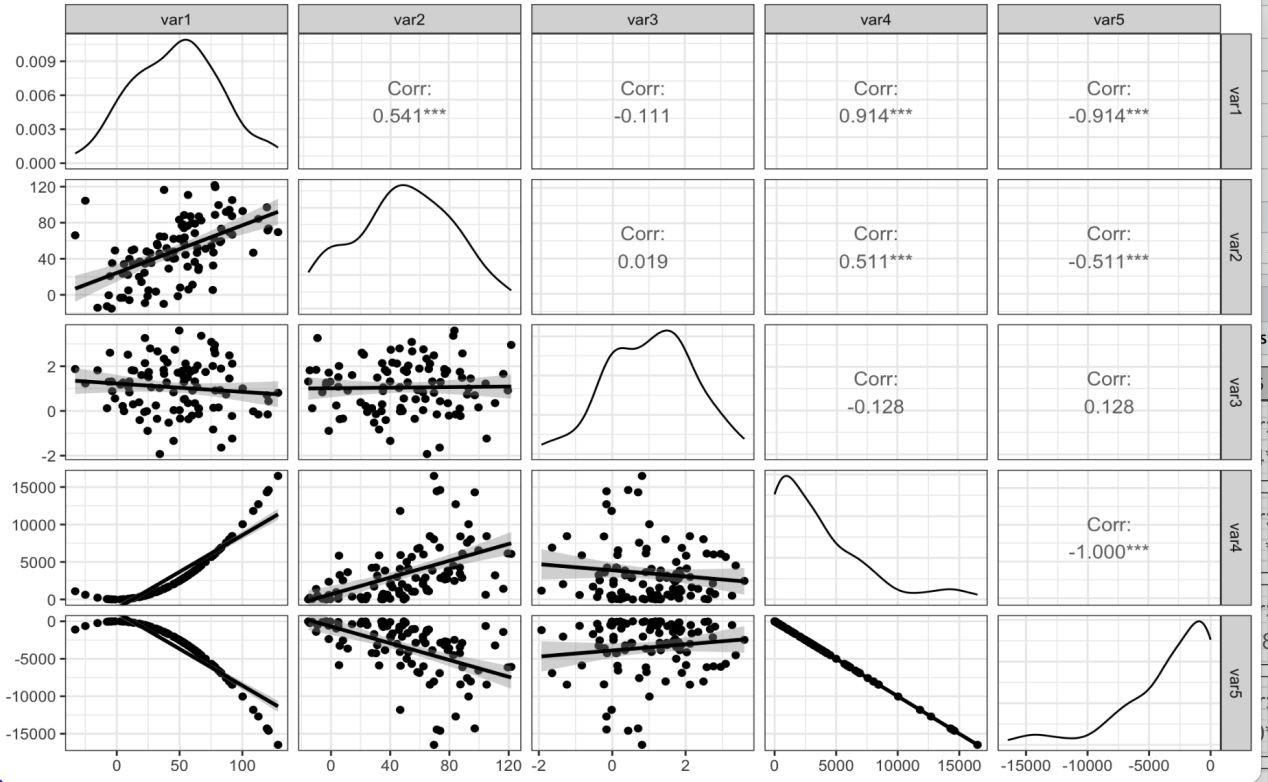
#根据分组来绘制相关性
data(flea)
ggpairs(flea, columns = 2:4, ggplot2::aes(colour=species))
#这是flea数据格式

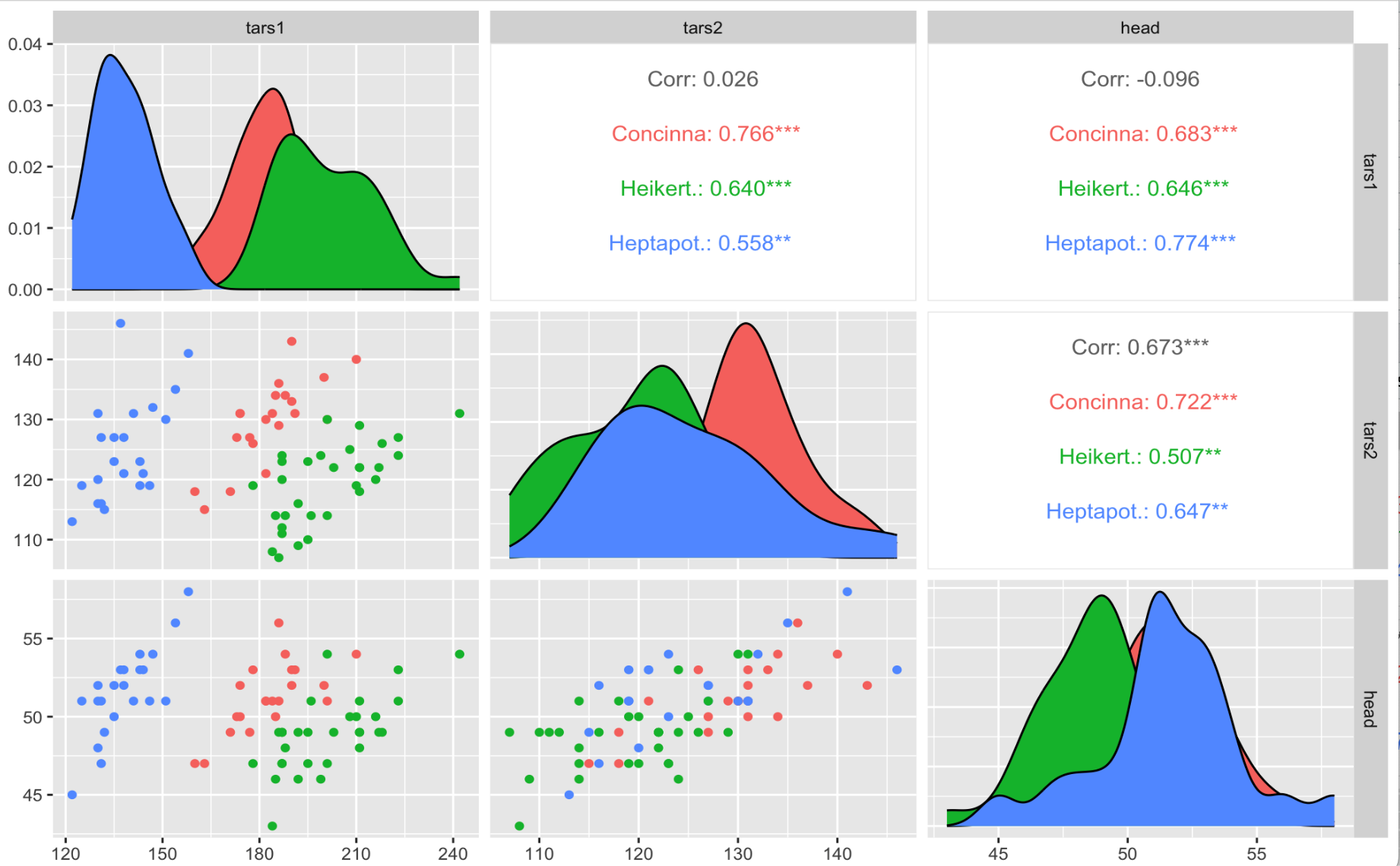
#使用wrap()设置ggally功能函数的参数,以调整绘图参数
ggpairs(flea,columns = 2:4,
ggplot2::aes(color = species), # 按species分组信息分别着色、计算相关性系数和拟合
upper = list(continuous = wrap(“cor”,method = “spearman”)), # 更改相关性系数,?ggally_cor,查看函数参数,默认pearson。
lower = list(continuous = wrap(“smooth”,method = “loess”,se = TRUE)), # 默认lm
diag = list(continuous = wrap(“densityDiag”,rescale=TRUE)))+ # 默认rescale=FLASE,此参数只对数值型数据起效。
theme_bw()
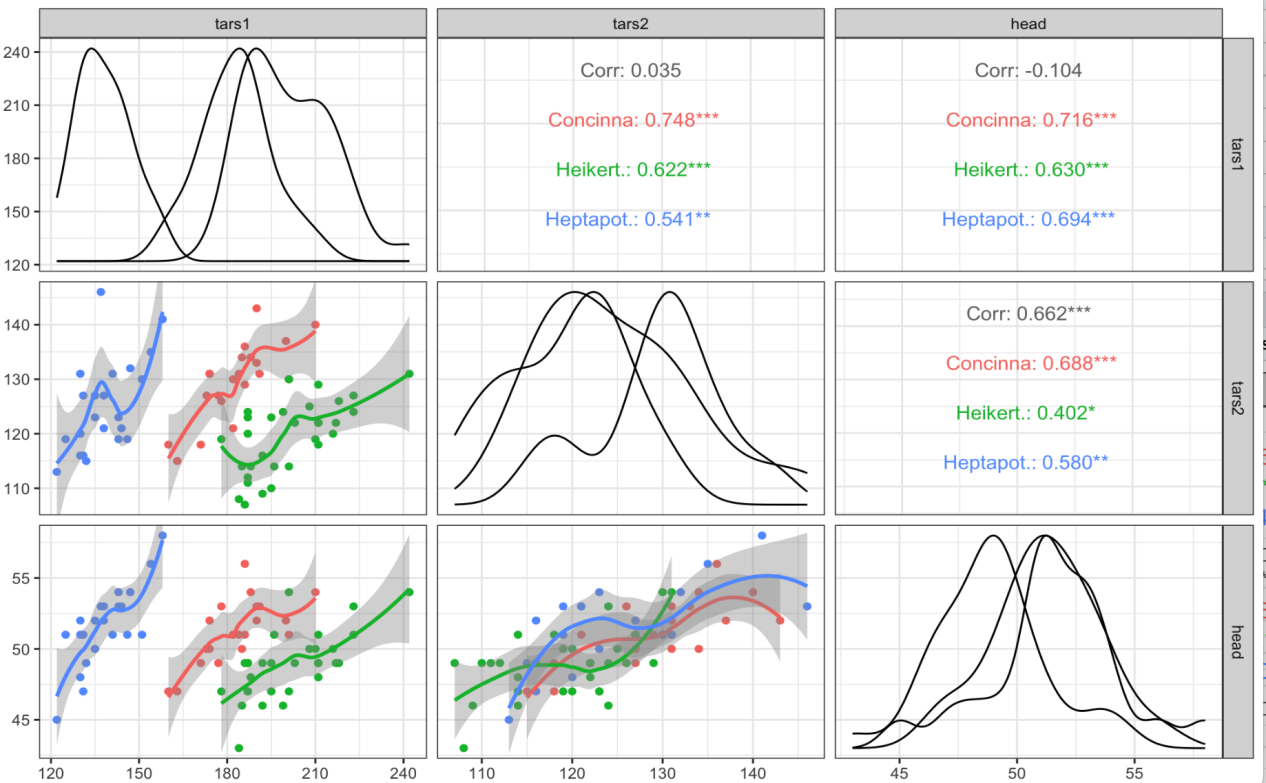
#自定义函数以调整绘图参数
my_density <- function(data, mapping, values = cols ) {
ggplot(data = data, mapping = mapping) +
geom_density() +
scale_color_manual(values = values)
} # 自定义函数
library(ggsci)
cols = pal_jco()(7) # 设置绘图颜色
ggpairs(flea,columns = 2:4,
ggplot2::aes(color = species), # 按分组信息分别着色、计算相关性系数和拟合
upper = list(continuous = “cor”),
lower = list(continuous = wrap(“smooth”,method = “loess”,se = FALSE)), # se = FALSE,不展示置信区间
diag = list(continuous = my_density))+
ggplot2::scale_color_manual(values = cols)+
theme_bw()
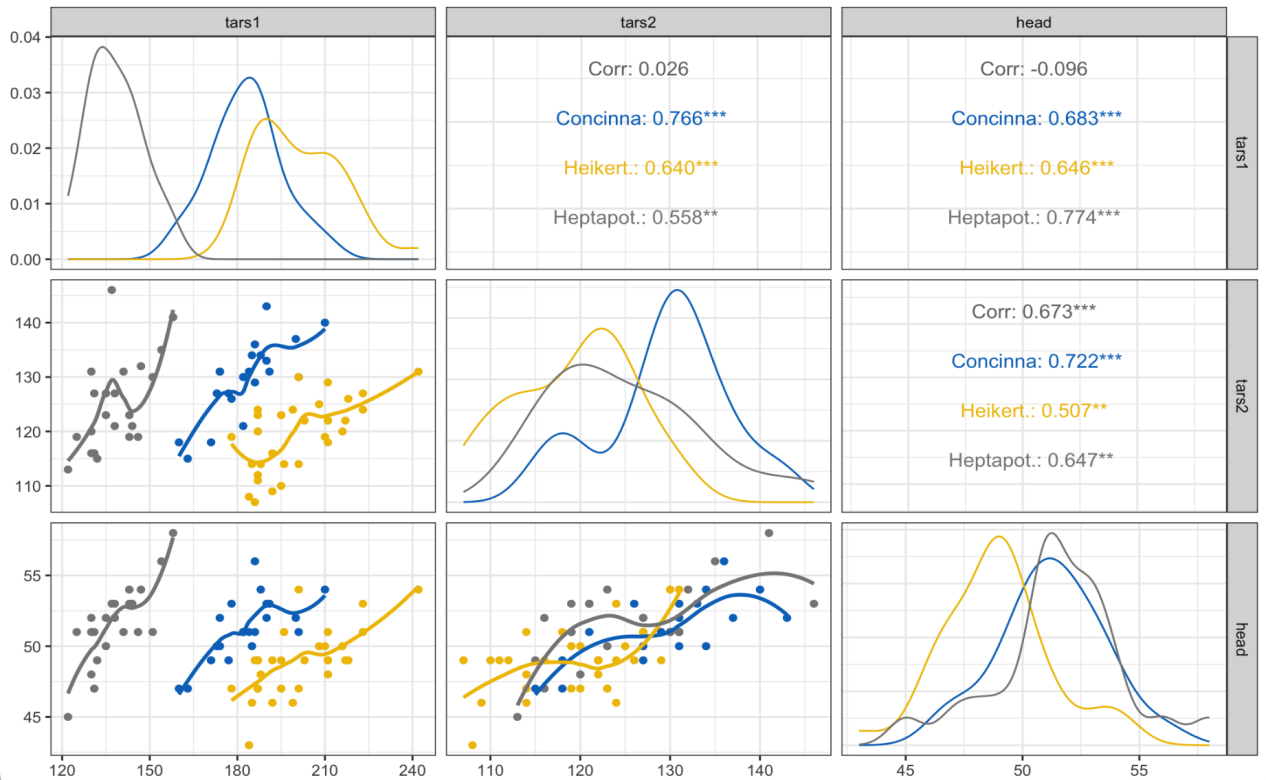
通过以上小果的介绍,相信你对这个ggpairs()函数已经有了一定的了解,想要画出好看的相关矩阵,需要你用心不断的调整相应的参数。小果相信,你在联系果以上代码后,可以得到自己想要的图。
如果遇到不懂的也可以借助线上的云平台哦~http://www.biocloudservice.com/home.html
好了今天的ggpairs就讲到这里,欢迎大家有问题与小果一起讨论哦~