你知道吗,有两个R包实现了t-SNE(t-distributed stochastic neighbor embedding)降维算法以及UMAP (Uniform Manifold Approximation and Projection )降维技术。今天小果就来为大家详细介绍一下这两个R包的用途并来教大家如何具体使用它,感兴趣的话就和小果一起看下去吧!
了解Rtsne包和umap包
Rtsne
Rtsne是一个在R语言中实现了t-SNE(t-Distributed Stochastic Neighbor Embedding)算法的包。t-SNE是一种非线性降维算法,用于将高维数据映射到二维或三维空间,以便进行可视化和聚类分析。
Rtsne包提供了一个名为Rtsne()的函数,可以使用t-SNE算法将高维数据降低到较低维度。该函数的常用参数包括:
- X:输入数据矩阵,包含要降维的特征。
- dims:输出降维后的维度数,通常为2或3。
- pca:一个逻辑值,指示是否在运行t-SNE之前对数据进行PCA降维。默认为TRUE,表示在进行t-SNE之前进行PCA降维。
- perplexity:控制t-SNE算法的困惑度(perplexity)参数,用于衡量每个点周围邻域的重要性。较大的值会产生更远的邻域效果。默认值为30。
- theta:控制t-SNE算法的角度参数,用于平衡近邻点之间的准确度和计算效率。较小的值会产生更精确的结果,但计算时间可能较长。默认值为0。
- initial_dims:在进行PCA降维之前,输入数据矩阵的初始维度数。默认值为50。
除了降维函数Rtsne(),该包还返回其他有用的信息,如转换后的数据、降维后的迭代次数以及最终代价(cost)值等。
通过使用Rtsne包,您可以轻松地在R语言中实现t-SNE算法,并将高维数据可视化,以便更好地理解数据的特征和聚类模式。
umap
umap(Uniform Manifold Approximation and Projection)是一个在R语言中实现了UMAP算法的包。UMAP是一种非线性降维算法,用于将高维数据映射到较低维度的空间,同样用于可视化和聚类分析。
umap包提供了一个名为umap()的函数,可以使用UMAP算法对高维数据进行降维。该函数的常用参数包括:
- X:输入数据矩阵,包含要降维的特征。
- n_neighbors:每个点的近邻数。默认值为15。
- n_components:输出降维后的维度数,通常为2或3。
- metric:用于计算距离的度量方法,默认为欧几里得距离。您还可以选择其他度量方法,如曼哈顿距离、余弦距离等。
- min_dist:控制低维空间中邻域之间的最小距离。较小的值会产生更密集的嵌入效果,而较大的值会产生更分散的嵌入效果。默认值为0.1。
除了降维函数umap(),该包还提供了其他有用的函数和工具,如可视化函数plot(),用于对降维结果进行可视化。
通过使用umap包,您可以轻松地在R语言中实现UMAP算法,将高维数据降维到较低维度,并进行可视化和聚类分析,以便更好地理解数据的结构和特征。
使用Rtsne包进行数据降维
了解完这个R包的基本用途后,进入我们今天的正题!
我们将学习如何使用Rtsne和umap这两个R语言包来降维和可视化“iris”数据集。通过降维和可视化,我们可以更好地理解iris数据的特征并发现其聚类模式。
下载并导入R包
首先,我们需要导入所需的R包,包括Rtsne、ggplot2和umap。
library(Rtsne)
library(ggplot2)
library(umap)
加载数据集
接下来,我们将加载R自带的iris数据集 。该数据集是一个经典的机器学习数据集,其中包含150个样本和4个特征。我们可以使用以下代码加载数据集:
iris_data <- unique(iris)
小果温馨提示,在这里我们使用了unique()函数来去除重复值,以确保每个样本只出现一次哦。
Rtsne进行数据降维
接下来,我们使用Rtsne来进行降维。Rtsne是一个用于t-SNE降维的R包,刚才小果也对其做了简单地介绍,我们现在来一起实际操作一下吧!我们可以使用以下代码进行降维:
tsne <- Rtsne(iris_data[,-5], pca=FALSE, theta=0)
在这里,我们将去除最后一列(数据的种类)作为输入,设置pca=FALSE以使用t-SNE算法而不是PCA算法进行降维,设置theta=0以使用默认的角度。
现在,我们通过以下代码查看降维后的数据:
names(tsne)
运行上述代码后,你将看到一系列返回值,其中包括N、Y、costs等。这些返回值包含了降维后的信息。

降维结果可视化
下一步,我们可以使用ggplot2包来可视化降维后的数据。使用以下代码:
as_tibble(tsne$Y) %>%
bind_cols(Species=iris_data$Species) %>%
mutate(t1=V1,t2=V2) %>%
ggplot(aes(t1, t2, col=Species, shape=Species)) +
geom_point(size=3)
上述代码将降维后的数据转换为tibble格式,并添加数据的种类标签。然后,我们使用ggplot2来绘制散点图,其中x轴表示t1,y轴表示t2,点的颜色和形状表示数据的种类。我们一起来看看可视化后的效果图是什么样吧!
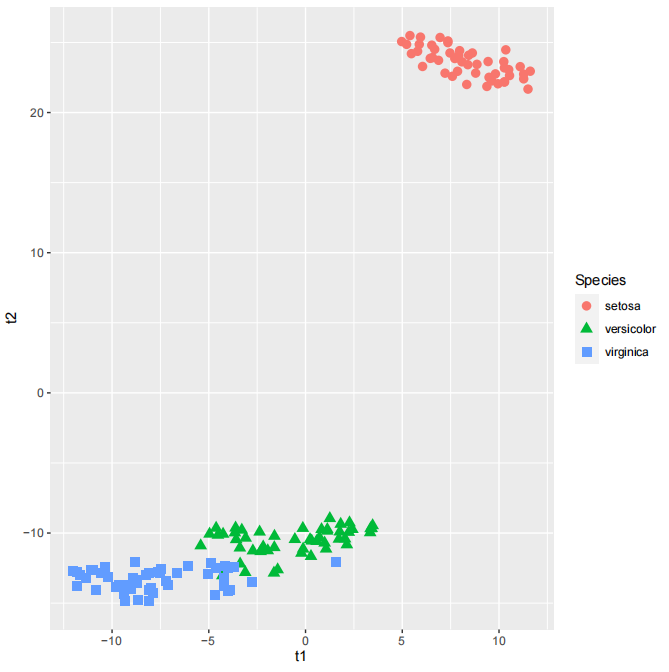
umap数据降维
第二个方法我们使用umap来进行降维和可视化。umap是一个用于UMAP降维的R包,我们一起来看看吧!
使用以下代码进行降维:
iris_umap <- umap(iris[,-5])
在这里,我们将去除最后一列作为输入。
现在,我们可以通过以下代码提取降维后的数据:
layout <- iris_umap$layout
结果如下:
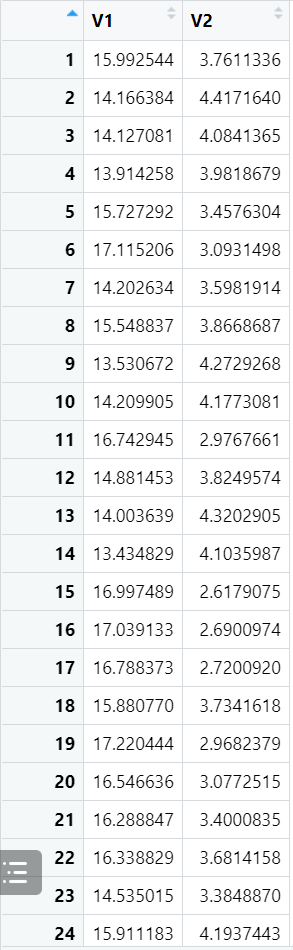
降维结果可视化
接下来,我们仍然选择使用ggplot2来可视化降维后的数据。使用以下代码即可完成:
layout %>%
as_tibble() %>%
ggplot(aes(V1, V2, col=iris$Species, shape=iris$Species)) +
geom_point(size=2)
上述代码将降维后的数据转换为tibble格式,并使用ggplot2绘制散点图,其中x轴表示V1,y轴表示V2,点的颜色和形状表示数据的种类。我们一起来看看可视化的结果图:
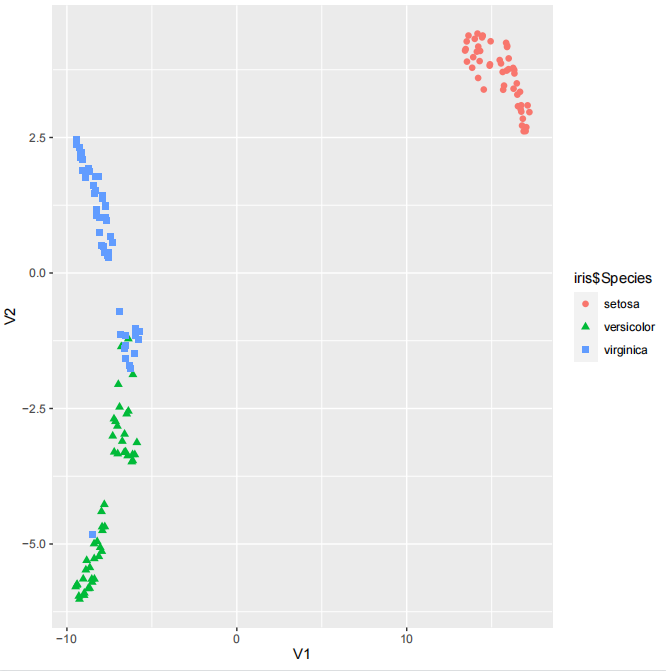
以上就是使用Rtsne和umap包对示例数据集进行降维和可视化的详细教程。怎么样,是不是很简单!你学会了嘛?希望能对你的分析有所帮助!更多学习干货敬请关注小果吧!