R语言包Pathview是一个用于生物学数据可视化和分析的工具,主要用于在代谢通路和信号通路等方面的研究中可视化基因组学数据。Pathview通过生成直观的图形展示,帮助研究人员更好地理解大规模生物学数据的内在模式和关系,从而促进对生物学过程的深入认识。
在生物学研究中,高通量技术如基因组测序和蛋白质质谱等产生了大量的数据,这些数据通常包含着复杂的信息。Pathview的核心功能之一是将这些数据与生物通路图集成,从而揭示不同通路中的差异表达基因或蛋白质的变化情况。这种集成能够帮助研究人员直观地观察到在不同条件下通路活性的变化,有助于发现与特定生物学过程相关的关键基因或蛋白质。
要使用pathview包,可以在R中使用以下命令进行安装和加载:
> install.packages(“pathview”) #安装pathview语言包
> library(pathview) #加载语言包
Pathview不仅提供了预定义的通路图,还支持用户上传自定义的通路图,以满足各种研究需求。用户可以根据自己的数据和问题,选择合适的通路图进行分析。Pathview会自动将基因或蛋白质的表达数据映射到通路图上的相应位置,生成可交互的图形展示。这种交互性使研究人员能够深入探索数据,进一步理解基因和蛋白质在通路中的作用和相互关系。
示例:
假设我们有一个包含基因表达数据的数据框gene_expression_data,其中每一行代表一个基因,每一列代表一个样本。我们还有一个基因注释数据框gene_annotations,其中包含了基因名字和对应的通路ID信息。
# 安装并加载需要使用的语言包
> install.packages(“pathview”)
> library(pathview)
# 使用gene_annotations中的通路ID信息进行基因注释
> pathway_annotations <- gene_annotations$PathwayID
# 运行pathview函数进行可视化
> pathview(gene.data = gene_expression_data,
+ pathway.id = pathway_annotations,
+ species = “hsa”, # 物种代码,这里以人类为例
+ limit = list(gene = 10), # 限制每个通路中显示的基因数量
+ kegg.native = TRUE) # 使用原生KEGG图进行可视化
# 生成可视化图表

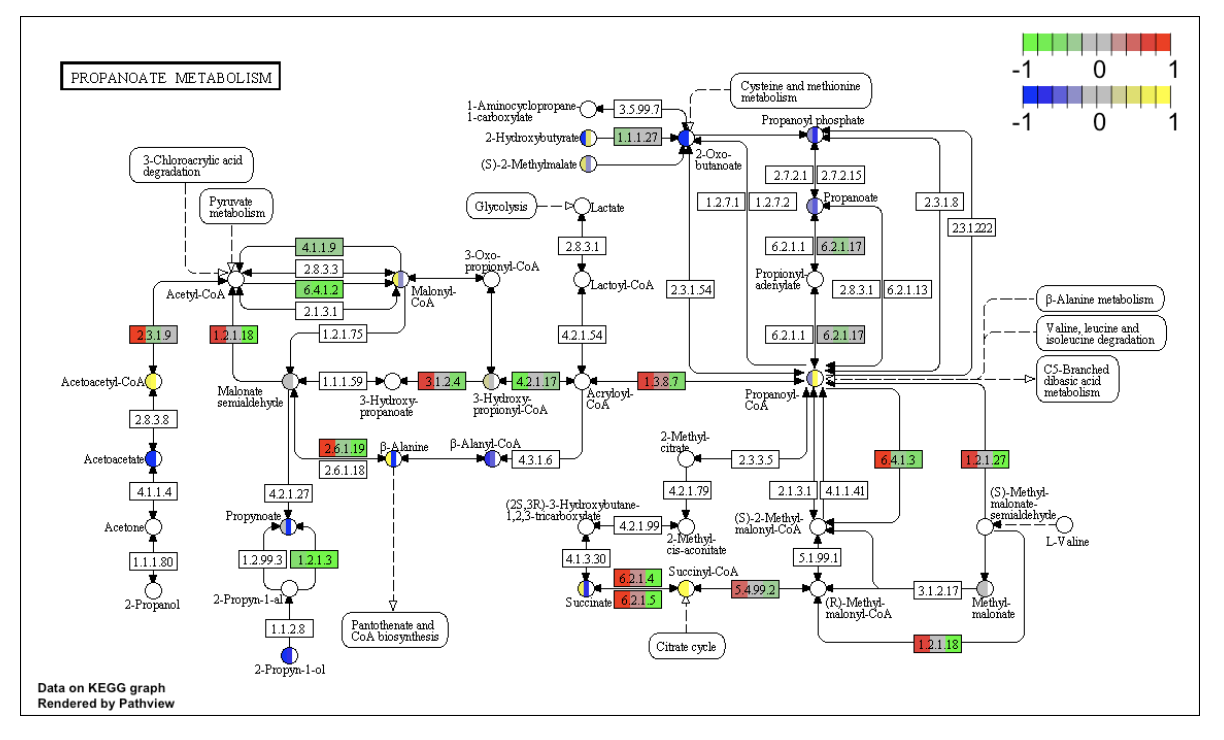
这段代码将基因表达数据与通路ID信息结合起来,然后使用Pathview生成可交互的代谢通路图。你可以在图表中看到每个通路中基因的表达水平,不同颜色表示不同的表达水平。此外,图表还提供了交互式功能,允许你进一步探索基因在通路中的分布情况。
在这个示例中,你可以从生成的图表中获得有关基因在代谢通路中表达的信息。你还可以根据需要进行调整,例如修改限制的基因数量,选择不同的物种等。
除了基本的可视化功能,Pathview还提供了一系列分析工具,用于对数据进行统计学分析和差异性分析。这些工具可以帮助研究人员鉴别出在不同条件下显著差异表达的基因或蛋白质,并进一步探究它们在生物通路中的功能。通过这些功能,Pathview不仅可以在数据层面上提供信息,还能够帮助解释这些信息在生物学过程中的意义。
以上就是对R语言包pathview的简单介绍啦,R语言包Pathview是一个强大的生物学数据分析工具,通过可视化和分析生物通路中的基因组学数据,帮助研究人员更好地理解复杂的生物学过程。它为生物学家们提供了一个直观的平台,用于探索基因和蛋白质在通路中的作用,从而推动生命科学领域的研究进展。无论是揭示疾病机制、发现新的治疗靶点,还是理解细胞信号传导网络,Pathview都为研究人员提供了有力的支持。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html
References: