今天小果带大家使用spaceranger进行空间转录组的分析。
空间转录组简介
传统的单细胞测序,是将细胞组织制备成细胞悬浊液,这样一来就丢失了细胞所处位置的空间信息。空间转录组(spatial gene expression), 简称ST-seq, 在此基础之上将转录组学,单细胞测序技术以及组织切片技术结合起来的技术。而组织切片将提供细胞的空间信息。

通过进行空间转录组测序,就能知道一片组织的细胞类型,及其位置,这对研究肿瘤组织会有很大的帮助。
10×genomics Visium 空间转录组测序平台
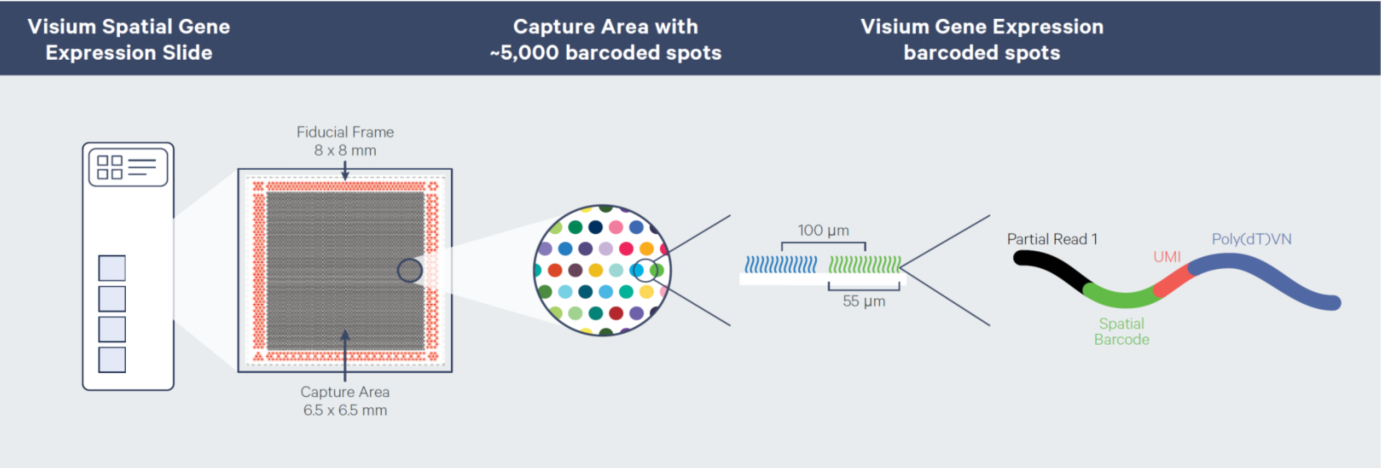
10x Visium 使用特制的玻片,玻片上有捕获区域(capture area),捕获区域是一个6.5*6.5mm 的正方形区域,每个捕获区域包含约 5000 个 spot,每个 spot 上结合有捕获序列,捕获序列有barcode序列记录spot 位置信息和 poly(dT)序列结合 mRNA的poly(A)尾,在捕获mRNA 的同时也保留了 mRNA 的位置信息。转录本的位置信息结合染色拍照结果即可还原转录本在组织的位置分布。
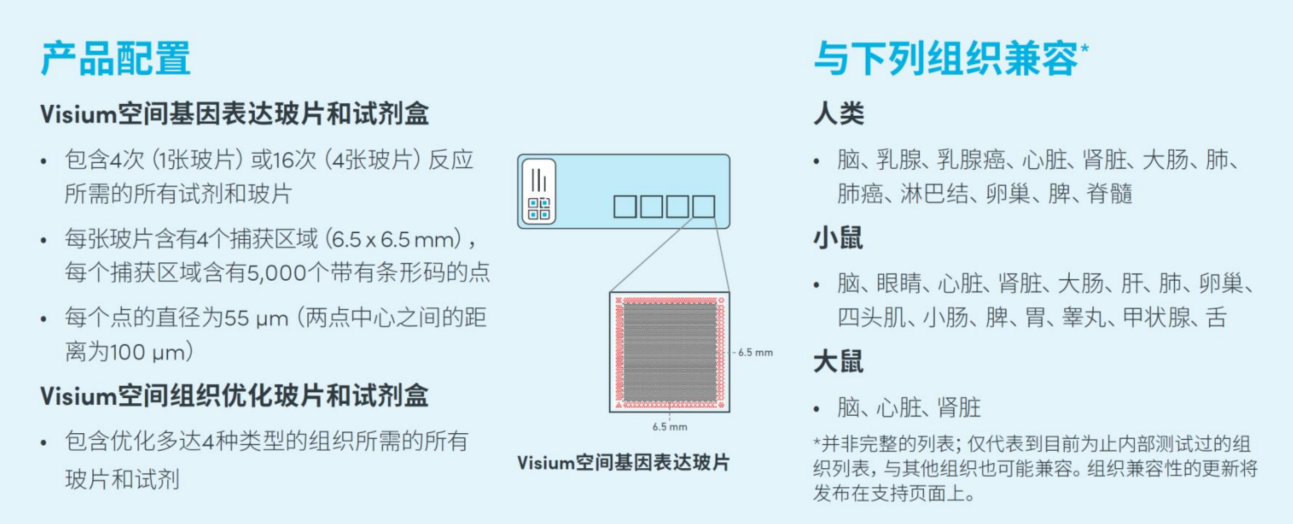
目前能做的主要还是人类和老鼠,其他的还不能做。
总体工作流程
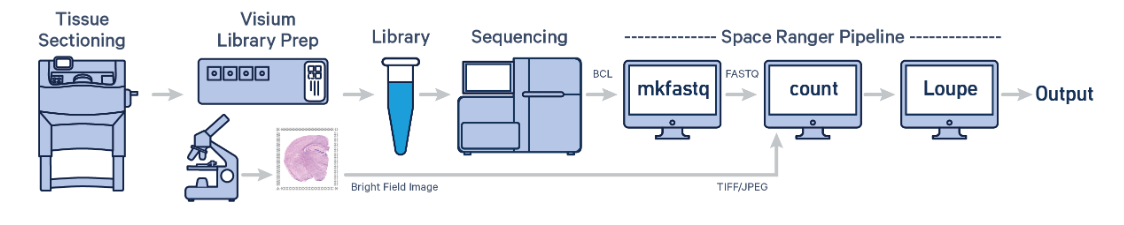
首先选择组织与切片,将切片放置在玻片上,剩下的建库测序就和二代测序一致了。
新鲜冷冻(FF)组织的空间基因表达是使用spaceranger计数软件来确定的,该软件以视觉载玻片的显微镜图像(TIFF或JPEG格式)和样本FASTQ文件作为输入。软件执行对齐、组织和基准检测以及条形码/UMI计数。输出捕获特征点矩阵、聚类和差异基因表达(DGE),这些可以通过在Loupe Browser中进行进一步分析和可视化。
准备工作
下载spacerranger软件
curl -o spaceranger-2.1.0.tar.xz “https://cf.10xgenomics.com/releases/spatial-exp/spaceranger-2.1.0.tar.xz?Expires=1691721798&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA&Signature=nKggndnkZPv4IEuizxHUM5KSXSbtMV7Tk1ICeslkhK09CuFDeOkPBJQcY1yti7s2fMz8i9b2lySWmBeoFdXMS23~ioITj-L88h0ldpTo7G~pqKvMgIssE5b67C4XZ3FprDdceGIaatl4nt~Vrjg~31FZcbG-JxJAQLFf92XLT1tSfSuovhTb5lFkY3oLTfR6Ozvo7Hbx6bibCqSh4MrEefajch4OvdKIlR0l3svzxX0ExETxBiKJw6PBZJu5qYX6vMtDz2anb0-N4-cbhv5Opl3NlT8Xd-7v3mKDCLN37zM-m4vO9EF6YWG2sBljc7ehxsWQnMWw60VhvtbKVb9ULw__”
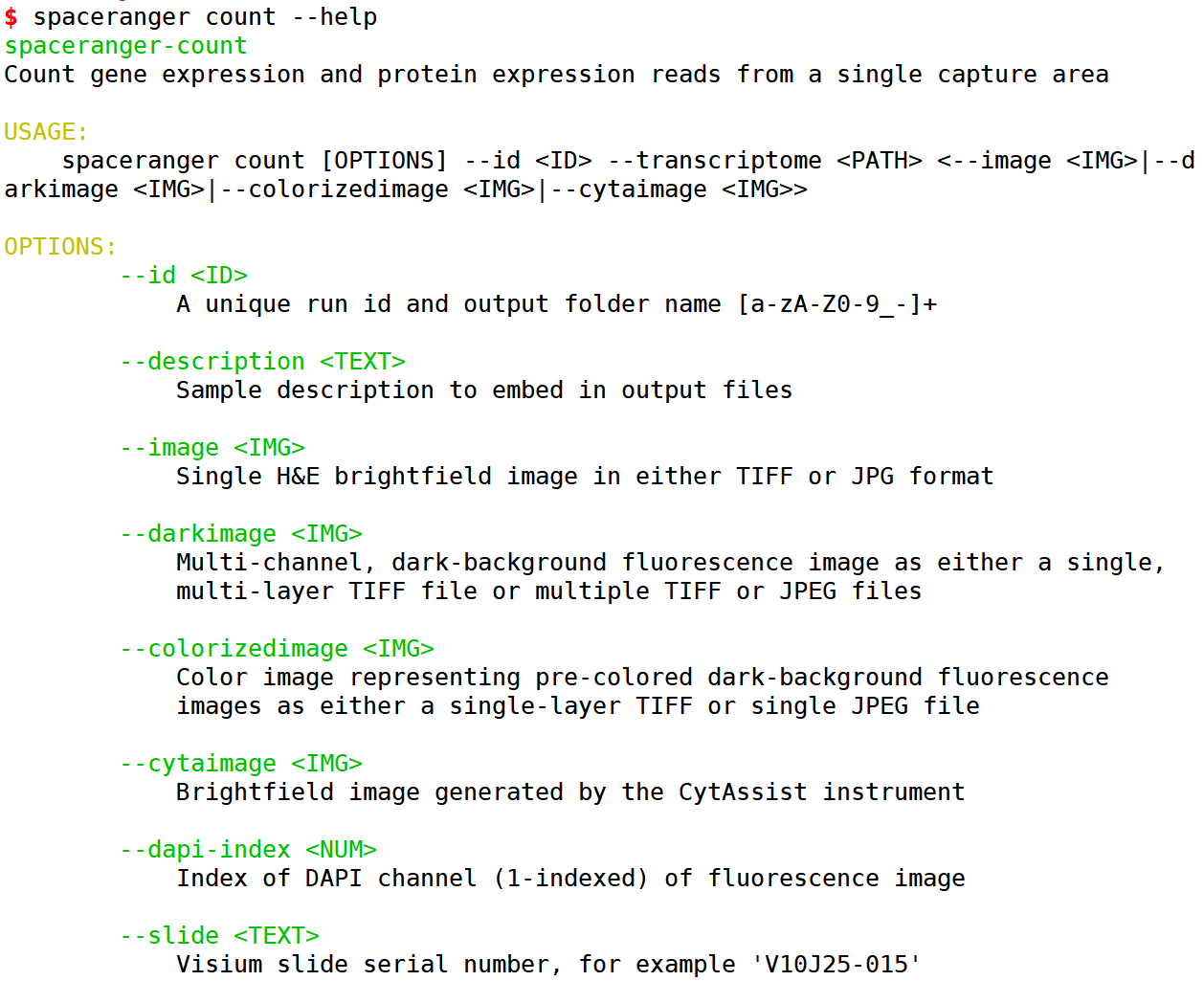
输出软件的帮助文档,即为安装成功。
数据集信息
小果在本次教程中使用的是小鼠大脑冠状切片数据集
数据集的主要信息包括:
10µm厚的组织切片
使用Nikon Ti2-E显微镜获取的H&E图像
测序深度:每个点115569个读取对
测序覆盖范围:reads 1-28bp(包括16bp的空间条形码,12bp的UMI);reads 2-120bp(转录本);i7样本指数-10bp;i5样本指数-10 bp
Visium幻灯片:V19L01-041
捕获区域:C1
示例数据集包括具有基准帧清晰视图的明场图像,从而能够使用spaceranger自动图像处理流水线。
下载数据集与参考序列
# Create datasets folder
mkdir datasets
# Download FASTQ to datasets folder
curl https://s3-us-west-2.amazonaws.com/10x.files/samples/spatial-exp/1.1.0/V1_Adult_Mouse_Brain/V1_Adult_Mouse_Brain_fastqs.tar -o datasets/V1_Adult_Mouse_Brain_fastqs.tar
# Download image file to datasets folder
curl https://cf.10xgenomics.com/samples/spatial-exp/1.1.0/V1_Adult_Mouse_Brain/V1_Adult_Mouse_Brain_image.tif -o datasets/V1_Adult_Mouse_Brain_image.tif
# Download mouse reference
curl -O https://cf.10xgenomics.com/supp/spatial-exp/refdata-gex-mm10-2020-A.tar.gz
解压缩数据集与参考序列
# 解压缩测序数据
tar -xvf V1_Adult_Mouse_Brain_fastqs.tar -C ./ && rm V1_Adult_Mouse_Brain_fastqs.tar
# Extract mouse reference transcriptome
tar -xzvf refdata-gex-mm10-2020-A.tar.gz && rm refdata-gex-mm10-2020-A.tar.gz
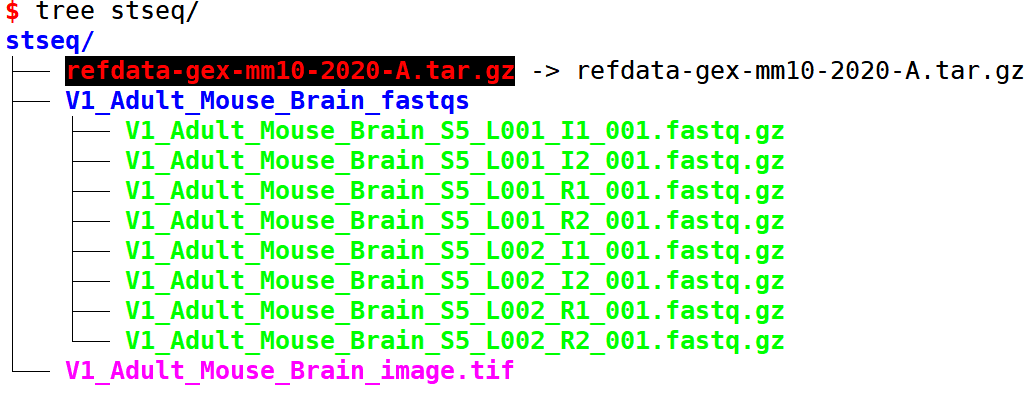
解压成功后将会包含以上文件
spaceranger分析
现在我们所有的输入文件(参考基因,测序文件,切片照片)都准备好了,就可以使用spaceranger来进行分析。
spaceranger count –id=”V1_Adult_Mouse_Brain” \
–description=”Adult Mouse Brain (Coronal)” \
–transcriptome=refdata-gex-mm10-2020-A \
–fastqs=V1_Adult_Mouse_Brain_fastqs \
–image=V1_Adult_Mouse_Brain_image.tif \
–slide=V19L01-041 \
–area=C1 \
–localcores=16 \
–localmem=128
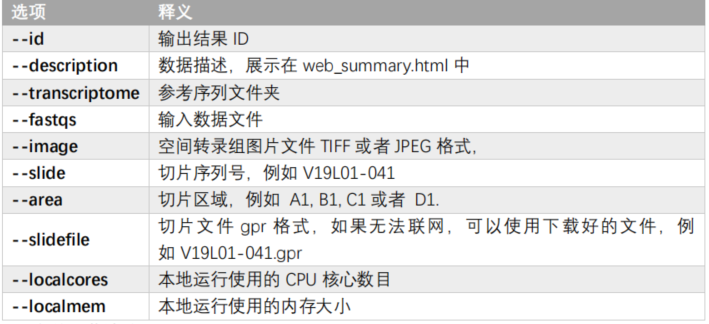
spaceranger参数含义

运行好后将会看到以上输出。
输出的文件包括
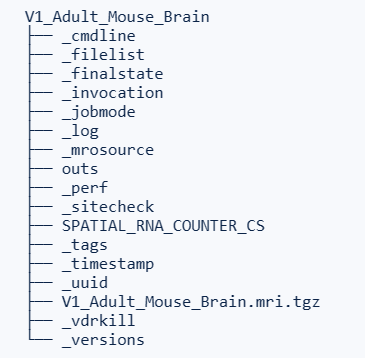
- outs包含所有最终生成的输出
- V1_Adult_Mouse_Brain.mri.tgz包含有助于 10x 基因组学支持解决任何错误的诊断信息
- _sitecheck捕获类似于 SiteCheck 子命令的系统配置
- _timestamp包含有关运行时的信息
- _cmdline捕获为运行而提供的命令count
- _versions包含运行中使用的软件版本
outs下的输出文件
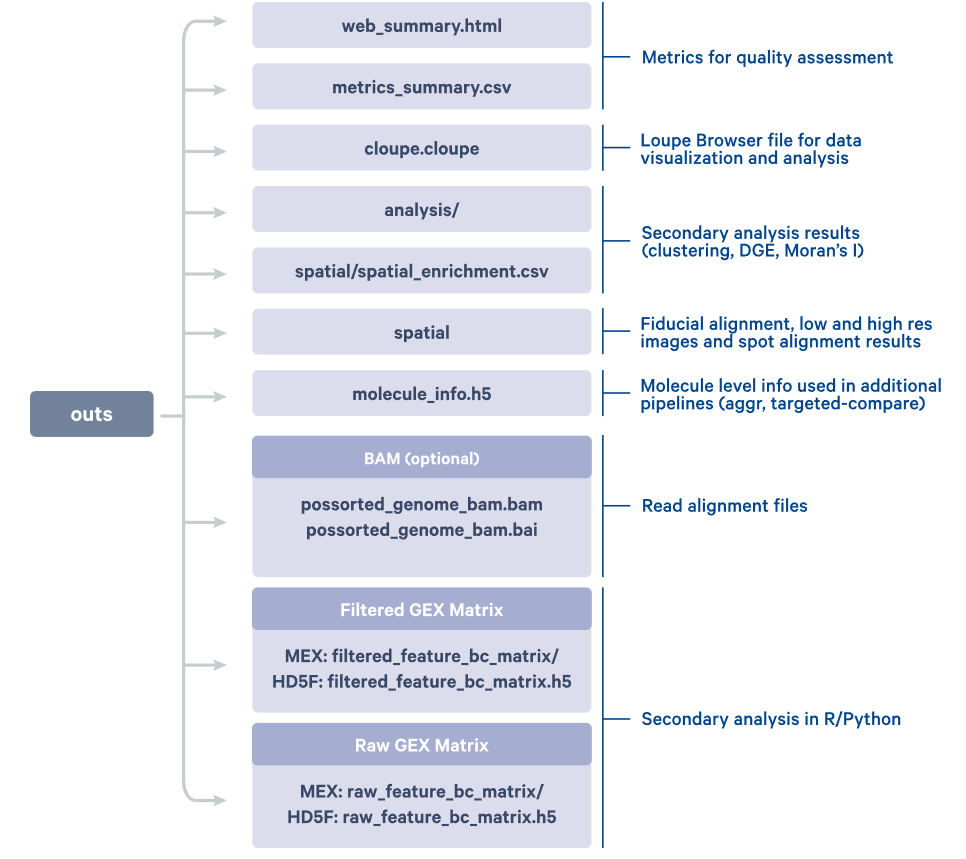
从上至下依次是,分析总结结果,loupebrowser分析的文件,二级分析结果(聚类、DGE、Moran’ s I),基准对准、低分辨率和高分辨率图像以及光斑比对结果,bam比对文件,下游分析的文件。
感兴趣的小伙伴自己动手试试吧。小果今天的空间转录组分析课就到这里了。我们下次见~
更多学习资源请大家移步云生信平台(云生信 – 学生物信息学 (biocloudservice.com))搜索更多资源哦!