hello,上一篇我们讲了如何安装和配置RepeatModeler以及RepeatMasker这两个软件,怎么样,你有没有和小果一起安装完成呢?今天我们进入我们的正题,也就是如何组合使用这两个软件对目标基因组进行重复序列的注释,感兴趣的话就和小果一起看下去吧!
什么是重复序列注释?
在对目标基因组基因组进行注释前,小果来介绍一下什么是重复序列的注释:
重复序列的注释是指对基因组中的重复序列进行识别、分类和标记的过程。在基因组中,重复序列是指在DNA序列中存在多次重复出现的片段,可以包括简单重复序列(如短串联重复、微卫星等)和复杂重复序列(如转座子、LTR反转录转座子等)。重复序列在基因组的结构和功能中起着重要的作用。
重复序列的注释过程通常使用各种计算方法和工具来完成,其中包括重复序列数据库的比对、模式匹配、序列比较、机器学习等技术。注释的目标是确定基因组序列中哪些部分是重复序列,并提供关于这些重复序列的相关信息。
重复序列注释可以提供以下信息:
- 重复序列的位置:注释会指示重复序列在基因组中的具体位置,以帮助研究人员了解它们在基因组中的分布情况。
- 重复序列的类别:注释可以将重复序列分为不同的类别,如低复杂度序列、转座子等,以便更好地理解它们的性质和功能。
- 重复序列的数量和长度:注释可以提供重复序列的数量和长度统计信息,帮助研究人员了解它们在基因组中的丰度和大小。
- 重复序列的家族关系:注释可以将相似的重复序列聚类成家族,并提供它们之间的家族关系信息,有助于研究重复序列的进化和起源。
重复序列的注释为研究人员提供了关于基因组结构、演化和功能的重要信息,对于进一步理解基因组的组成和机制具有重要意义。
RepeatModeler+RepeatMasker组合使用
数据准备
对于我们即将注释的数据文件,大家可以通过NCBI官网搜索自己感兴趣的物种对应的基因组序列并下载即可哦,在这里小果以香蕉最新发布的完整基因组文件(DH pahang栽培品种)为例,在这里给出下载链接:
Musa acuminata malaccensis | Banana Genome Hub (southgreen.fr)
RepeatModeler构建重复序列库
在进行重复序列注释前,我们需要使用RepeatModeler来预测基因组重复序列,以为下一步的RepeatMasker建立重复序列库,具体的操作主要分为以下三步:
建库
step1:
./BuildDatabase -name musa1 -engine ncbi /share/home/xiongchu/downloads/complete-gene-multi/banana/Musa_acuminata_pahang_v4.fasta
# -engine ncbi: 表示使用rmblast为搜索引擎
# -name musa1: 表示建立数据库的名字为musa1
# Musa_acuminata_pahang_v4.fasta:香蕉基因组文件
操作完成后,我们得到以下文件:
其它参数说明:
#建库,利用rice 一个实例fasta文件
BuildDatablase –name ricedb –engine ncbi rice.sample.2.fa
#参数说明
-name <database name>
The name of the database to create.
-engine <engine name>
The name of the search engine we are using. I.e abblast/wublast or rmblast.
-dir <directory>
The name of a directory containing fasta files to be processed. The
files are recognized by their suffix. Only *.fa and *.fasta files
are processed.
-batch <file>
The name of a file which contains the names of fasta files to
process. The files names are listed one per line and should be fully
qualified.
RepeatModeler -database ricedb -pa 5
#参数说明
-database
The name of the sequence database to run an analysis on. This is the
name that was provided to the BuildDatabase script using the “-name”
option.
-pa #
Specify the number of parallel search jobs to run. RMBlast jobs will
use 4 cores each and ABBlast jobs will use a single core each. i.e.
on a machine with 12 cores and running with RMBlast you would use
-pa 3 to fully utilize the machine.
-recoverDir <Previous Output Directory>
If a run fails in the middle of processing, it may be possible
recover some results and continue where the previous run left off.
Simply supply the output directory where the results of the failed
run were saved and the program will attempt to recover and continue
the run.
step2:
./RepeatModeler -database musa1 -engine ncbi -pa 20 &> musa1.out &
# -database 要和上一步一致
# -engine 要和上一步一致
# -pa 表示线程数
这一步的操作需要的时间较长,大家耐心等待哦~
操作完成后将产生以下文件:

而对于之后重复序列的注释工作,我们要用到的文件也就是musa1-families.fa文件,作为我们产生的重复序列库文件。
RepeatMasker进行重复序列注释
接下来,我们就可以使用RepeatMasker进行重复序列的注释工作了,在执行注释命令之前,需要提前创建一个文件夹来存放我们注释后的结果文件哦,在这里小果创建了名为bnrpt的文件夹作为结果文件夹,大家根据自己的情况来创建哦!
然后我们执行以下命令
./RepeatMasker -e rmblast -lib /share/home/xiongchu/downloads/repeat-soft/RepeatModeler-2.0.1/musa1-families.fa -pa 16 -dir bnrpt1 /share/home/xiongchu/downloads/complete-gene-multi/banana/Musa_acuminata_pahang_v4.fasta
总结:
操作命令的基本语法为:
./RepeatMasker -e 搜索引擎名称 -lib 重复序列库的路径 -pa 并行线程数 -dir 结果文件夹名称 基因组文件路径
 运行后我们来看一下产生的目录:
运行后我们来看一下产生的目录:
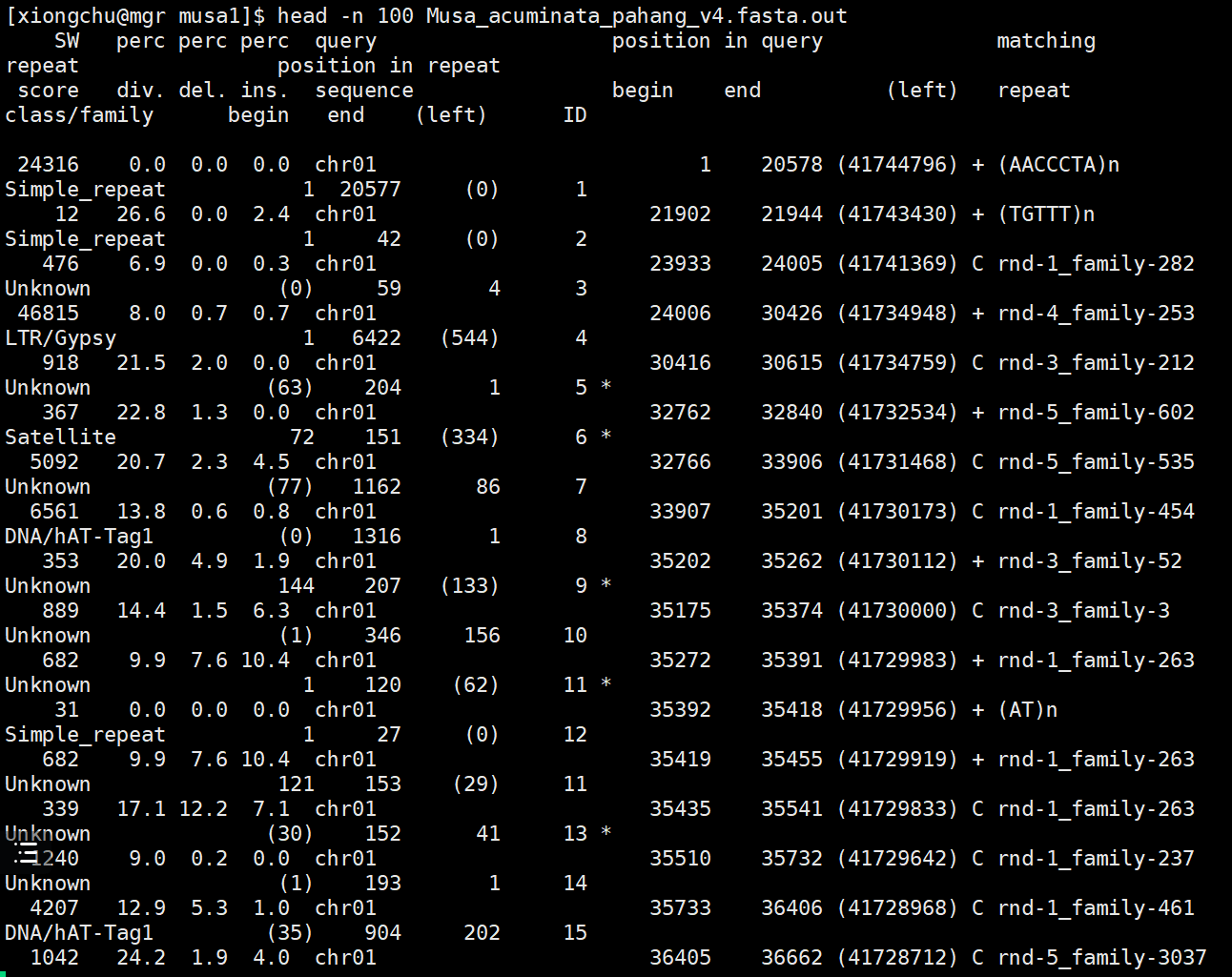
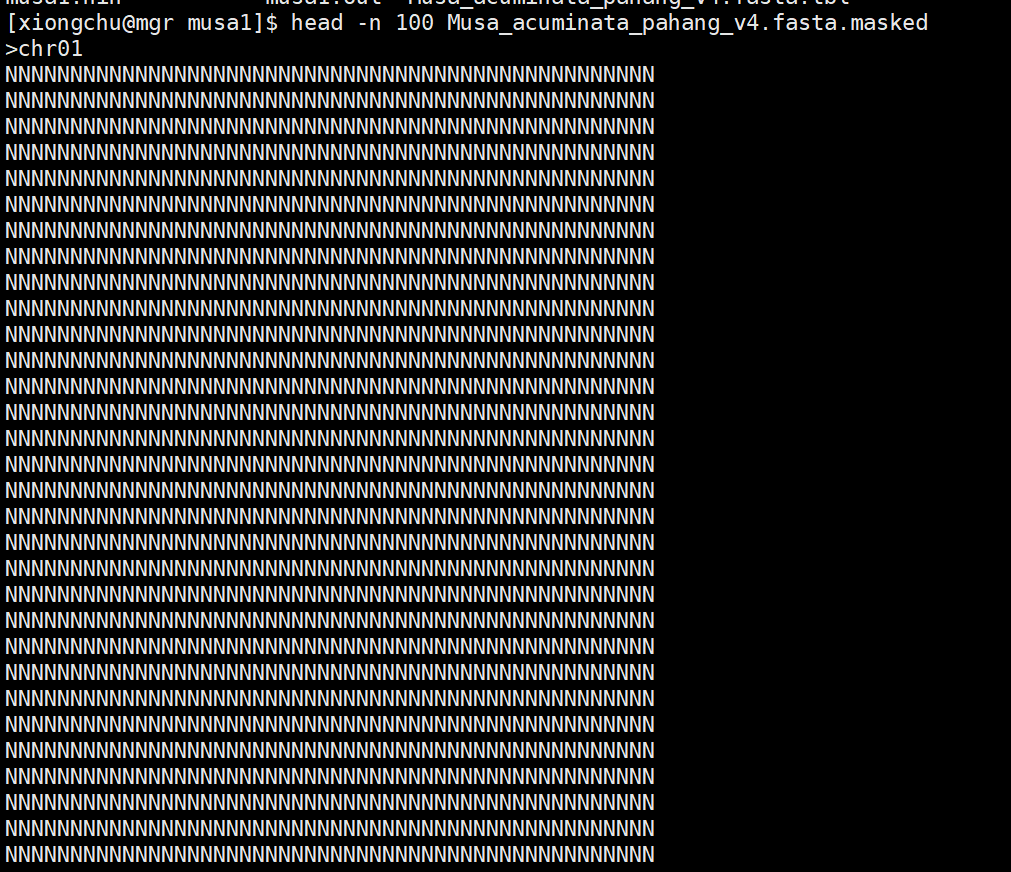
其中,.tbl文件为重复序列注释完成后的汇总信息文件,.out文件为基因组中所有被注释的区域汇总文件,.masked文件为被掩蔽后的基因组文件,我们一起来看一下这几个文件的结构:
.tbl文件:
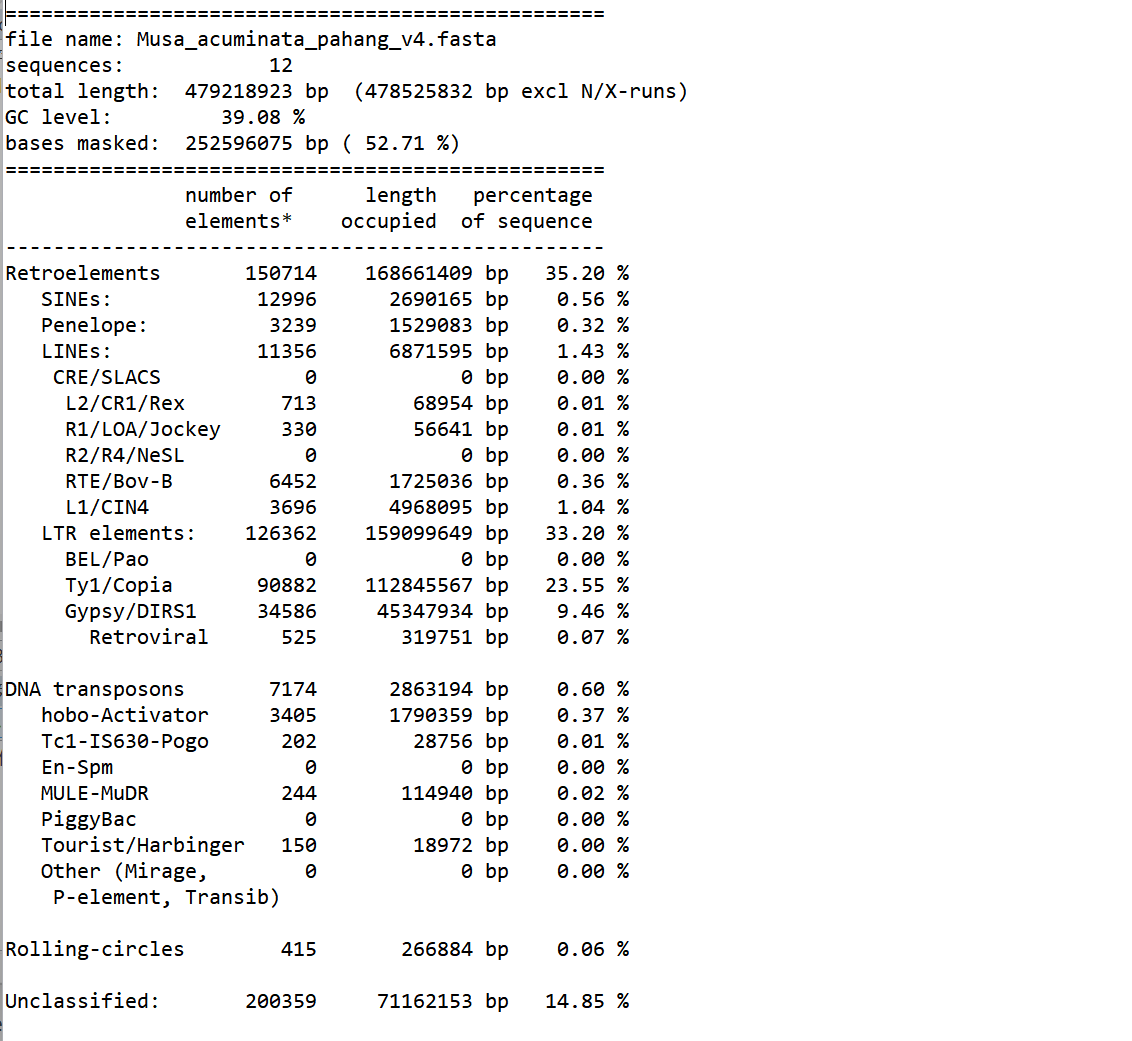
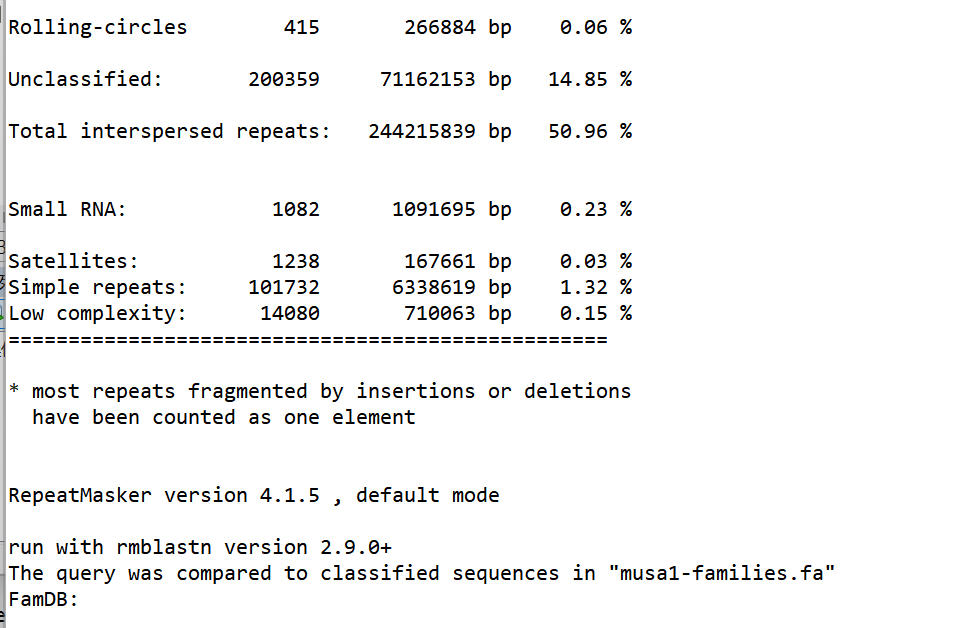
.out文件:
.masked文件:
怎么样,以上就是这两个软件组合使用的教程,你学会如何给基因组进行重复序列注释了嘛?快和小果一起操作起来!!