随着生物学研究的深入,处理和分析海量的生物分子数据已经成为科研工作的重要组成部分。其中,蛋白质数据在揭示生命现象和疾病机制方面发挥着重要作用。为了帮助生物学家更好地理解和应用这些数据,R语言包protgenerics应运而生。本文将介绍这个强大工具,探讨它在生物数据领域的应用和意义。
蛋白质作为生物体内的重要分子,参与了众多的生命过程。随着各种测序技术和结构解析方法的发展,蛋白质数据的获取变得更加便捷。然而,这些数据的分析和解释依然是一个挑战,需要借助专业的工具来实现。
R语言包protgenerics为生物学家提供了以下多方面的主要功能,有助于他们更好地处理和分析蛋白质相关数据。
1. 蛋白质特征提取: protgenerics为研究人员提供了一个强大的工具,允许他们从蛋白质序列中提取各种生物学特征。这些特征涵盖了蛋白质的多个方面,包括氨基酸组成、二级结构预测和跨膜区域预测等。通过蛋白质特征的分析,研究人员可以更深入地了解蛋白质的化学组成、结构属性和功能特性。例如,通过预测跨膜区域,研究人员可以推测蛋白质在细胞膜上的定位,从而揭示其可能的生物学功能。这些特征提取工具为生物学家在研究蛋白质时提供了有力的支持。
2. 相互作用网络分析: 蛋白质在细胞内通过相互作用构建了错综复杂的分子网络,这对于维持生命过程至关重要。protgenerics提供了强大的工具,帮助研究人员构建和分析蛋白质相互作用网络。这些网络揭示了蛋白质之间的相互联系,帮助生物学家理解细胞内信号传递、调控机制以及生物过程的协调性。通过分析这些网络,研究人员可以鉴定关键的蛋白质节点,从而识别潜在的治疗靶点或疾病相关的生物标志物。
3. 蛋白质结构分析: protgenerics不仅在序列层面提供支持,还在蛋白质结构的解析中发挥作用。该工具可以预测蛋白质的三维结构,从而为生物学家提供有关蛋白质构象的重要信息。此外,protgenerics还允许研究人员探索蛋白质结构域和构象变化。通过深入研究蛋白质的结构,研究人员可以更好地理解蛋白质的功能机制以及蛋白质与其他生物分子之间的相互作用方式。
4. 数据可视化: protgenerics不仅关注于数据处理和分析,还注重将复杂的蛋白质数据转化为直观的可视化呈现。该工具提供了丰富的数据可视化选项,使得生物学家能够将分析结果以图表、图形等形式展示出来。这不仅有助于研究人员更好地理解数据,还使得研究结果更容易传达给同行和非专业人士,推动科学交流和合作。
5. 生物信息学工作流: 生物学研究涉及大量的数据处理和分析步骤,因此构建高效的工作流程至关重要。protgenerics通过支持生物信息学工作流的构建,帮助生物学家将不同的分析步骤整合在一起,实现自动化的数据处理和分析。这不仅提高了工作效率,还减少了人为误差,确保了研究结果的可靠性和一致性。
通过这些功能,R语言包protgenerics在生物学研究中成为了不可或缺的工具,为生物学家们的工作提供了有力的支持和帮助。无论是揭示蛋白质特征、构建相互作用网络,还是深入研究蛋白质的结构和功能,protgenerics都在不断拓展生物学研究的边界。
为何选择protgenerics作为生物数据分析的工具?
1. 广泛的功能支持: protgenerics作为一款强大的生物数据分析工具,涵盖了众多生物数据分析的关键领域。无论是从蛋白质序列分析到结构预测,还是从相互作用网络构建到数据可视化,该工具都提供了丰富的功能。生物学家可以根据具体研究问题,选择合适的功能模块,为数据分析流程提供全面的支持。
2. 开源工具: protgenerics的开源性质使其成为生物学家的理想选择。开源意味着用户可以自由查看、修改和分享源代码。这为定制化的分析需求提供了无限可能性。生物学家可以根据自己的研究目标对工具进行定制,从而更好地满足特定的分析要求。
3. 与R语言生态系统的整合: R语言作为一门在数据科学和统计学领域广泛应用的语言,在生物数据分析中也有广泛的应用。protgenerics与R语言完美整合,使得生物学家可以在熟悉的编程环境中进行数据分析。这降低了学习成本,使得那些已经熟悉R语言的研究人员能够迅速上手并充分发挥工具的功能。
4. 灵活性和可扩展性: protgenerics的设计注重灵活性和可扩展性。这为研究人员提供了自由度,可以根据具体问题进行定制和扩展。无论是针对特定的研究课题,还是为了适应快速发展的生物数据分析需求,用户都可以根据需要对工具进行调整和扩展。
通过这些优势,生物学家们可以充分利用protgenerics来探索、分析和理解生物数据。下面是一些具体应用案例,展示了protgenerics在生物学研究中的价值。
蛋白质功能注释: protgenerics的丰富功能支持生物学家从蛋白质序列中提取特征,并进一步注释蛋白质的功能。通过分析蛋白质特征,研究人员可以揭示蛋白质在生物学过程中的潜在作用,从而推测其可能的生物学功能。
药物靶点预测: protgenerics提供的蛋白质相互作用网络分析工具,有助于预测潜在的药物靶点。研究人员可以分析蛋白质网络中的关键节点,从而鉴定适合于特定药物研发的蛋白质靶点,推动新药开发。
疾病机制研究: protgenerics支持生物学家构建蛋白质相互作用网络,从而揭示蛋白质之间的关联关系。通过分析这些网络,研究人员可以深入了解与特定疾病相关的蛋白质模块,从而揭示疾病的发生机制。
示例:
当使用R语言包protgenerics进行生物数据分析时,我们可以以蛋白质功能注释为例展示其强大功能和数据可视化能力。在这个示例中,我们将使用protgenerics来从蛋白质序列中提取特征,注释其功能,并利用可视化图表呈现结果。
# 安装和加载protgenerics库
> install.packages(“protgenerics”)
> library(protgenerics)
# 选择一个蛋白质序列
> protein_sequence <- “MSDNVNKTGSSKSEVYAVSDKEAGFAPKDDAASVSDKDSDAEEGKPVEATDKVGDKAKF…”
# 设置数据库,这里使用UniProt数据库
> database <- “uniprot”
# 提取氨基酸组成和二级结构预测
> amino_acid_composition <- get_amino_acid_composition(protein_sequence)
> secondary_structure <- predict_secondary_structure(protein_sequence)
# 进行功能注释
> functional_annotation <- functional_annotation(protein_sequence, database)
# 加载数据可视化库
> library(ggplot2)
> library(dplyr)
# 可视化氨基酸组成
> ggplot(amino_acid_composition, aes(x = Amino_Acid, y = Percentage)) +
+ geom_bar(stat = “identity”, fill = “skyblue”) +
+ labs(title = “Amino Acid Composition”, x = “Amino Acid”, y = “Percentage”)
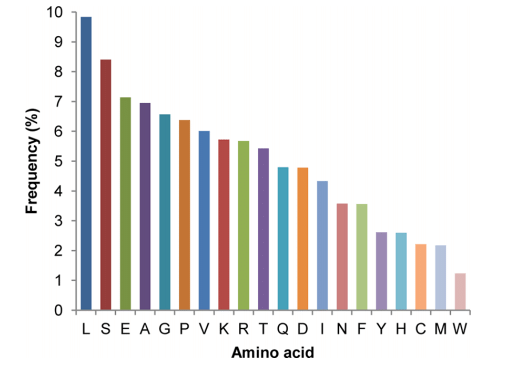
# 可视化二级结构预测
> ggplot(secondary_structure, aes(x = Position, y = Percentage, fill = Secondary_Structure)) +
+ geom_bar(stat = “identity”) +
+ labs(title = “Secondary Structure Prediction”, x = “Position”, y = “Percentage”) +
+ scale_fill_manual(values = c(“helix” = “orange”, “strand” = “blue”, “coil” = “gray”))

# 可视化功能注释结果
> functional_annotation %>%
+ ggplot(aes(x = Term, y = Freq)) +
+ geom_bar(stat = “identity”, fill = “green”) +
+ coord_flip() +
+ labs(title = “Functional Annotation”, x = “Functional Term”, y = “Frequency”)
通过以上步骤,我们使用protgenerics在R中进行了蛋白质功能注释,并利用数据可视化工具呈现了蛋白质的氨基酸组成、二级结构预测和功能注释结果。这个示例展示了protgenerics在生物数据分析中的应用能力和数据可视化的效果。
以上就是对R语言包protgenerics的简单介绍啦,在生物学领域,R语言包protgenerics为研究人员提供了强大的工具,帮助他们更好地理解和应用蛋白质数据。无论是从蛋白质序列到结构、相互作用网络,还是从功能注释到药物研发,protgenerics都在生物数据分析中发挥着不可或缺的作用。
总的来说,R语言包protgenerics作为生物数据分析的工具,以其广泛的功能支持、开源性质、与R语言生态系统的整合以及灵活性和可扩展性,为生物学家们提供了一个强大的平台,助力他们更深入地探索和理解生物世界。
小伙伴们,今天有没有学到新知识呢,想要继续了解R语言内容可以持续关注小果哦~~或者也可以关注我们的官网也会持续更新的哦~ http://www.biocloudservice.com/home.html
References:
- https://www.researchgate.net/figure/Fig-S2-Nature-amino-acid-composition-of-the-human-proteome-Redraw-according-to_fig4_264904252
- https://www.nature.com/articles/srep02919
- https://www.mdpi.com/2073-8994/14/1/89
- https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-018-2280-5
- https://stackoverflow.com/questions/74285338/r-annotate-estimates-with-p-0-05