小伙伴们大家好呀~在之前的分享中,小果给大家详细地讲解了分子对接的操作流程以及Libdock和GOLD的详细教程,那么这一次小果就给大家带来分子对接优化——CDOCKER的详细教程啦,废话不多说,让我们直接开始吧~
为什么需要CDOCKER?
在前面的内容中,小果曾经给大家讲解过,libdock和gold属于high-throughput screening分栏,而CDOCKER和In situ ligand minimization属于docking optimization分栏。
High-throughput screening(高通量筛选)是一种药物发现和化合物筛选的技术和方法。它通过自动化和并行化处理大规模化合物库,快速测试和评估潜在的药物分子或化合物集合与特定生物目标之间的相互作用。而Docking optimization(对接优化)是一种用于分子对接模拟的计算方法,旨在预测蛋白质和小分子之间的结合方式和亲和力。通过对接优化,可以研究蛋白质和小分子之间的相互作用,从而更好地理解它们之间的结合机制和影响因素。
对接优化的目标是寻找最佳的蛋白质-小分子结合位姿,并预测它们之间的结合亲和力。这对于药物设计和药物发现非常重要,因为药物的活性通常与其在靶点上的结合方式和亲和力密切相关。
看到这里,小伙伴们虽然应该学会了这两大专栏各自的侧重和特点,但还不能很直观地理解两大专栏的区别在哪里,现在小果就给大家说一说它们的区别:
- High-Throughput Screening(高通量筛选): HTS分栏主要关注快速且高通量地筛选大量化合物,以发现具有潜在药物活性的候选分子。该分栏提供了一系列工具和方法,用于处理和评估化合物库。通过高效的计算和筛选流程,HTS可帮助用户迅速缩小化合物范围,并优先考虑具有较高结合亲和力或药效的化合物。其中常用的工具包括虚拟筛选、化学指纹分析等。
- Docking Optimization(对接优化): Docking Optimization分栏用于进一步优化和改善分子对接的结果。它主要关注如何通过调整配体的构象和位姿来提高配体与蛋白质的结合能和准确性。这一分栏提供了针对分子对接的多种工具和算法,如基于引导式模拟和分子动力学模拟的方法,以及结合了遗传算法的优化方法。用户可以使用这些工具来进行系统性的对接优化,以获得更可靠和准确的分子结合模型。
因此,HTS分栏主要用于快速筛选和初步评估大量化合物,找到具有潜在活性的候选分子;而Docking Optimization分栏则专注于对初始对接结果进行进一步优化,以提高对接精度和可靠性。两个分栏在药物发现过程中扮演不同的角色,帮助用户进行不同层次和目标的工作。
从我们使用者的角度来说,大家可以理解为需要先进行HTS分栏的操作,然后在使用Docking Optimization分栏进行优化,这么说完小伙伴们肯定可以理解透彻并且融汇贯通啦。这也是为什么小果先给大家详细分享了Libdock和GOLD然后分享CDOCKER的原因。
聪明的小伙伴一定还有疑问,Docking Optimization分栏下的In situ ligand minimization和CDOCKER有什么区别呢?
- 方法原理:In situ ligand minimization是通过应用能量最小化算法对单个配体进行优化,调整其构象和能量,以改善与蛋白质的结合性能。CDOCKER则使用引导式分子动力学模拟方法,在结合位点中模拟配体的动力学行为,通过搜索位构象并考虑动力学行为来优化配体的位姿和构象。
- 优化策略:In situ ligand minimization主要关注配体的构象和能量的优化,通过能量最小化算法寻找更低能量状态的构象。CDOCKER则综合考虑配体的位构象、能量和动力学行为进行优化,利用分子动力学模拟和遗传算法来搜索配体的更优位构象。
- 考虑因素:In situ ligand minimization主要关注配体的静态性质,如构象和能量,以改善静态结合性能。CDOCKER更关注配体的动态性质,通过模拟动力学行为来优化配体的位构象,考虑配体在结合位点内的灵活性和动态变化。
综上所述,In situ ligand minimization主要通过调整配体的构象和能量进行优化,而CDOCKER则利用分子动力学模拟方法,并综合考虑配体的位构象、能量和动力学行为。两种方法在优化策略和所考虑的因素上有所不同,小伙伴们可根据需求选择合适的方法进行分子对接的优化工作。小果现在就给大家举几个他们适用的例子来更直观地让大家感受一下:
In situ ligand minimization(原位配体最小化)适用于以下情况:
- 初始结构优化:当已有一个初始的配体-蛋白质结合模型时,可以使用原位配体最小化来进一步优化配体的构象和能量,改善结合性能。
- 配体修饰:当需要对已知的配体进行修饰以获得更好的结合性能时,可以使用原位配体最小化来通过调整构象和能量来评估和改善新的配体构象。
- 蛋白质突变分析:在研究蛋白质突变对配体结合的影响时,可以使用原位配体最小化来比较不同突变体的配体结合能力,从而评估突变对结合性能的影响和机制。
而CDOCKER适用于以下情况:
- 高度柔性的配体:当配体具有高度柔性性质,或者在结合过程中可能发生显著的构象变化时,CDOCKER可以更全面地考虑配体的动态性质和灵活性,通过模拟动力学行为来优化配体的位构象。
- 多个配体分子对接:当需要对多个配体分子进行对接优化时,CDOCKER能够利用分子动力学模拟和遗传算法来搜索多个配体的更优位构象,并考虑它们之间的相互作用。
- 结合位点预测:在进行结合位点预测时,CDOCKER可以用于预测配体在潜在结合位点中的位构象,进而评估配体与蛋白质的结合性能。
CDOCKER参数
以下是CDOCKER的全部参数:
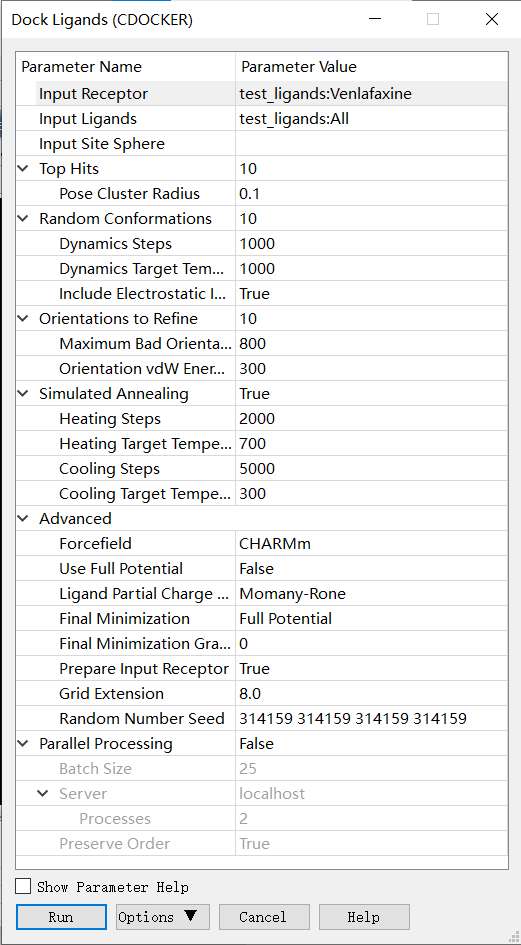
CDOCKER官方文档


CDOCKER参数官方文档


参数解读:
基础设置
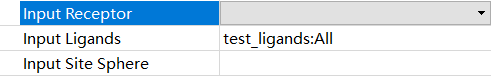
Input Receptor: 选择受体,一般设置为蛋白名:蛋白名;(注意:受体不应包含交替构象)
Input Ligands: 选择对接的小分子,一般设置为小分子名:All;(MDL MOL/SD文件)
Input Site Sphere: 参数为定义好的活性位点的坐标;小伙伴们可以在前面定义活性位点时查看每个位点的坐标,从而在这里进行选择。这一项也可以填写空白,在下一项Input Site Atoms中通过受体原子索引选择。
小果提醒大家:
- 如果配体已经放置在活动位点中,则不需要球体定义。在这种情况下,球体的坐标和半径分别从配体的中心和旋转半径计算得出。
- 球体的半径可能会影响对接结果,因为能量网格的位置会发生变化。
姿态聚类
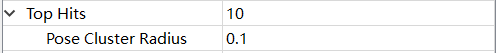
Top Hits:保存的多样化顶部姿态的数量。多样性可以通过姿态聚类半径来控制。
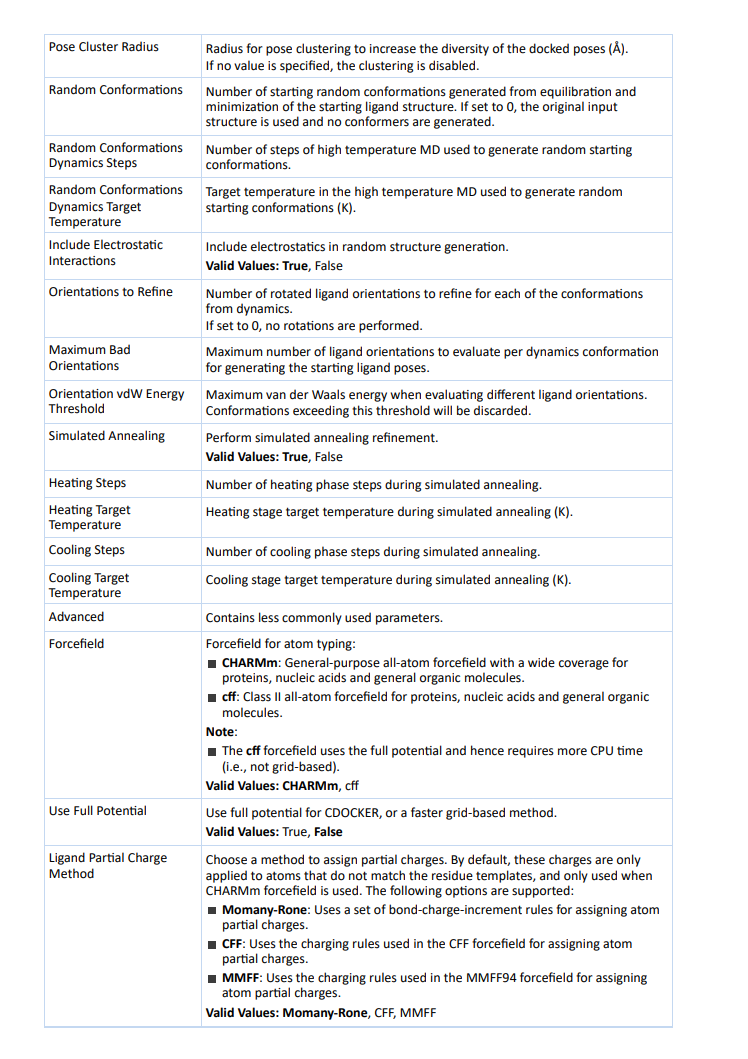
Pose Cluster Radius:设置姿态聚类的半径,以增加对接姿态的多样性(以Å为单位)。如果未指定值,则禁用聚类。
构象生成
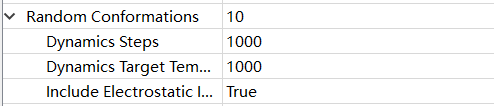
Random Conformations:从起始配体结构的平衡和最小化中生成的随机构象的数量。如果设置为0,则使用原始输入结构,并且不会生成构象。
Random Conformations Dynamics Steps:用于生成随机起始构象的高温分子动力学模拟步数。
Random Conformations Dynamics Target Temperature:用于生成随机起始构象的高温分子动力学模拟的目标温度(以K为单位)。
Include Electrostatic Interactions:在随机结构生成过程中是否考虑静电效应。
旋转方向
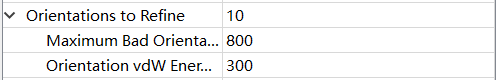
Orientations to Refine:根据动力学提炼每个构象的旋转配体方向的数目。如果设置为0,则不执行旋转。
Maximum Bad Orientations:用于生成起始配体姿势的动力学构象每个构象要评估的最大配体方向数量。
Orientation vdW Energy Threshold:在评估不同配体方向时的最大范德华能量阈值。超过此阈值的构象将被丢弃。
模拟退火

Simulated Annealing:执行模拟退火精炼。
Heating Steps:在模拟退火过程中的加热阶段步数的数量。
Heating Target Temperature:在模拟退火期间的加热阶段目标温度(以K为单位)。
Cooling Steps:在模拟退火过程中的冷却阶段步数的数量。
Cooling Target Temperature:在模拟退火过程中的冷却阶段目标温度(以K为单位)。
其余参数
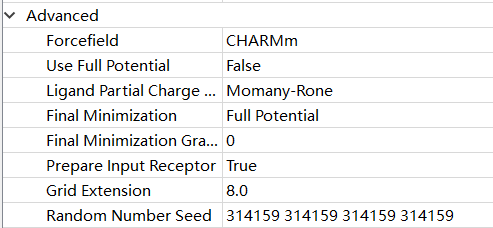
Advanced:一些不太常用的参数。
Forcefield:原子类型的力场: CHARMm:通用的全原子力场,适用于蛋白质、核酸和一般有机分子。 cff:用于蛋白质、核酸和一般有机分子的二级全原子力场。 小果提醒大家:cff 力场使用完整势能,因此需要更多的 CPU 时间(即非基于网格的方法)。
Use Full Potential:对于CDOCKER,可以选择使用完整势能的方法,或者使用更快的基于网格的方法。
Ligand Partial Charge Method:选择一个方法来分配部分电荷。默认情况下,这些电荷仅适用于不符合残基模板的原子,并且仅在使用CHARMm力场时使用。
支持以下选项:
Momany-Rone:使用一组用于分配原子部分电荷的键电荷增量规则。
CFF:使用CFF力场中用于分配原子部分电荷的充电规则。
MMFF:使用MMFF94力场中用于分配原子部分电荷的充电规则。
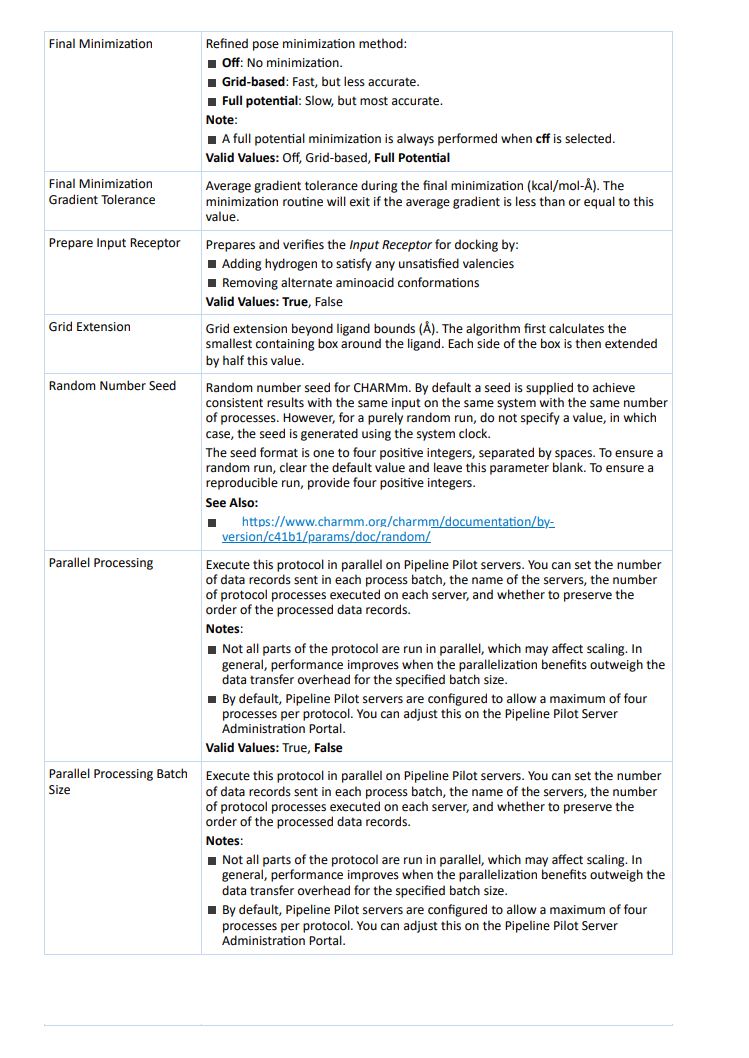
Final Minimization:精细位姿最小化方法
off:不进行最小化。
grid-based:快速,但准确性较低。
full potential:慢速,但准确性最高。
小果提醒大家:当Ligand Partial Charge Method选择cff时,总是执行完整势能最小化。
Final Minimization Gradient Tolerance:最终最小化过程中的平均梯度容限(kcal/mol-Å)。如果平均梯度小于或等于该值,则最小化例程将退出。
Prepare Input Receptor:通过以下方式准备和验证用于对接的输入受体:
- 添加氢原子以满足任何未满足的价键。
- 移除交替的氨基酸构象。
Grid Extension:配体边界之外的网格扩展范围(Å)。算法首先计算围绕配体的最小包含框。然后,将框的每个边延展此值的一半。
Random Number Seed:CHARMm的随机数种子。默认情况下,为了在相同的输入、相同的系统和相同的进程数下获得一致的结果,会提供一个种子。然而,如果需要进行完全随机的运行,则不要指定一个值,此时种子将使用系统时钟生成。
种子的格式是一个到四个正整数,用空格分隔。为了确保随机运行,请清除默认值并将此参数留空。为了确保可重现的运行,请提供四个正整数。
参考链接:https://www.charmm.org/charmm/documentation/by-version/c41b1/params/doc/random/
并行执行
Parallel Processing:在Pipeline Pilot服务器上并行执行此协议。
Parallel Processing Batch Size:在Pipeline Pilot服务器上并行执行此协议。我们可以设置每个进程批次中发送的数据记录数量,服务器的名称,每个服务器上执行的协议进程数量,以及是否保留已处理数据记录的顺序。
小果提醒大家:
不是协议的所有部分都会并行运行,这可能会影响扩展性。一般而言,当并行化的好处超过指定批次大小的数据传输开销时,性能会提高。 默认情况下,Pipeline Pilot服务器配置为允许每个协议最多四个进程。我们可以在Pipeline Pilot Server Administration Portal上进行调整。
Parallel Processing Server:若要执行并行处理,需要提供一个逗号分隔的服务器列表(服务器名称:端口号)。
例如:
- 192.168.1.100:8080, 192.168.1.101:8080, 192.168.1.102:8080
- myserver1:8000, myserver2:8000, myserver3:8000
如果要包括本地服务器,请添加 “localhost”。这在多CPU/多核系统上(将子协议作业生成在同一台机器上)和Pipeline Pilot Linux集群上是有用的(其中 “localhost” 请求在同一集群上生成子协议作业)。
Parallel Processing Server Processes:需要提供一个逗号分隔的列表,表示每个服务器上可以同时执行的最大批次数。列表中的每个值必须与服务器列表中的条目对应。此参数中的条目数量必须等于服务器的数量。
例如:
- 5, 10, 8 (表示第一个服务器最多同时执行5个批次,第二个服务器最多同时执行10个批次,第三个服务器最多同时执行8个批次)
Parallel Processing Preserve Order:保留数据记录的初始顺序。
说明
上面的参数小果虽然已经详细的描述了,但是可能有一些内容小伙伴们还是不太明白,小果在这里进行一些说明:
姿态聚类
CDOCKER中的多样化顶部姿态聚类是一种用于对复合物结构进行聚类和筛选的技术。它在CDOCKER的分子对接过程中起到了重要的作用。
在分子对接中,CDOCKER会生成多个可能的复合物构象,每个构象都有不同的得分和位置。然而,有时候一些构象可能非常相似,只是微小的构象变化或者旋转而已。为了减少冗余和提高计算效率,多样化顶部姿态聚类被引入。
多样化顶部姿态聚类的主要思想是将所有的复合物构象根据其结构特征进行聚类,然后从每个聚类中选择一个代表性的构象作为结果。这样可以在保留重要结构信息的同时,减少构象数目,提高计算效率。
具体实现上,多样化顶部姿态聚类包括以下步骤:
- 根据CDOCKER产生的多个构象,使用分子对比算法计算它们之间的结构相似性。
- 应用聚类算法,如层次聚类或k-means聚类,将相似性高的构象归为同一类别。
- 从每个聚类中选择一个代表性的构象作为该聚类的顶部姿态。
- 最终,将所有选定的顶部姿态作为输出,供后续分析和评估使用。
通过多样化顶部姿态聚类,可以从大量的复合物构象中筛选出具有代表性和多样性的结构,从而帮助我们更好地理解蛋白质-小分子相互作用,并选择最有潜力的分子构象进行后续研究。
那么小伙伴们可能会想,如何从每个聚类中选择一个代表性的构象作为该聚类的顶部姿态呢?小果在这里给大家举几个例子方便大家思考:
- 能量最低:选择每个聚类中能量最低的构象作为代表性构象。这是常见的选择策略,因为能量最低的构象通常代表了该聚类中最稳定和最有可能出现的构象。
- 聚类质心:计算每个聚类的质心,将质心作为代表性构象。质心是指聚类中所有构象的平均值,代表了该聚类构象的中心趋势。选择质心作为代表性构象可以在一定程度上保持聚类中构象的平衡。
- 最具代表性:根据某种评价指标,选择每个聚类中最具代表性的构象。这可以基于一些特定的结构性质或关键特征来定义评价指标,例如分子间相互作用能、构象稳定性指标等。通过选择最具代表性的构象,可以更好地捕捉每个聚类的特征。
- 聚类密度最高:对于每个聚类,选择聚集在一起密度最高的构象作为代表性构象。这可以通过计算聚类中构象的密度分布或使用密度估计方法来实现。选择密度最高的构象作为代表性构象可以更好地表示聚类内部的分布情况。
姿态(pose)和构象(conformation)
在上文提到的姿态(pose)和构象(conformation)相信细心的小伙伴们已经注意到了,在英文中他们的单词不同,那么代表的含义自然也有所不同,小果在这里给大家解释一下:
- 姿态(Poses):在CDOCKER中,姿态通常指的是配体(ligand)与受体(receptor)之间的相对位置和方向。姿态表示了分子对接时配体与受体之间的空间关系和相互作用方式。每个姿态都描述了一个可能的配体-受体复合物构型,它包含了配体和受体之间的相互作用、键的角度和距离等信息。CDOCKER会对配体在受体表面上进行多个初始位置和方向的采样,生成一组不同的姿态。
- 构象(Conformations):在CDOCKER中,构象通常指的是配体或受体的单独空间结构。构象描述了分子在自由状态下的稳定几何结构,即分子的内部键角和空间排列。在分子对接过程中,CDOCKER会考虑配体的多个构象或受体的不同构象,以增加对配体-受体复合物的探索范围。CDOCKER通过采样不同的构象来搜索最佳的配体-受体结合方式,以找到稳定的复合物。
总的来说,姿态用于描述配体和受体之间的相对位置和方向,而构象用于描述配体或受体的空间结构。CDOCKER利用姿态和构象的组合来搜索最佳的配体-受体结合模式,并预测最有可能的分子对接复合物。
模拟退火
模拟退火是一种启发式的优化算法,灵感来自固体物质退火过程中的原子结构调整。它基于随机游走和温度控制来搜索配置空间,并以某个目标函数(如能量最小化)为优化目标。
在CDOCKER中,模拟退火通过以下步骤实现:
- 初始化小分子的初始位置和蛋白质的固定位置。
- 随机扰动小分子的位置,并计算得分(如能量评估)。
- 根据Metropolis准则,接受或拒绝新的构象。如果新构象具有更低的得分,它将被接受;否则,根据概率决定是否接受它。
- 逐步降低“温度”,即降低接受高能构象的概率。这有助于从高能区域跳出,并逐渐收敛到能量最低的构象。
- 重复步骤2-4直到满足停止准则,如达到最大迭代次数或能量变化小于一定阈值。
通过模拟退火算法,CDOCKER可以搜索并优化蛋白质-小分子复合物的构象空间,以寻找最稳定和具有高亲和力的结合模式。
高温分子动力学模拟
在CDOCKER中,高温分子动力学模拟(high-temperature molecular dynamics simulation)是一种通过模拟分子在高温条件下的运动来研究分子结构和动力学行为的方法。它可以用于模拟蛋白质、配体和受体等生物分子在高温环境中的行为,以及分子对接等相关问题。
以下是CDOCKER中高温分子动力学模拟的一般步骤:
- 输入准备:首先,需要准备要模拟的分子系统,包括配体和受体的原子坐标、拓扑文件和参数文件等。这些信息通常可以从分子数据库或其他计算工具中获取。
- 初始构象:根据需要,选择一个初始构象作为模拟开始的结构。可以使用实验结构、计算预测的构象或其他合适的构象作为初始结构。
- 系统参数:设置模拟所需的参数,如温度、压力和模拟时间步长等。在高温分子动力学模拟中,温度通常设置较高以模拟高温环境。
- 分子力场:选择适当的分子力场参数来描述分子间相互作用和键角等属性。CDOCKER通常使用CHARMM力场或其他经验参数。
- 分子动力学模拟:进行高温分子动力学模拟,通过求解牛顿运动方程来模拟分子的时间演化。CDOCKER使用经典的分子动力学方法,将分子中的原子粒子进行随机速度初始化,然后根据力场势能和牛顿定律进行模拟。
- 模拟过程:在模拟过程中,分子会根据模拟时间步长积分,原子之间会发生相互作用、键的伸缩和角度改变等。高温条件下,分子可能会发生结构扭曲、局部去折叠等现象。
- 结果分析:分析模拟结果,包括分子构象的变化、键的角度和距离的变化、能量变化以及其他感兴趣的参数。可以通过可视化工具或其他分析软件进行结果可视化和数据处理。
高温分子动力学模拟可以用于研究蛋白质的热稳定性、构象变化以及配体-受体相互作用等问题。它为理解分子的动态行为和功能提供了重要的信息,并对药物设计等研究具有一定的指导意义。
步数与温度
在上面的设置中,我们可以设置高温分子动力学模拟的步数和温度,那么他们会造成什么影响呢?小果现在就给大家讲解一下:
- 步数对时间演化的精度影响:较小的步长可以提高模拟的精度,因为它更细致地捕捉分子的运动。通过减小步长,可以更准确地模拟键角的振动、构象变化和反应路径等细节。然而,小步长也会增加计算的复杂性和计算资源的需求。
- 步数对计算时间的影响:步数的选择直接影响模拟所需的计算时间。较大的步数可以加快模拟速度,但可能会导致模拟结果的精度下降。相反,较小的步数可以提高模拟的精度,但会增加计算时间。因此,在实际应用中需要根据研究目的和可用资源权衡步数的选择。
- 温度对分子动力学行为的影响:在高温分子动力学模拟中,温度设置较高以模拟高温环境。温度的选择会直接影响分子的动力学行为和结构特征。较高的温度会增加分子的热运动和振动,导致分子结构的扭曲、局部去折叠等现象。较低的温度则会减少分子的热运动,使得体系更加稳定。因此,温度设置需要根据研究对象和研究目的进行选择。
静电效应
在随机结构生成过程中,静电效应可以对结构的形成和性质产生重要影响,比如:
- 形成稳定结构:静电相互作用是由带电粒子之间的库仑相互作用引起的。在随机结构生成过程中,静电效应可以在粒子之间产生吸引力或斥力,进而促使它们形成稳定的结构。通过控制静电相互作用的强度和范围,可以驱动粒子自组装形成特定的结构,例如晶体、聚合物等。
- 结构的形状和排列:静电相互作用可以影响粒子的排列方式和相互之间的距离。正电荷和负电荷之间的相互作用可以导致粒子形成有序排列或者形成特定的结构形状。例如,正负电荷交替排列的晶格可以产生离子晶体的结构。
- 电荷平衡:静电效应可以影响系统的电荷平衡。在随机结构生成过程中,如果粒子带有不同的电荷,静电相互作用可以促使电荷分布趋于平衡,减少电荷不平衡的情况。这可能导致结构的特定电荷分布和电荷密度。
- 物理性质的改变:静电相互作用可以影响结构的物理性质。例如,在随机结构生成过程中,静电相互作用可以影响材料的导电性、热导性、光学性质等。通过调控静电效应,可以改变材料的电子结构和能带结构,进而调节其电学、热学和光学性质。
总的来说,静电效应在随机结构生成过程中起着重要作用。它可以驱动粒子自组装形成有序结构,影响结构的形状和排列方式,并改变材料的物理性质。因此,在设计和控制随机结构生成过程时,需要考虑和利用静电效应的影响。
结语
小果提醒大家,上面图中的参数不是小果推荐大家设置的参数,只是小果某一次对接时的参数,大家要根据自己的需求设置自己的参数哦~
到这里为止,CDOCKER分子对接优化的全部参数详细信息以及如何设置和适用范围小果都给大家分享完毕啦,小伙伴们可能会觉得内容太多记不住,没有关系,小果特意给大家按功能进行了小标题分类,小伙伴们在看完之后大概记得住有哪些功能,在使用时再翻出来这篇教程查询就可以啦~
小果最后还要再次强调,参数根据自己的需求设置,没有统一标准,但每一项参数的设置都应该十分谨慎,因为会对后续研究造成很大的影响,对于没有把握的参数一定要谨慎查阅教程,不可以随便修改哦~
那么本次的分享就到这里结束啦,如果小伙伴们平时在生信分析的操作过程中遇到困难,欢迎大家使用小果开发的生信工具平台http://www.biocloudservice.com/home.html哦。我们下次再见啦,拜拜~