最近几年,生信分析的热度越来越高,其不用耗费大量的时间在实验室,不需要花费大量的资金,甚至仅仅通过公开数据库,只要你的想法新颖,同样可以做出满意的结果,发表不错的文章!但随着其热度的上涨,越来越多的研究者涌入此领域,生信分析逐渐呈现出较为饱和的状态,所以今天小果给大家带来的教程是刚刚兴起没多久的生信新蓝海-影像组学!
“影像组学”,我们把它们拆成“影像”和“组学”两个词来说。这里的“影像”通常指的就是放射影像,是这种方法研究的对象,目前大家选择最多的是CT、MR等影像。组学,英文为Omics,想必作为生信人的大家一点都不陌生吧,是目前生物和医药前沿研究领域最流行的后缀了,是高通量数据的代名词。所以,影像组学的研究对象是医学影像,研究方法是将影像中的所有信息提取出来再进行系统化分析,确切来说,影像组学是采用自动化算法从影像的感兴趣区(ROI)内提取出大量的特征信息作为研究对象,并进一步采用多样化的统计分析和数据挖掘方法,从大批量信息中提取和剥离出真正起作用的关键信息,最终用于疾病的辅助诊断、分类或分级。
听到这里,大家是不是有些熟悉呢,没错,其实从医学影像中提取出来的数据形式和我们的单细胞数据十分的类似,一张影像可能对应着上千条特征,所以所谓“组学”,真的名副其实。今天给大家手把手教学如何从医学影像中提取影像特征,感兴趣的小伙伴快来跟我一起实战练习吧。特别提示,本次操作需要通过Python进行,并且对电脑配置有一定需求,建议大家使用服务器运行,如果没有自己的服务器欢迎联系我们来租赁服务器哦~
- 环境配置
首先需要大家配置好Python的基础环境哦,这里小果用的是Python 3.7.13,
如果大家对Python配置有问题的话,也可以联系小果哦,小果会耐心给大家解答的,如果用我们的服务器更是会事半功倍的哦~
- 需要的Python软件包
#安装软件包,命令行模式下,任意位置输入:
pip install SimpleITK
pip install tqdm
pip install pyradiomics
#加载软件包
import os
import SimpleITK as sitk
from tqdm import tqdm
from pandas.core.frame import DataFrame
import time
import pandas as pd
import radiomics
from radiomics import featureextractor
3.加载数据并转换数据
可以是直接使用的数据是nii.gz或者nii格式的数据,很多公开数据库是直接以此格式为准的,但如果我们拿到的是自己的数据,往往是DICOM格式的,如下图(下图该脑部CT序列共有247张图像),一个患者可能包含一个或者多个连续序列的图像,因为一次检查获得的是该器官全方位的断层图像。
所以我们需要整理成我们可用的格式,这里我们就用到了simpleITK这个库或者叫软件包,SimpleITK可以很便捷的读取医学图像的格式,如二维的.dicom格式以及三维图像的.nii格式,并对图像中的诸如体素,方向等等基本信息进行处理。SimpleITK包含了R语言,Java,Python等等常见语言的接口,本次给大家带来的是在Python中的应用。这里也给大家放上SimpleITK的官方文档链接:https://insightsoftwareconsortium.github.io/SimpleITK-Notebooks/。
#遍历包含患者DICOM图像的文件
path1 = os.listdir(‘pic/’)
#利用读取器得到目录下所包含的序列ID
reader = sitk.ImageSeriesReader()
series_IDs = reader.GetGDCMSeriesIDs(dir_path)
# 根据一个单张的DICOM文件,读取这个series的metedata,即可以获取这个序列的描述符,之后可将不同检查类别的图像序列给分类出来。
形成nii.gz的格式,如下图:

此外,大家可能还会碰到其他格式的医学图像,如nrrd,大家也可以用此代码哦simpleITK对于常见医学图像的各种格式都可以相互转换的。
可能有的小伙伴会问了,这种医学图像特别是nii这种格式的图像,怎么才能打开呢,能看看到底是什么样。这里给大家推荐一个软件:ITK-SNAP(http://www.itksnap.org/pmwiki/pmwiki.php)

ITK-SNAP是一款免费开源的医学图像可视化和分析软件。它允许用户加载和操作各种医学图像数据,包括MRI、CT和PET扫描,并执行分割、配准和标记等任务。此外,其具有用户友好的界面,支持各种文件格式。下图是其界面示意图:
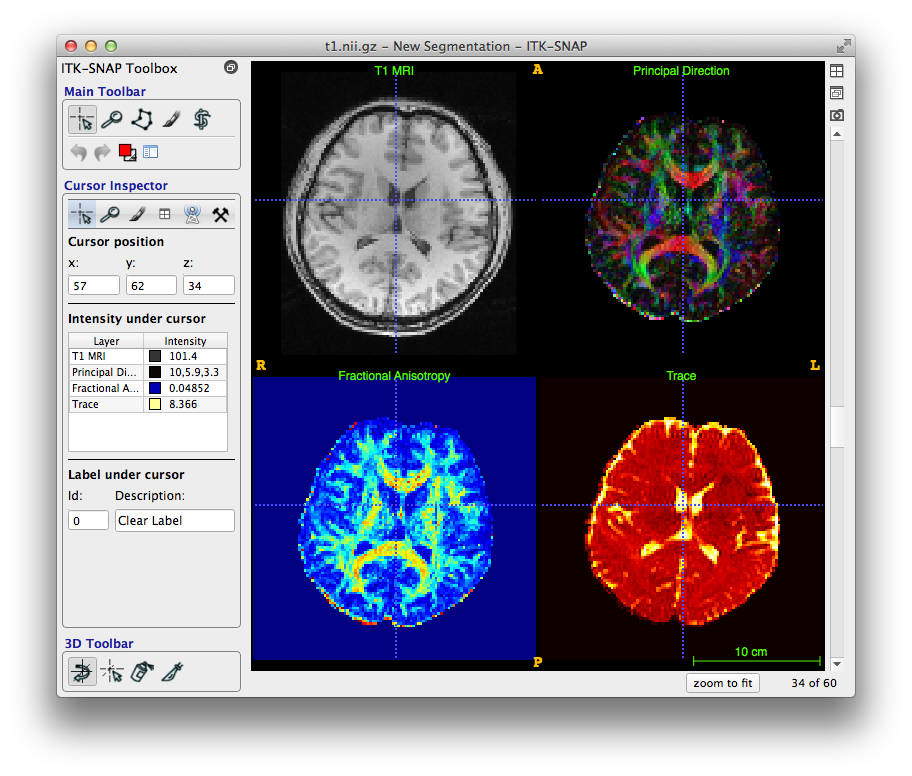
因为影像组学往往涉及到医学图像感兴趣区域(ROI)的勾画,而ITK-SNAP是非常常用的一款软件,简单易上手,是十分重要的工具。p.s,如果大家对这款软件感兴趣的话,我们也会专门出一期教程哦~
4.特征提取并保存为可读形式
#加载pyradiomics库
from radiomics import featureextractor
#设置提取器
extractor = featureextractor.RadiomicsFeatureExtractor(para)

大家最终获得特征被保存为xlsx的格式,可以直接用Excel打开查看,可以发现两个序列的图像最终获得了上万条的特征,其中大家需要注意的是,前几列是关于图像的版本和像素坐标等我们不需要的信息,后面数值型的数据才是我们需要的特征,并且每一个特征都有对应的描述,如此多的特征,现在大家应该知道为什么会被称为“影像组学”了吧。
小果拿一个患者的示例数据,给大家手把手完整演示了如何提取影像组学特征,大家学会了之后,可以在公开数据库或者手头里完整的数据进行尝试哦,提取出来的这些特征大家就可以根据常规的步骤,进行特征筛选,构建模型,甚至可以尝试和其他组学数据联合建模,说不定有意外惊喜哦!
如果需要完整的代码可以点击付费获取哦!今天小果的分享就到这里,如果小伙伴有其他数据的分析需求,可以尝试本公司新开发的生信分析小工具云品台,里面有成熟的代码,只要输入合适的数据就可以直接出想要的图呢,链接:http://www.biocloudservice.com/home.html