各位小伙伴,大家好啊,很高兴小果又和大家见面了,其实有一个问题一直困扰着我:就像大家一直用的功能强大的ggplot2、简单的热图工具pheatmap还有画circle图的R包circlize,他们虽然能展示很多信息,但形式单一,仅能通过颜色的调节来改变整个图的格调,那会不会有一款R包能以一种立体的形式,来展示咱们获得的三个基因的相互作用或者是三个PCA成分所占比例呢?答案是:当然啦。今天小果给大家带来的这个R包–scatterplot3d就能实现3个变量的相互作用哦。
scatterplot3d是R语言中用于创建3D散点图的包。这个包提供了一种可视化数据的方式,能帮助大家在三个维度中同时探索数据点的分布与关系哦。那么小果在这里呢,给大家列举了一些scatterplot3d包的一些应用场景:首先是使用scatterplot3d包,大家可以将三维数据以散点图的形式呈现,从而更全面的了解数据点在三个维度上的分布情况。其次大家都知道三维散点图有助于观察不同变量之间的关系,各位小伙伴可以将不同变量映射到三维空间的坐标轴上哦,并通过观察散点的分布情况就能了解他们之间的趋势和关联性啦。然后,大家通过观察三维散点图,还有益于发现数据中的异常值或离群点,因为他们可能在三维空间中呈现出独特的模式,这肯定是有利于我们对数据的筛选哦。该包还有益于观察数据点是否在三个维度上聚集成群或类别,这对我们的平行试验或分组讨论很有用哦。最后,如果大家有多个自变量和一个因变量,也可以使用三维散点图来可视化他们之间的关系,进而了解变量对因变量的影响哦。
那根据以上介绍,相信大家也能或多或少的了解了不同数据之间具有着不同关系,而针对基因组或转录组等大数据组的信息的提取,还是推荐大家使用服务器命令行的形式进行,因为这会更加的方便快捷,如果没有自己的服务器欢迎联系我们进行服务器租赁哦~
说了这么多,让我们这就开始scatterplot3d的学习吧!
#安装R包及调用
install.packages(‘scatterplot3d’)
library(scatterplot3d)
library(RColorBrewer)
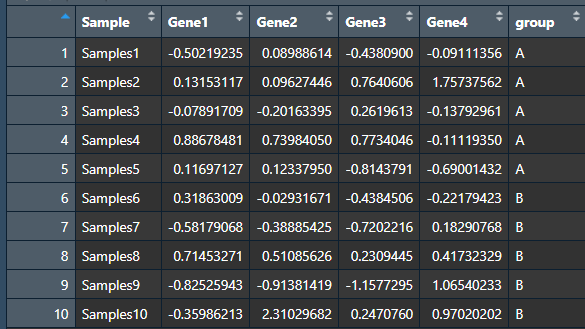
#读入数据
data <- read.table(‘输入数据.txt’,sep = ‘\t’,row.names = 1,header = T)
data
#设置颜色,因为每个基因设置了10个数据,因此选取两个色系
Set1 <- brewer.pal(n=6,name = ‘Set1’)
Set2 <- brewer.pal(n=6,name = ‘Set2’)
Set <- unique(c(Set2,Set1))

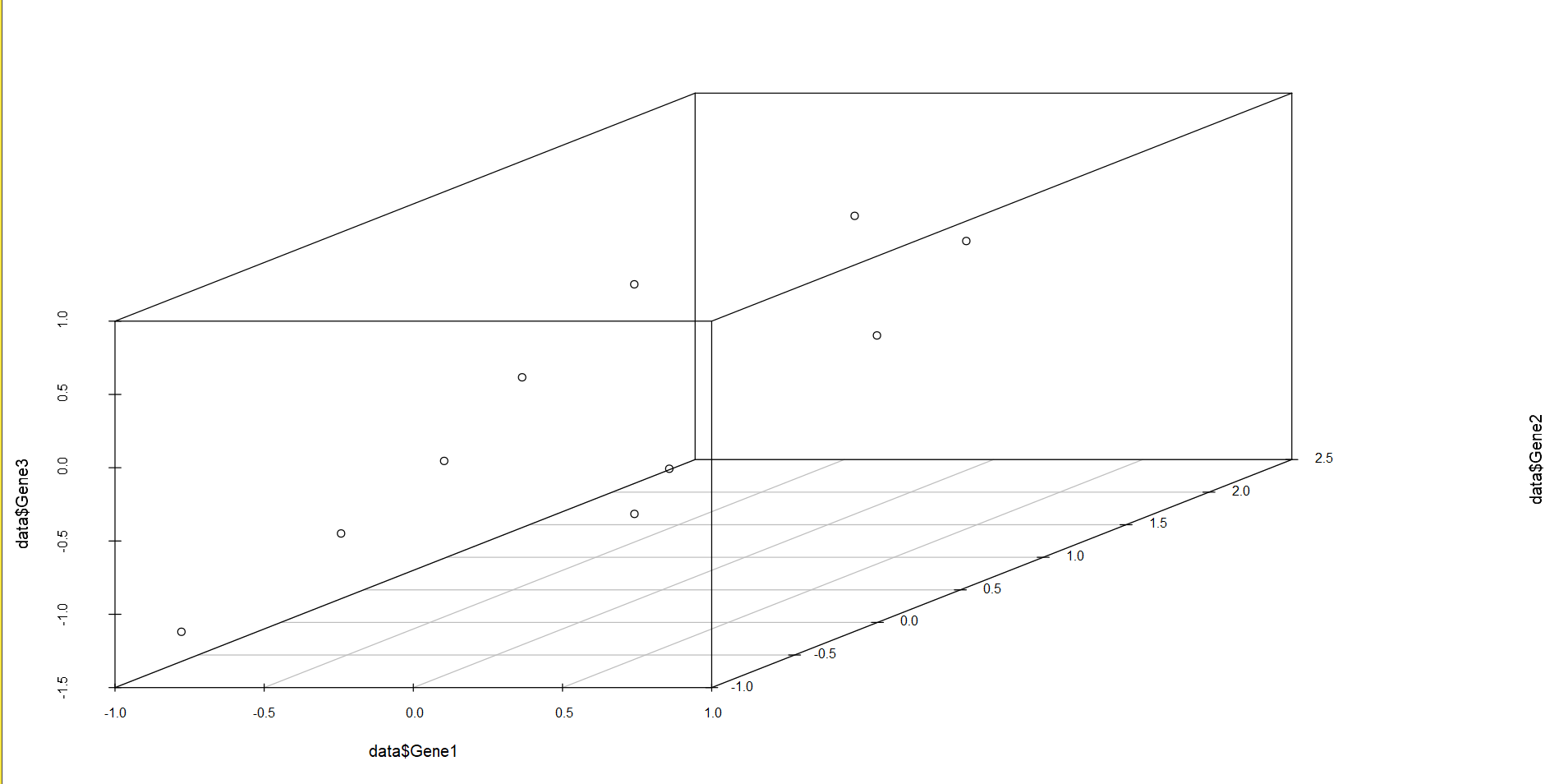
#简单出图
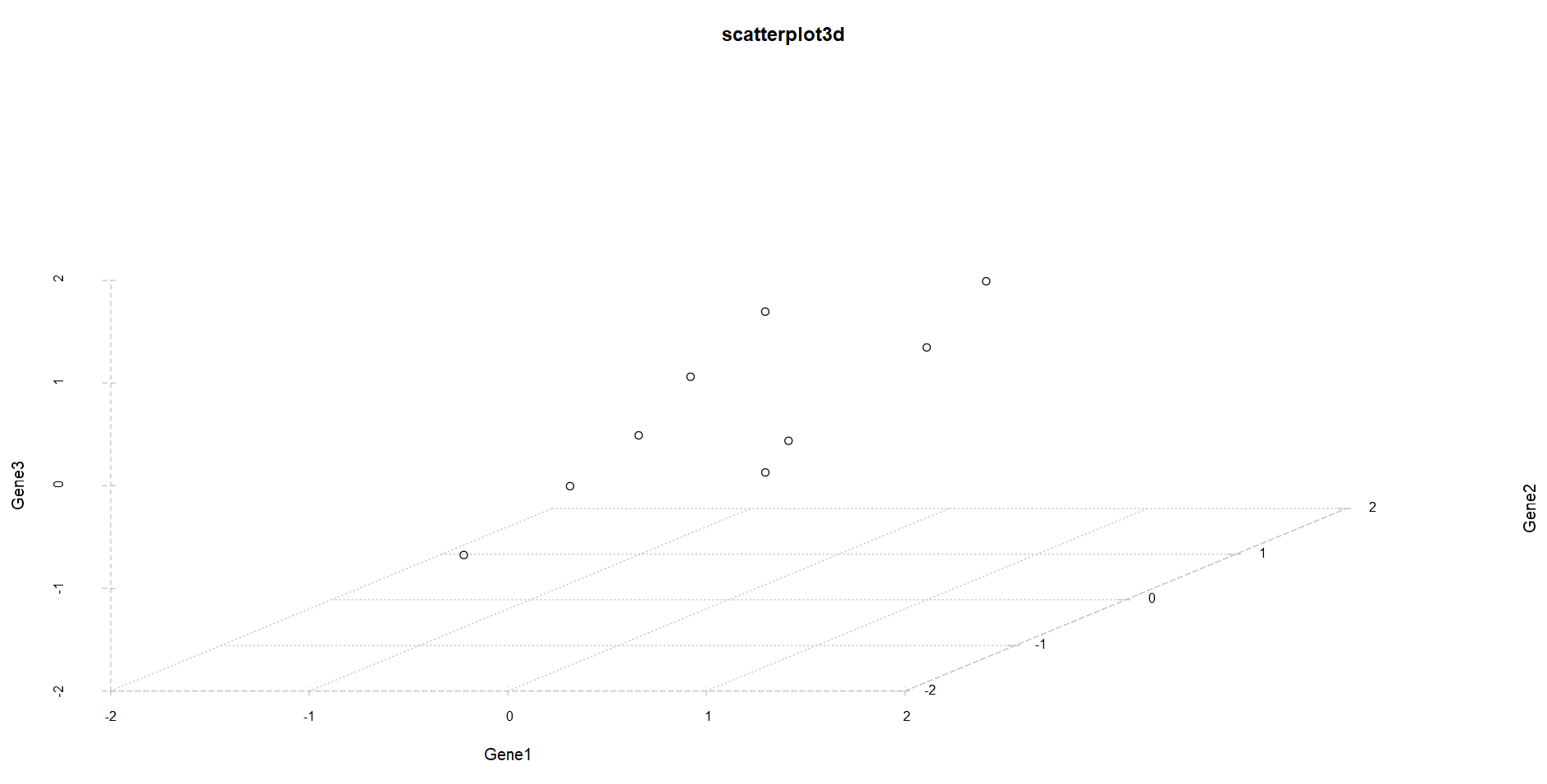
P1 <- scatterplot3d(x = data$Gene1,y = data$Gene2,z = data$Gene3)
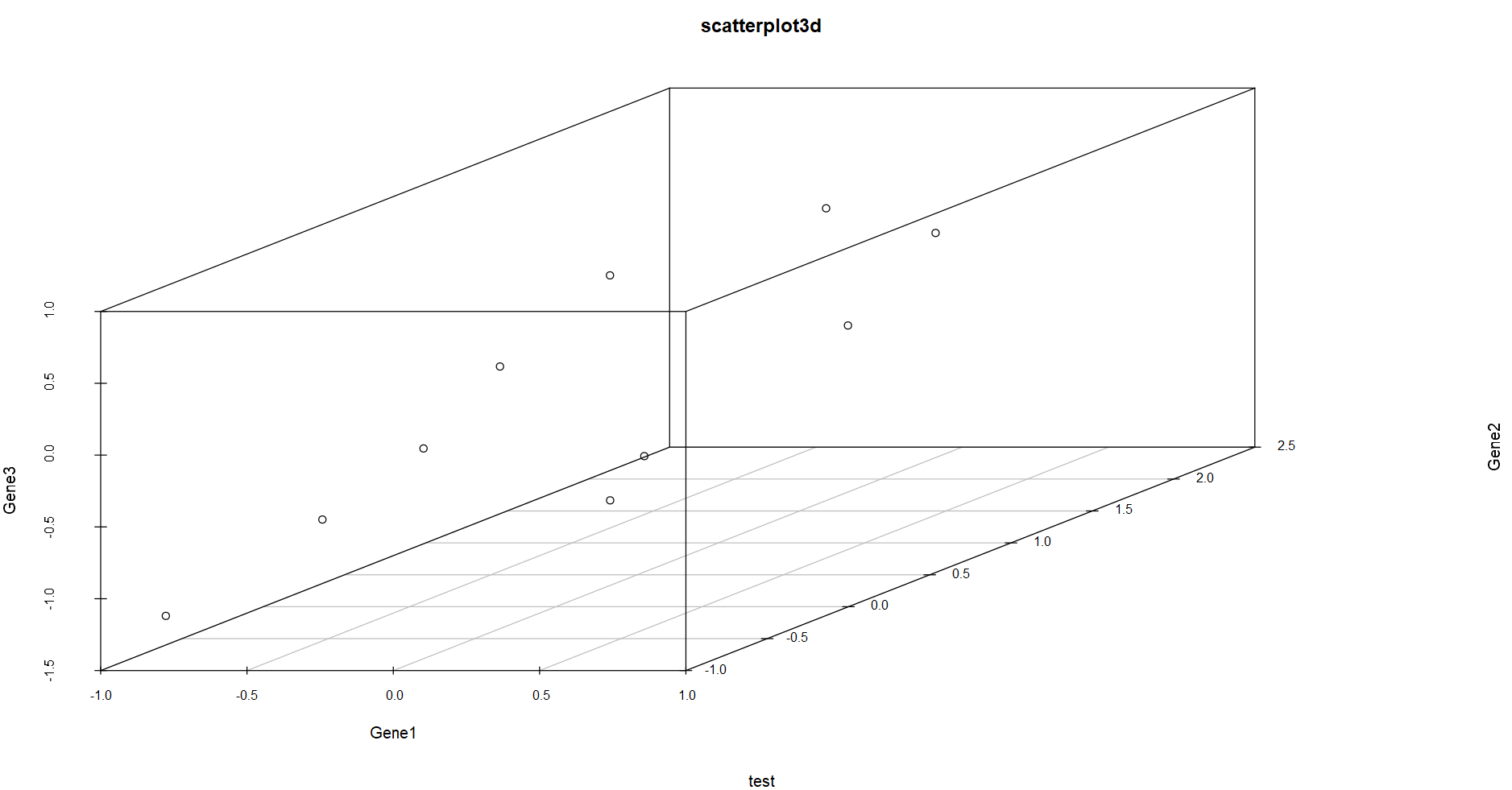
#对轴标题的调节
P2 <- scatterplot3d(x = data$Gene1,y = data$Gene2,z = data$Gene3,
xlab = ‘Gene1’,ylab = ‘Gene2’,zlab = ‘Gene3’,
main = ‘scatterplot3d’,sub = ‘test’)
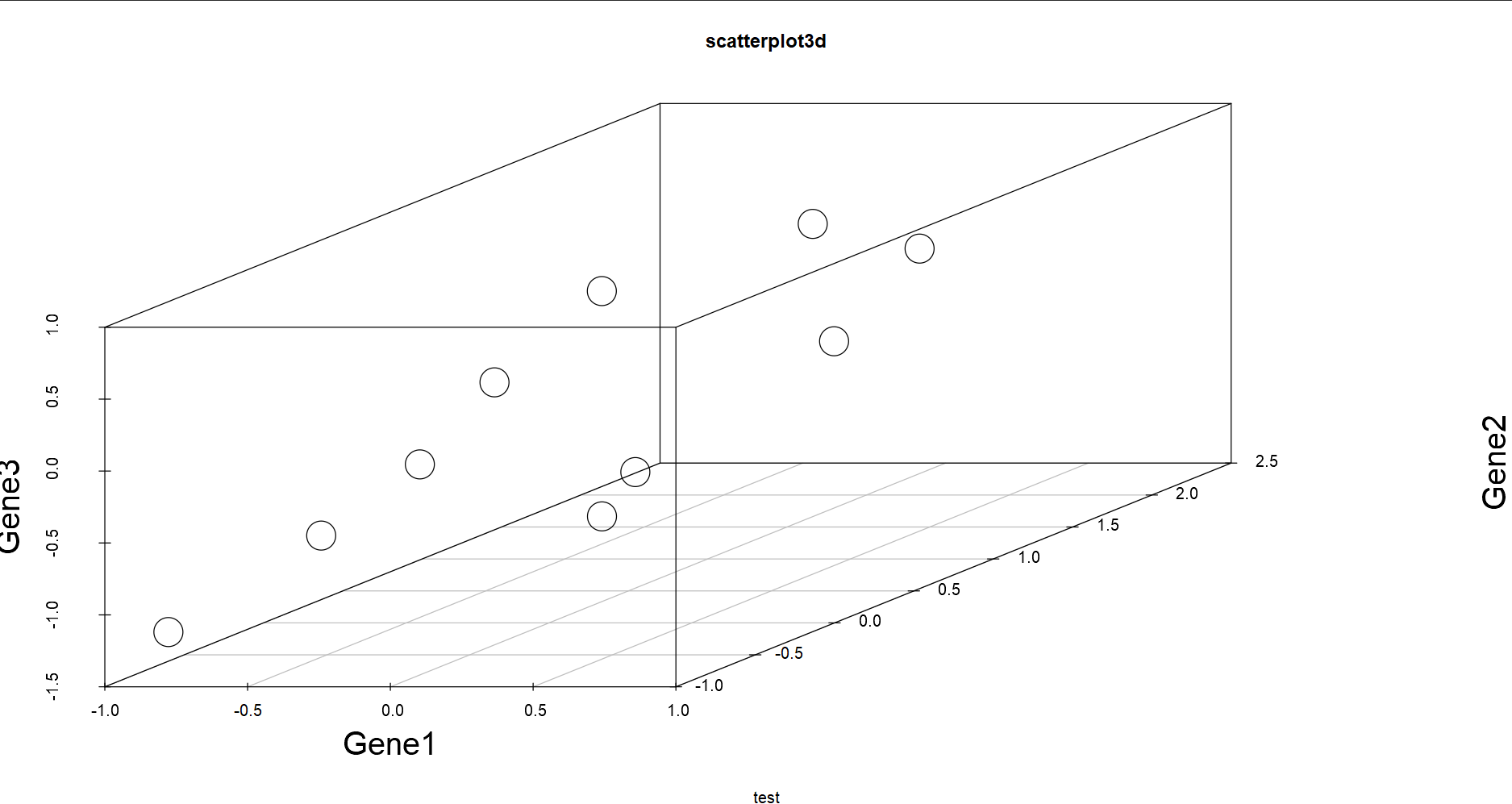
#对字体大小进行调节
P3 <- scatterplot3d(x = data$Gene1,y = data$Gene2,z = data$Gene3,
xlab = ‘Gene1’,ylab = ‘Gene2’,zlab = ‘Gene3’,
main = ‘scatterplot3d’,sub = ‘test’,
cex.lab = 2,cex.axis = 1,cex.symbols = 4, #分别是轴标题,轴标签及点的大小
font.lab = 45)
#对线进行调节
P4 <- scatterplot3d(x = data$Gene1,y = data$Gene2,z = data$Gene3,
xlab = ‘Gene1’,ylab = ‘Gene2’,zlab = ‘Gene3’,main = ‘scatterplot3d’,sub = ‘test’,
grid = T, #底面是否有横线
box = F, #是否添加其余边
#x.ticklabs = colnames(data), #用于指定刻度
lty.grid = 3, #底面线的类型
lty.axis = 2, #坐标轴线的类型
ylim = c(-2,2),xlim = c(-2,2),zlim = c(-2,2), #各坐标轴的长度
col.grid = ‘grey’,col.axis = ‘grey’ #分别对底线/坐标轴的颜色进行改变
)
#给定形状参数
shape <- c(16,17)
#其余小函数
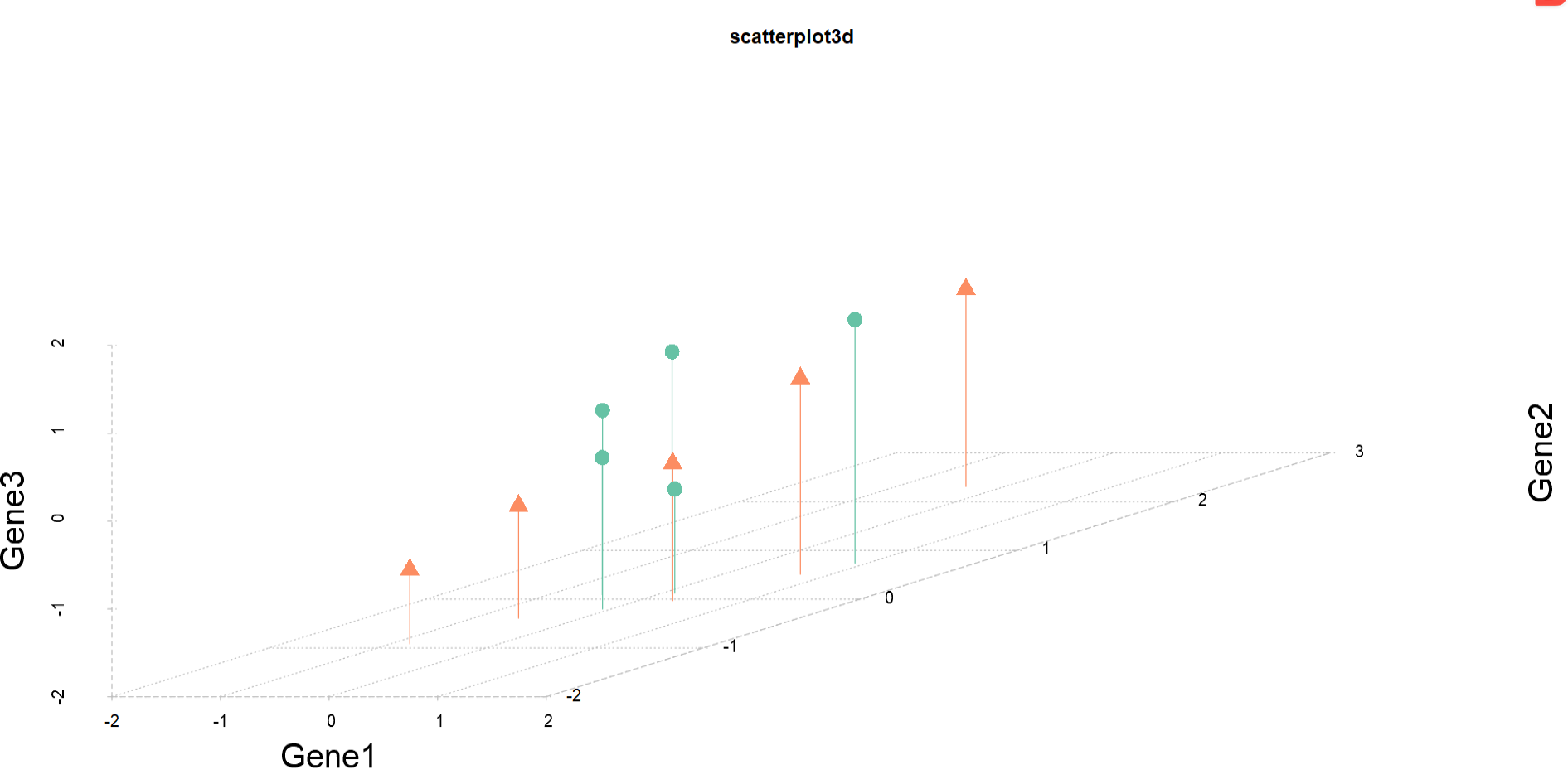
P5 <- scatterplot3d(x = data$Gene1,y = data$Gene2,z = data$Gene3,
xlab = ‘Gene1’,ylab = ‘Gene2’,zlab = ‘Gene3’,main = ‘scatterplot3d’,sub = ‘test’,
pch = shape[factor(data$group)],color = Set[factor(data$group)], #点的形状及颜色
cex.lab = 2,cex.axis = 1,cex.symbols = 2, #分别是轴标题,轴标签及点的大小
ylim = c(-2,2.5),xlim = c(-2,2),zlim = c(-2,2), #各坐标轴的长度
scale.y = 2, #y轴单个方框的宽度
angle = 25, #长方体整体角度
lab.z = c(3,5),grid = T, #底面是否有横线
box = F, #是否添加其余边框
#表示点的状态,‘p’是只有点,‘l’是只有线,‘h’是既有点到底面的垂线,‘b’是点与线互相连接
type = ‘h’,
#x.ticklabs = colnames(data), #用于指定刻度
lty.grid = 3, #底面线的类型
lty.axis = 2, #坐标轴线的类型
col.grid = ‘grey’,col.axis = ‘grey’,col.lab = ‘black’ #分别对底线/坐标轴的颜色进行改变
)
#添加图例和文本信息:
legend(“topright”, legend = c(“A”, “B”),
col = Set, pch = c(16, 17))
#前面的数字是根据底线的条数来决定的哦,但是中间的数的确是根据z轴坐标而定
text(1.1,-0.2,1,labels = ‘sample00’) #当然是可以改变颜色和大小的啊
text(1,0.5,-0.3,labels = ‘samplexx’,col = ‘red4’,cex = 1.5)
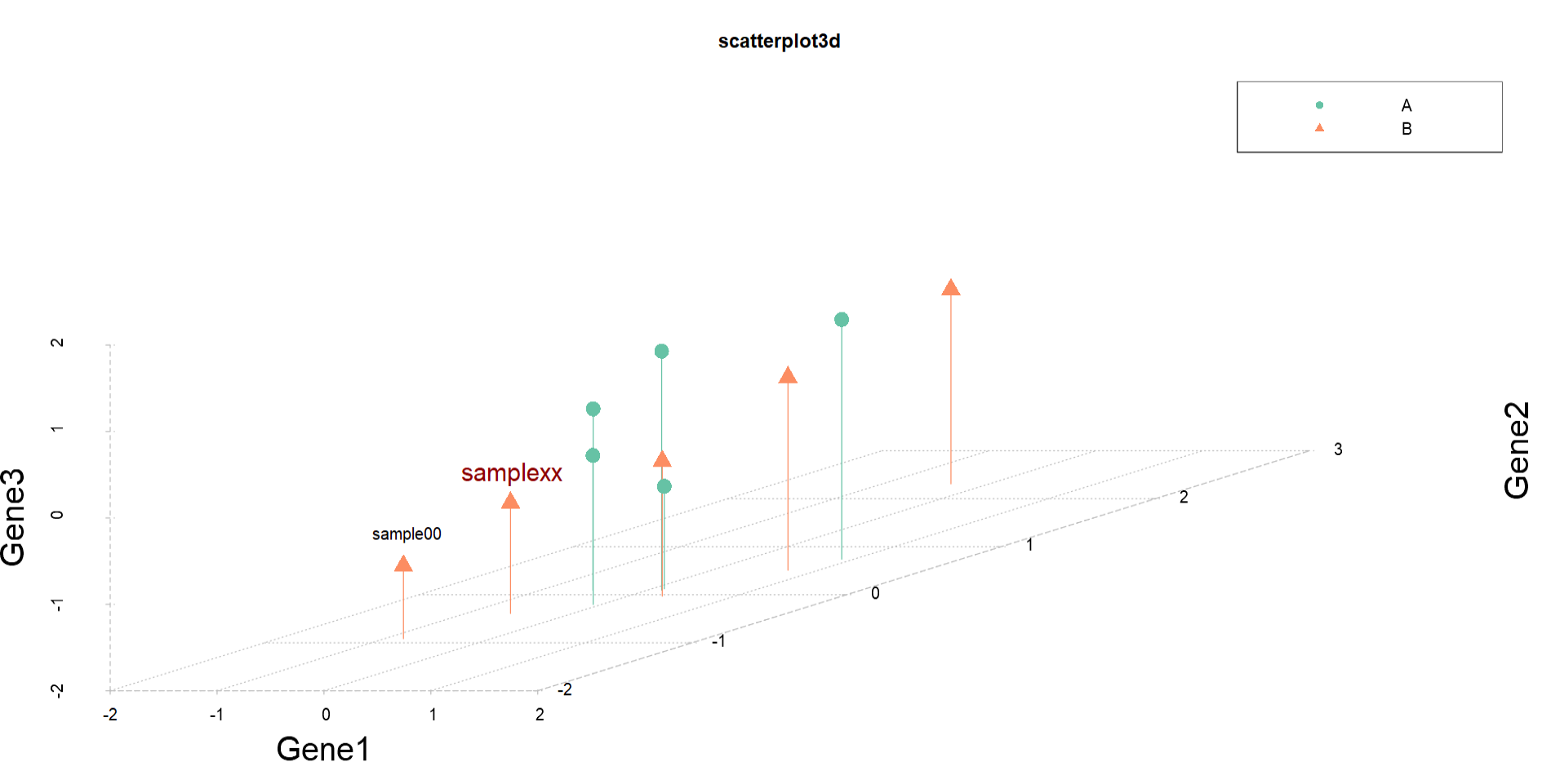
好啦,这就是我们今天的全部代码代练哦,不知道小伙伴们有没有加深对三维散点图绘制的印象呢,不知道大家是否能熟练应用这个R包了呢,赶快和小果一起练习吧!那既然这个包能展示多维数据,他到底对我们生物信息学有什么帮助呢,下面是小果对根据自己的研究方向及查阅资料所获得的生信中的适应范围:
基因表达与生物特性的关联分析:在基因组学和转录组学的研究中可以使用该包可视化基因表达水平与生物特性(如药物反应、疾病发展等)之间的关系。将基因表达数据与生物特性数据(如临床参数,实验测量等)以三维形式展示,有助于识别特定基因或基因组模式与生物学现象之间的相关性。
药物筛选和效果分析:在药物研发过程中,可使用该包可视化药物剂量、效力和治疗效果之间的关系。将药物剂量、细胞存活率和时间点等因素以三维图展示,有助于评估药物在不同条件下的效果,知道药物筛选及优化。
蛋白质互作网络分析:在蛋白质组学研究中,该包可对不同蛋白质之间的关联进行可视化。将蛋白质相互作用的强度、结合亲和力和功能相关性等因素以三维图形呈现,有助于理解蛋白质网络的复杂性和功能特性。
细胞信号传导途径研究:在研究细胞信号传导途径时,该包可用于显示不同信号分子的浓度、激活状态和时间依赖性。这有助于了解细胞信号传导通路中分子之间的相互作用和动态变化。
看到了吧,就因为这个包在生信领域有很频繁的使用率,也有很多的应用范围,所以小果才对它推崇备至哦!它在可视化和分析复杂的多变量生物数据中真是很方便很强大呢!通过这种方式,各位小伙伴能够更直观地观察和理解生物系统中的关系、趋势和模式哦,相信也能为实验设计、数据解释和结果分析提供更深入的洞察力。
这回各位小伙伴们知道这个包的强大之处了吧,希望大家通过自己的练习能够加深对该包的认识,能够熟练应用该包。如果大家在实操过程中遇到什么困难,也可以联系效果我,赶快加入我们吧!!虽然今天介绍的R包较为简单,可其应用范围可谓是多种多样,但的确有些数据也不适应于该包,且该包所可视化的样式可能略有古板,若各位小伙伴想探索更细致、更精美的可视化图表,可以来我们的云生信平台,里面有成熟的代码,只需要你输入数据就可以直接出图,点击下方链接尝试吧!
http://www.biocloudservice.com/home.html