富集分析是我们在做生信分析时经常用到的一种方法,但是富集分析有很多种类型,在进行数据分析的时候应该如何选择呢?这个问题困扰了小果一段时间,经过小果不懈的努力,终于理清了它们之间的关系,小果还特地找了一些代码来进行区分,如果小伙伴们也对如何区分富集分析感兴趣,那就接着看下去吧!
首先,富集分析是用于分析一组基因或物质在某些功能节点或通路上是否比随机水平更多地出现,从而揭示它们的生物学意义的一种方法。可以帮助研究者理解基因表达、代谢、蛋白质相互作用等数据背后的生物学机制。具体来说:
GO分析是一种基于基因本体论(Gene Ontology,GO)的功能注释和富集分析方法,是一个用于描述基因和蛋白质功能的标准化语言,包括三个方面:分子功能(Molecular Function),生物过程(Biological Process)和细胞组件(Cellular Component)。分析的目的是找出一个基因集合中与某些功能或过程相关的基因,从而揭示基因集合的生物学意义。
KEGG分析是一种基于KEGG数据库的功能注释和富集分析方法。KEGG数据库是一个整合了基因组、化学和系统功能信息的数据库,包括代谢通路、分层分类、基因、基因组等。KEGG分析的目的是找出一个基因集合中与某些代谢通路相关的基因,从而揭示基因集合的生物学意义。KEGG和GO的区别主要体现在,GO是一条条的线路(GO term),每一个线路里面有自己的基因集,线路彼此之间是没有任何联系的,而KEGG是网状的,不仅有基因集,还定义了基因和代谢物之间的复杂的相互关系。
GSEA分析是一种基于基因集的富集分析方法,可以评估一个预定义的基因集在两种生物状态之间是否有显著的表达差异。与GO,KEGG不同的是,GO和KEGG计算的p值是需要人为划定阈值的,而GSEA不需要划定阈值,GSEA将输入数据与GSEA中数据库比对,对基因打分。
SSGSEA是一种用于评估每个样本中特定基因集的相对富集程度的方法。在得到每个样本和基因集的富集得分后,可以用于免疫细胞浸润分析,基因通路分析,以及其他基于基因集的生物学研究。SSGSEA是GSEA方法的延申,是针对单个样本无法做GSEA而设计的。
由于GO分析和KEGG分析小果已经介绍过了,所以今天小果将带着大家一起来做一下GSEA分析与SSGSEA分析~本次分析使用的数据来自于一篇有关药物的靶向预测的文章,目的是找到与抑制剂TAK-243相关的信号通路。本次复现所需的代码与数据小果都已经帮大家整理好了,关注公众号后即可获取,租赁服务器还可以获得专属生信分析思路定制哦。
接下来让我们正式开始吧!
Step 1 GSEA分析
首先先根据 GSEA 预排序分析的显着性和相关性生成基因排序,如下图所示。
#根据 GSEA 预排序分析的显着性和相关性生成基因排序
dir.create(“/results/rnks”)
SCLC_rnk<-GenerateGSEA(bt_EC50, “/results/rnks/SCLC1.rnk”, retRNK=T, plotRNK=F)
SCLC_rnk2<-GenerateGSEA(bt_EC502, “/results/rnks/SCLC2.rnk”, retRNK=T, plotRNK=F)
YAP1_rnk<-GenerateGSEA(bt_EC50_YAP, “/results/rnks/YAP1_1.rnk”, retRNK=T, plotRNK=F)
YAP1_rnk2<-GenerateGSEA(bt_EC50_YAP2, “/results/rnks/YAP1_2.rnk”, retRNK=T, plotRNK=F)
ASCL1_rnk<-GenerateGSEA(bt_EC50_ASCL1, “/results/rnks/ASCL1.rnk”, retRNK=T, plotRNK=F)
ND1_rnk<-GenerateGSEA(bt_EC50_ND1, “/results/rnks/NEUROD1.rnk”, retRNK=T, plotRNK=F)
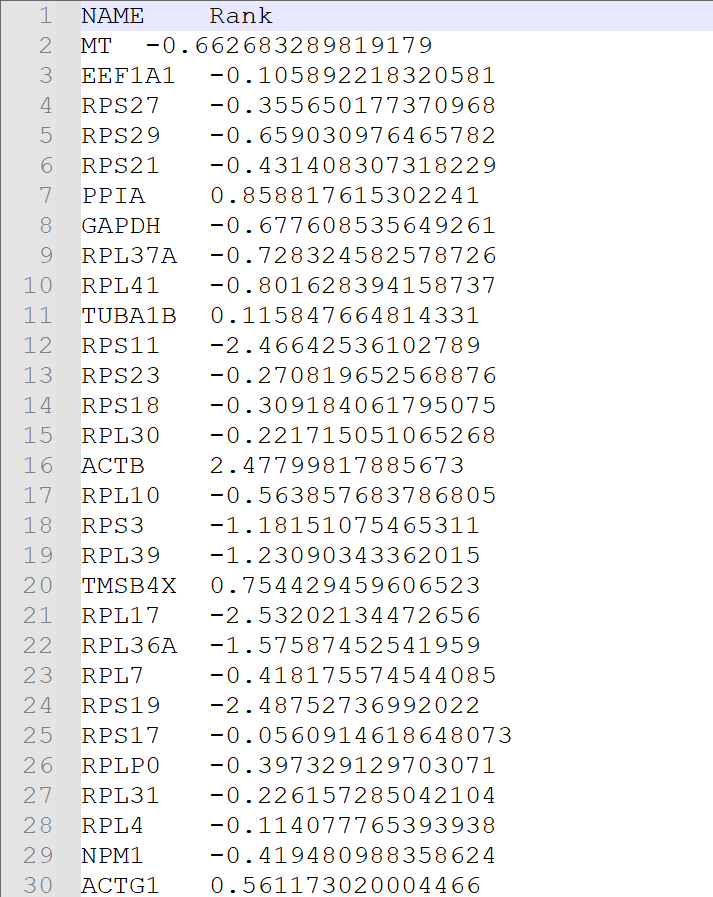
SCLC1.rnk
文件中对基因以及排序进行了明确的整理,有助于我们后续寻找与相应的靶点。在排序之后,设置基因集.
#设置基因集
gmt<-“/data/GSEA_gmt.gmt”
接着进行基因集富集分析GSEA,绘制排序前沿的抵抗基因和敏感基因的路径,如下图所示,结果图中的红色和蓝色分别表示两种生物状态的基因集富集得分(Enrichment Score)。红色表示正的富集得分,说明该基因集在差异表达基因的顶端富集,与第一种状态相关。蓝色表示负的富集得分,说明该基因集在差异表达基因的底端富集,与第二种状态相关。富集得分的绝对值越大,表示富集程度越高,也就是基因集与生物状态的相关性越强。
#使用 Bader Lab 途径的数据对每个预排序分析进行基因集富集分析 (GSEA)
pdf(“/results/TAK243_GSEA_.pdf”)
SCLC_gsea<-GSEA(SCLC_rnk, gmt, pval=1)
SCLC_gsea2<-GSEA(SCLC_rnk2, gmt, pval=1)
YAP1_gsea<-GSEA(YAP1_rnk, gmt, pval=1)
YAP1_gsea2<-GSEA(YAP1_rnk2, gmt, pval=1)
ASCL1_gsea<-GSEA(ASCL1_rnk, gmt, pval=1)
ND1_gsea<-GSEA(ND1_rnk, gmt, pval=1)
dev.off()
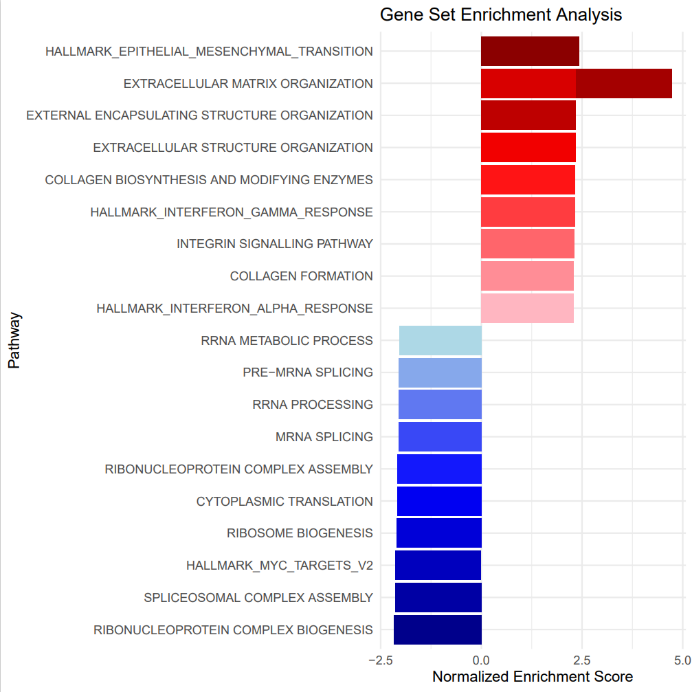
TAK243_GSEA_
然后绘制排序前沿的抵抗基因和敏感基因的路径,如图所示,是MYC_TARGETS(敏感)和AEROBIC_RESPIRATION(抵抗)的绘图。我们通过GSEA选择前沿基因,从而识别SCLC细胞系以及亚型中的敏感(左)和抵抗(右)途径的代表示例。前沿基因(突出显示的橙色条)可以用作TAK-243反应的潜在生物标志物。
#绘制排序前沿的抵抗基因和敏感基因的路径
png(“/results/enPlot_AerobicRespiration.png”)
enPlot(ASCL1_gsea[which(ASCL1_gsea$pathway %in% “AEROBIC RESPIRATION%GOBP%GO:0009060”),], rnk=ASCL1_rnk, gmt=gmt)
dev.off()
png(“/results/enPlot_MYCTargets.png”)
enPlot(SCLC_gsea2[which(SCLC_gsea2$pathway %in% “HALLMARK_MYC_TARGETS_V1%MSIGDB_C2%HALLMARK_MYC_TARGETS_V1”),], rnk=SCLC_rnk2, gmt=gmt)
dev.off()

enPlot_AerobicRespiration

enPlot_MYCTargets
对GSEA的结果进行比较,如图所示,是一系列的基因集富集分析(GSEA)的比较图,可以显示不同的生物状态之间的基因集富集得分(Enrichment Score)的相关性。横轴和纵轴分别表示两个生物状态的基因集富集得分,每个点表示一个基因集,颜色表示该基因集的p值或q值,大小表示该基因集的基因数目,可以发现不同生物状态之间的共同或特异的功能或通路。
结果图上方的trend是指两个生物状态的基因集富集得分(Enrichment Score)之间的线性相关性。trend的值越接近1或-1,表示两个生物状态的基因集富集得分越正相关或负相关,即越有相同或相反的功能或通路。trend的值越接近0,表示两个生物状态的基因集富集得分越无关。
#GSEA 结果的比较
pdf(“/results/GSEA_comparisons.pdf”)
comp1<-compGSEA(SCLC_gsea, YAP1_gsea, title=”SCLC vs YAP1″, xlab=”SCLC”, ylab=”YAP1″, p=0.05)
comp2<-compGSEA(SCLC_gsea, ASCL1_gsea, title=”SCLC vs ASCL1″, xlab=”SCLC”, ylab=”ASCL1″, p=0.05)
comp3<-compGSEA(SCLC_gsea, ND1_gsea, title=”SCLC vs NEUROD1″, xlab=”SCLC”, ylab=”NEUROD1″, p=0.05)
comp4<-compGSEA(YAP1_gsea, ASCL1_gsea, title=”YAP1 vs ASCL1″, xlab=”YAP1″, ylab=”ASCL1″, p=0.05)
comp5<-compGSEA(YAP1_gsea, ND1_gsea, title=”YAP1 vs NEUROD1″, xlab=”YAP1″, ylab=”NEUROD1″, p=0.05)
comp6<-compGSEA(ASCL1_gsea, ND1_gsea, title=”ASCL1 vs NEUROD1″, xlab=”ASCL1″, ylab=”NEUROD1″, p=0.05)
comp7<-compGSEA(SCLC_gsea2, YAP1_gsea2, title=”SCLC vs YAP1″, xlab=”SCLC”, ylab=”YAP1″, p=0.05)
comp8<-compGSEA(SCLC_gsea2, ASCL1_gsea, title=”SCLC vs ASCL1″, xlab=”SCLC”, ylab=”ASCL1″, p=0.05)
comp9<-compGSEA(SCLC_gsea2, ND1_gsea, title=”SCLC vs NEUROD1″, xlab=”SCLC”, ylab=”NEUROD1″, p=0.05)
comp10<-compGSEA(YAP1_gsea2, ASCL1_gsea, title=”YAP1 vs ASCL1″, xlab=”YAP1″, ylab=”ASCL1″, p=0.05)
comp11<-compGSEA(YAP1_gsea2, ND1_gsea, title=”YAP1 vs NEUROD1″, xlab=”YAP1″, ylab=”NEUROD1″, p=0.05)
comp12<-compGSEA(SCLC_gsea, SCLC_gsea2, xlab=”SCLC”, ylab=”SCLC without NCIH196″, p=0.05)
comp13<-compGSEA(YAP1_gsea, YAP1_gsea2, xlab=”YAP1″, ylab=”YAP1 without NCIH196″, p=0.05)
comp13<-comp13[order(comp13$x, decreasing=T),]
dev.off()


GSEA_comparisons.pdf
根据比较的结果,查找并比对反相关的路径,并从每次分析中提取前沿基因。
#查找并比对反相关路径,并从每次分析中提取前沿基因
YAP1_res_bioM2<-tryCatch(gsea_gmt(rownames(subset(comp7, Direction==”Negative” & y>0)), gmt=YAP1_gsea2, leadingEdge = T), error=function(x) return(list(c(“”))))
YAP1_sen_bioM2<-tryCatch(gsea_gmt(rownames(subset(comp7, Direction==”Negative” & y<0)), gmt=YAP1_gsea2, leadingEdge=T), error=function(x) return(list(c(“”))))
try(ASCL1_res_terms<-plotVenn(list(ASCL1_1=rownames(subset(comp2, Direction==”Negative” & y>0)),
ASCL1_2=rownames(subset(comp8, Direction==”Negative” & y>0))), retVals=T)$Only_1)
ASCL1_res_bioM<-tryCatch(gsea_gmt(ASCL1_res_terms, gmt=ASCL1_gsea, leadingEdge = T), error=function(x) return(list(c(“”))))
try(ND1_res_terms<-plotVenn(list(ND1_1=rownames(subset(comp3, Direction==”Negative” & y>0)),
ND1_2=rownames(subset(comp9, Direction==”Negative” & y>0))), retVals=T)$Only_1)
try(ND1_sen_terms<-plotVenn(list(ND1_1=rownames(subset(comp3, Direction==”Negative” & y<0)),
ND1_2=rownames(subset(comp9, Direction==”Negative” & y<0))), retVals=T)$Only_2)
ND1_res_bioM<-tryCatch(gsea_gmt(ND1_res_terms, ND1_gsea, leadingEdge=T), error=function(x) return(list(c(“”))))
ND1_sen_bioM<-tryCatch(gsea_gmt(ND1_sen_terms, ND1_gsea, leadingEdge=T), error=function(x) return(list(c(“”))))
dev.off()
pdf(“/results/GSEA_Venns.pdf”)
SCLC_res_terms1<-plotVenn(list(YAP1=rownames(subset(comp1, Direction==”Positive” & x>0)),
ASCL1=rownames(subset(comp2, Direction==”Positive” & x>0)),
ND1=rownames(subset(comp3, Direction==”Positive” & x>0))),”Resistors”, retVals=T, scale=F)$n123
SCLC_sen_terms1<-plotVenn(list(YAP1=rownames(subset(comp1, Direction==”Positive” & x<0)),
ASCL1=rownames(subset(comp2, Direction==”Positive” & x<0)),
ND1=rownames(subset(comp3, Direction==”Positive” & x<0))),”Sensitizers”, retVals=T, scale=F)$n123
SCLC_res_terms2<-plotVenn(list(YAP1=rownames(subset(comp7, Direction==”Positive” & x>0)),
ASCL1=rownames(subset(comp8, Direction==”Positive” & x>0)),
ND1=rownames(subset(comp9, Direction==”Positive” & x>0))),”Resistors – no H196″, retVals=T, scale=F)$n123
SCLC_sen_terms2<-plotVenn(list(YAP1=rownames(subset(comp7, Direction==”Positive” & x<0)),
ASCL1=rownames(subset(comp8, Direction==”Positive” & x<0)),
ND1=rownames(subset(comp9, Direction==”Positive” & x<0))), “Sensitizers – no H196”, retVals=T, scale=F)$n123
SCLC_sen_terms<-plotVenn(list(SCLC_1=SCLC_sen_terms1, SCLC_2=SCLC_sen_terms2), “SCLC Sensitizers”, retVals=T, scale=F)$n12
SCLC_res_terms<-plotVenn(list(SCLC_1=SCLC_res_terms1, SCLC_2=SCLC_res_terms2), “SCLC Resistors”, retVals=T, scale=F)$n12
dev.off()
SCLC_res_bioM<-tryCatch(gsea_gmt(SCLC_res_terms, SCLC_gsea, T), error=function(x) return(list(c(“”))))
SCLC_sen_bioM<-tryCatch(gsea_gmt(SCLC_sen_terms, SCLC_gsea2, T), error=function(x) return(list(c(“”))))


GSEA_Venns
最后从已有的基因集,筛选出生物标记基因集的重叠部分,如下图。
#根据已有的基因集,查看生物标记基因集的重叠部分
pdf(“/results/Pathway_Biomarker_Venns.pdf”)
try(plotVenn(list(SCLC=unique(unlist(SCLC_res_bioM)),
YAP1=unique(unlist(YAP1_res_bioM2)),
ASCL1=unique(unlist(ASCL1_res_bioM)),
NEUROD1=unique(unlist(ND1_res_bioM))), title=”Leading Edge Resistors”))
try(plotVenn(list(SCLC=unique(unlist(SCLC_sen_bioM)),
YAP1=unique(unlist(YAP1_sen_bioM2)),
#ASCL1=unique(unlist(ASCL1_sen_bioM)),
NEUROD1=unique(unlist(ND1_sen_bioM))), title=”Leading Edge Sensitizers”))
try(finalbioM<-list(SCLC.res=SCLC_res_bioM, SCLC.sen=SCLC_sen_bioM,
YAP1.res=YAP1_res_bioM2, YAP1.sen=YAP1_sen_bioM2,
ASCL1.res=ASCL1_res_bioM, NEUROD1.res=ND1_res_bioM,
NEUROD1.sen=ND1_sen_bioM))
dev.off()
#输出 GSEA 分析的基因集
write.gmt(unlist(finalbioM, recursive = F), “/results/gmts/Final_BioMarker_List.gmt”)
save.image(“/results/TAK243_Biomarker.RData”)


Pathway_Biomarker_Venns
可以非常清楚的看到处于前沿的重叠基因。现在小果已经带着大家将可能的基因筛选出来了,接下来要做的就是预测SCLC细胞系的TAK反应了。
Step 2 预测 SCLC 细胞系的 TAK 反应(
在这一步,我们的目的是对SCLC中可能对TAK进行反应的通路进行预测。首先将基因集导入,将基因组组合为一个对象,然后删除少于两个基因的基因集,以及来自回归分析的重叠的基因集。接着使用组合的基因集对所有 SCLC 细胞系表达数据运行 ssgsea,将 TAK243 响应数据(EC50 和 AUC)添加到 gsPDX 的前沿。最后绘制热图。
#读取存储在 /results/gmts/ 中的基因集
PathwayBioM<-fgsea::gmtPathways(“/data/Final_BioMarker_List.gmt”)
Resistors1<-fgsea::gmtPathways(“/data/Resistor_Genes1.gmt”)
Resistors2<-fgsea::gmtPathways(“/data/Resistor_Genes2.gmt”)
Sensitizers1<-fgsea::gmtPathways(“/data/Sensitizer_Genes1.gmt”)
Sensitizers2<-fgsea::gmtPathways(“/data/Sensitizer_Genes2.gmt”)
SCLC_rnk<-SCLC_rnk[order(SCLC_rnk, decreasing=T)]
SCLC_rnk2<-SCLC_rnk2[order(SCLC_rnk2, decreasing=T)]
YAP1_rnk<-YAP1_rnk[order(YAP1_rnk, decreasing=T)]
YAP1_rnk2<-YAP1_rnk2[order(YAP1_rnk2, decreasing=T)]
ASCL1_rnk<-ASCL1_rnk[order(ASCL1_rnk, decreasing=T)]
ND1_rnk<-ND1_rnk[order(ND1_rnk, decreasing=T)]
#将基因组组合为一个对象
combGS<-c(PathwayBioM, Resistors1, Resistors2, Sensitizers1, Sensitizers2)
names(combGS)<-c(names(PathwayBioM), paste(names(Resistors1),”res1″, sep=”.”), paste(names(Resistors2),”res2″, sep=”.”),
paste(names(Sensitizers1), “sen1″,sep=”.”), paste(names(Sensitizers2),”sen2″, sep=”.”))
#删除少于两个基因的基因集,以及来自回归分析的重叠的基因集
to_remove<-unique(c(which(lengths(combGS)<2), grep(“SCLC_”, names(combGS)),
grep(“YAP1_”, names(combGS)), grep(“ASCL1_”, names(combGS))))
combGS<-combGS[-to_remove]
#使用组合的基因集对所有 SCLC 细胞系表达数据运行 ssgsea(单样本基因集富集分析)
gsPDX<-GSVA::gsva(as.matrix(fixRows(CCLE, “counts”)),combGS , method=”ssgsea”)
#检查药物反应和 gsPDX 细胞系的顺序是否相同
all.equal(sapply(strsplit(colnames(gsPDX),”_”,T),'[[‘,1), rownames(x))
#将 TAK243 响应数据(EC50 和 AUC)添加到 gsPDX 的前沿
gsPDX<-rbind(x$EC50, x$AUC, gsPDX)
#z-score it
tmp<-t(scale(t(gsPDX)))
tmp[c(1:2),]<-scales::rescale(tmp[c(1:2),], to=c(min(tmp[3:nrow(tmp),]), max(tmp[3:nrow(tmp),])))
#读取热图的注释数据
annodf<-read.csv(“/data/Annodf1.csv”, row.names=1)
#清晰化基因组来源
annodf$Source<-ifelse(annodf$Source==”Pathway”,”Pathway_Analysis”,”Regression_Analysis”)
#注释数据框
colAnno<-data.frame(row.names=colnames(gsPDX),
Subtype=typing$type[which(typing$Cell.line %in% colnames(gsPDX))])
#设置热图上注释的颜色
annoCols<-list(Subtype=colPal(“Dark2”)[1:6],
Annotation=colPal(“Paired”)[1:length(unique(annodf$Annotation))],
Status=colPal(“Dark2”)[5:6],
Source=colPal(“Paired”)[1:2])
names(annoCols$Annotation)<-unique(annodf$Annotation)
names(annoCols$Subtype)<-c(“ASCL1″,”YAP1″,”NEUROD1″,”SCLC”,”ATOH1″,”POU2F3″)
names(annoCols$Status)<-c(“Sensitizer”,”Resistor”)
names(annoCols$Source)<-c(“Pathway_Analysis”,”Regression_Analysis”)
#绘制热图
pdf(“/results/ssGSEA_Biomarker_Heatmap.pdf”)
pheatmap::pheatmap(tmp, show_rownames = F, annotation_row = annodf, cluster_rows=F, treeheight_row=0, annotation_col=colAnno, annotation_colors = annoCols, main=”ssGSEA Enrichment of Biomarker Gene Sets”,na_col=”black”, gaps_row=2)
dev.off()
ssGSEA_Biomarker_Heatmap.pdf

上图是从回归或通路分析中鉴定的每组生物标记基因的ssGSEA浓缩评分的热图,用于鉴定TAK-243在SCLC细胞系亚群中的反应,并随后应用于每个SCLC细胞系。图中的绿色表示敏感性基因通路,黄色表示抵抗性基因通路。
以上就是小果今天给大家介绍的全部内容~不知道大家有没有成功学会GSEA和SSGSEA呢?如果有任何疑问欢迎关注公众号向小果提问,我们有专门的生信分析服务哦。同时,如果大家没有做出来也不要灰心,欢迎租赁我们的服务器,我们会提供全套的代码以及复现思路~