5分钟就够了!小果带你用sf包绘制发表级地图
各位小伙伴大家好啊!小果又带着好东西和大家见面啦,是不是很开心呢。几天看到同事利用arcgis画了一版世界地图,森林沙漠、山川河流、高低起伏,特别好看,让小果也是心生向往啊!随后立刻进行了反思,R语言能不能进行地图的绘制呢,不必特别精美,只需要在进行群体遗传分析时,对某个国家、某个地区或全世界的区域进行展示,随后以便帮助我们进行基因渐渗等分析。
皇天不负苦心人,小果果真找到了一款能进行地图绘制的R包—sf。这款R包简单易上手,在与ggplot2包的合作下,能满足对世界、国家等多种范围的绘图需要。在使用过程中仅需注意要将输入数据的格式改变成‘sf’格式即可。
但虽然该包在空间绘图上功能强大,可是却不自带画世界地图的资料,因此我们需要导入另一个资料包—rnaturalearth,rnaturalearth提供了用于获取和操作全球地理空间数据的功能,不仅免费,还包含各个国家和地区的地理、人口和自然环境等信息,能为我们地图的绘制提供帮助。Rnaturalearthdata同样是带有世界地理资源的R包,相较于上一个R包而言,该包的数据将更为精细,所以在之后绘制的过程中,希望各位小伙伴根据自己的需要择优而选哦。
说完了图形的绘制,咱们再聊一下关于图例的绘制,与以往的legend不同,我们这里选择使用ggspatial包,从名字可以看出,它与ggplot2互通,因此可以在ggplo2的底图的基础之上增加想获得的样式,让你的图片更为绚烂多彩。
以上就是我们应用R语言绘制地图的具体R包,并且这种大尺度的图形也多用于空间较大的数据,而对这种大空间尺度的数据,小果更推荐使用服务器进行数据处理,如果大家没有自己的服务器的话,可以咨询小果进行服务器的租赁哦~
好啦,咱们话不多说,开始今天的代码代练吧!
#软件安装与调用
install.packages(c( “ggplot2”, “ggspatial”, “sf”, “rnaturalearth”, “rnaturalearthdata”))
library(ggplot2)
library(sf)
library(rnaturalearth)
library(rnaturalearthdata)
library(ggspatial)
#获得世界地图的数据
world <- ne_countries(scale = ‘medium’, #地图返回的比例尺,共有110, 50, 10 or ‘small’, ‘medium’, ‘large’
#上述返回值也影响着地图所获得的信息的多与少
returnclass = “sf” #返回结果的格式。一般选择’sf’
)
![]()
可以看到,当我们选择‘medium’类型的数据时,共有241列64行的数据,较之‘large’要少很多,这需要根据各位小伙伴的画图内容进行自主选择了。
class(world)
![]()
随后我们查看了world的类型,属于‘sf’和‘data.frame’类型,其中的‘sf’正是该包画图所需要的类型。
#简单绘制
P1 <- ggplot(world) +
geom_sf()
P1 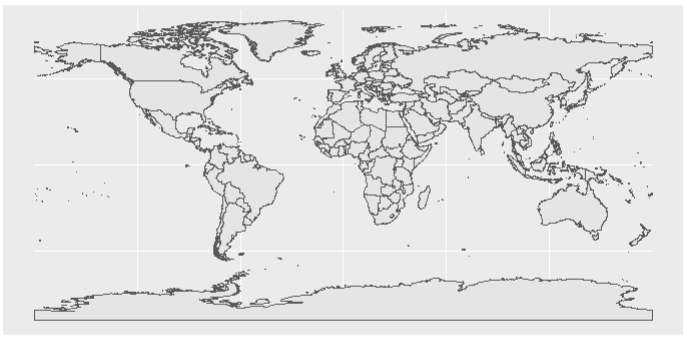
如此就生成了一张世界地图,如果各位小伙伴相对单个国家进行地图的绘制,我们可以采用world$name命令调取各个国家的名称。
world$name
随机挑选一个国家(Thailand)进行地图绘制
Thailand <- ne_countries(scale = ‘medium’,
returnclass = “sf”,
country = ‘Thailand’ #此时对应world$name中的返回名
)
#创建图形
P2 <- ggplot(Thailand) +
geom_sf()
P2
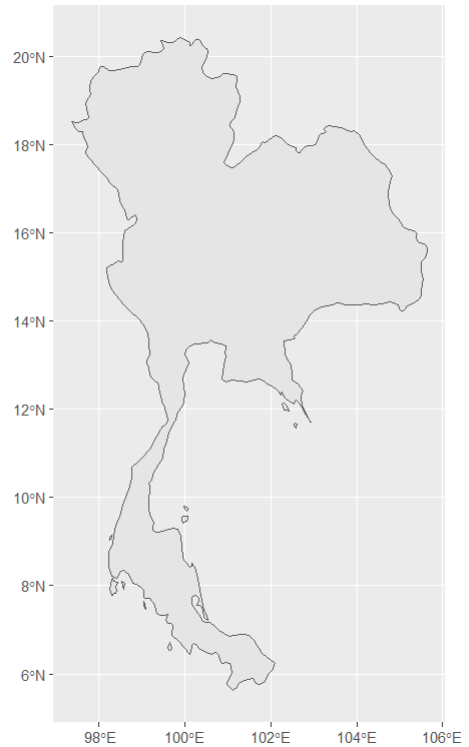
#改变颜色
P3 <- ggplot(data = world) +
geom_sf(color = “black”, fill = “darkred”)
P3
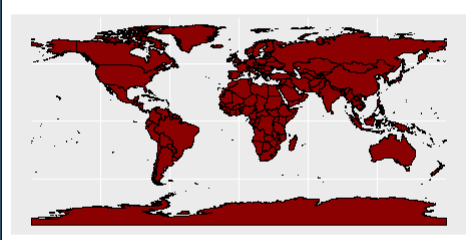
#改变渐变夜色
P4 <- ggplot(world) +
geom_sf(aes(fill = pop_est)) + #pop_est是人口
#这个调色板属于viridis包中,还有magma,inferno,plasma,viridis,cividis,rocket,mako
scale_fill_viridis_c(option = “turbo”,
#根据数值的平方根进行透明度调整,还有根据’log‘对数进行调整,或者默认不调整‘identity’
trans = “sqrt”)
P4
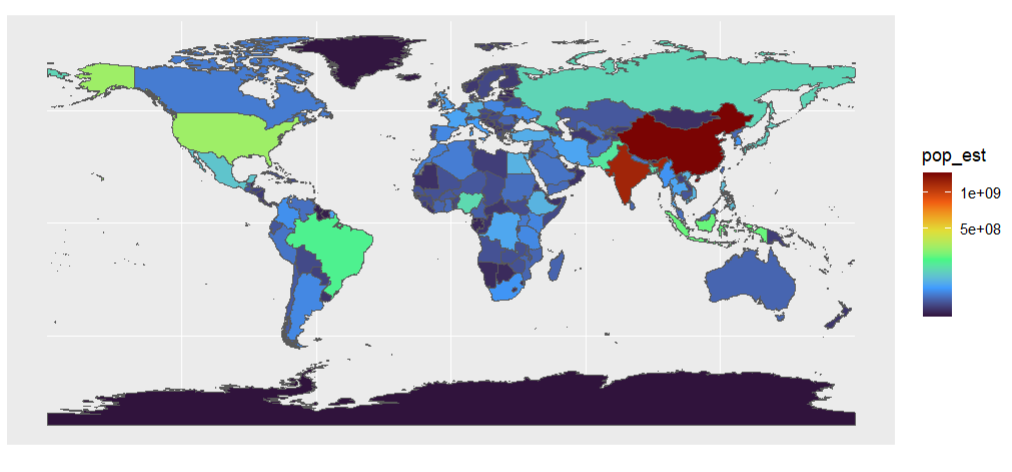
#对心仪位置进行绘画
P5 <- ggplot(data = world) +
geom_sf() +
coord_sf(xlim = c(60, 140), ylim = c(0, 60), #经纬度
expand = FALSE) #是否不用系统进行微调
P5
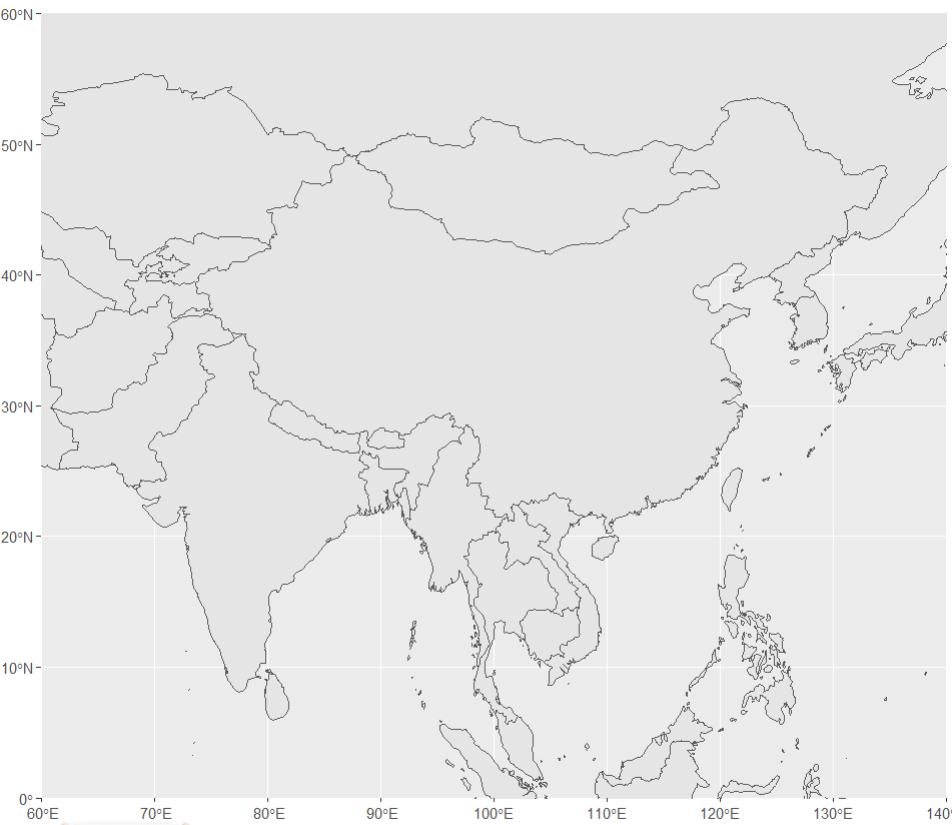
#添加标签
P6 <- ggplot(world) +
geom_sf()+
coord_sf(xlim = c(60, 140), ylim = c(0, 60),
expand = FALSE) +
#对标签进行绘制
annotation_scale(width_hint = 0.2,location = ‘bl’) +
#对指向进行绘制
annotation_north_arrow(location = “bl”, #位置,左上、右上、右下、左下
which_north = “true”, #是否指向北方
pad_x = unit(0.25, “in”), pad_y = unit(0.5, “in”), #指北针与边框的距离
style = north_arrow_fancy_orienteering)
P6
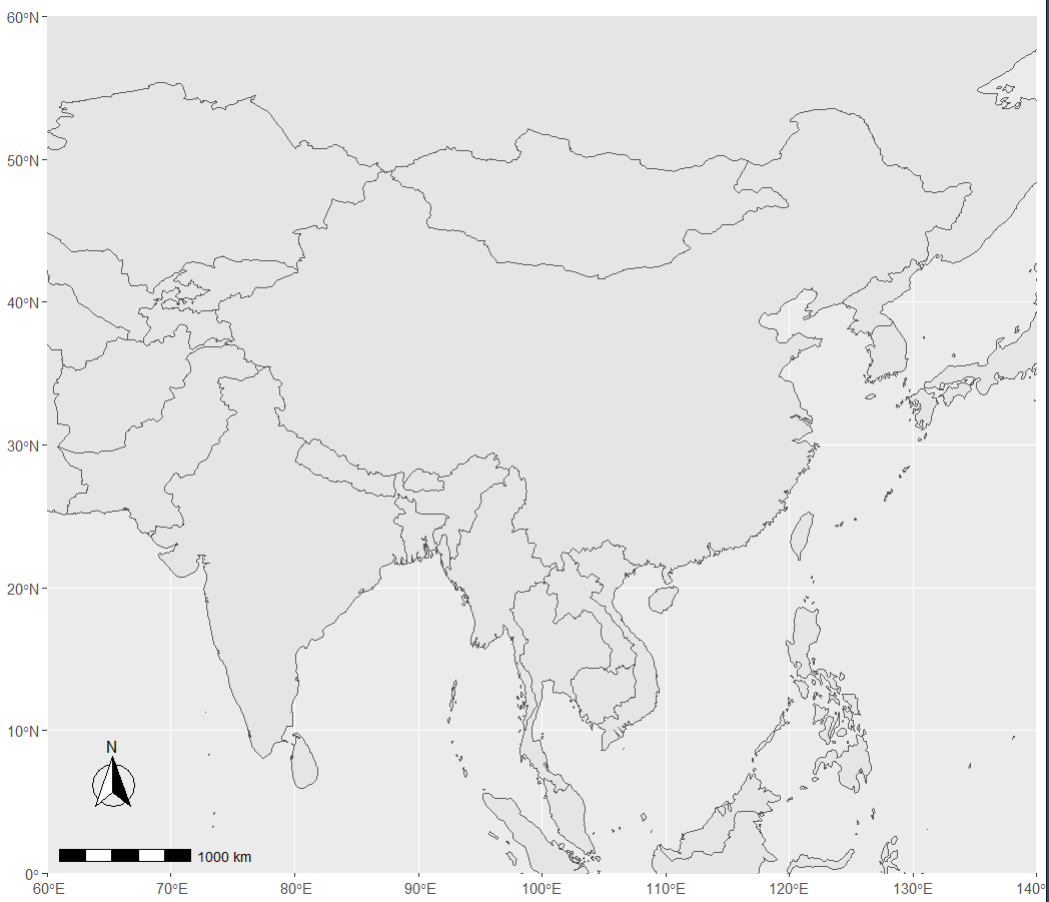
好啦,以上就是咱们今天的全部代练内容哦,是不是特别简单呢。其实针对这种简单的、模式的可视化方式,我更建议大家是在应用中去创新,即多元化应用场景。
小果个人喜欢在三个场景中使用到地图,一是物种分布,如在对基因组进行比较时,我们所选取的物种的采样地,或是在资源调查中展示我们的样品所在地。另一种就是在群体遗传学中的应用了,比如各位小伙伴做了基因渐渗或是群体结构分析,推导出A是由B分化而来,这时候大家可以在地图上展示基因走向或是物种迁移、入侵方向。三是可以用柱状图或饼图的形式展示多地区环境微生物或群落组成的不同,以直观地方式展示出地理位置对物种组成的影响。
比如咱们现在大致绘制了东南亚的行政区域,那我们完全可以拟定某一菌株是从南向北传播的,因此可以大概用箭头进行标识,以此展示传播路线;或可以在北纬30°每隔10个经度进行取样,以饼状图或柱状图标出微生物的变化情况,以此关联地理位置,这就是形式上的创新哦,请各位小伙伴们开动脑筋,加油吧!
话说回来,如果各位小伙伴并不想在数据可视化上花费太大的功夫,可以来我们的云生信平台,里面有多种成熟的可视化代码,只要大家的数据格式正确无误,就可以直接出图哦,方便快捷,还支持颜色和字体的自定义配置,非常适用大家哦~
http://www.biocloudservice.com/home.html