ggcor包是很难安装的,直接使用install.package()和使用Bioc是无法安装的,在R的镜像中也是不存在的,很多人都是将上传到GitHub中。如果你运气好,那么一次就可以安装,但如果你没有一点运气,那就需要花费很长的时间。小果自己安转就花费了很多的时间,如果你按照小果的方法安装,就会省去你很多不必要的麻烦哦。

ggcor包介绍:ggcorr: correlation matrixes with ggplot2 (briatte.github.io)

下载安装
首先下载ggcor包到本地,
本文相关代码及数据都已传到百度云网盘:
链接:https://pan.baidu.com/s/1K9XGIaIiRfTdj2dJgLaMyw?pwd=ea0l
提取码:ea0l
###解压ggcor_master.zip,切换到文件夹目录,安装
devtools::install_local(“ggcor_master”, force = TRUE)
下面是ggcor安装的具体实列及代码:
#使用ggcor绘制图形
library(vegan)#加载vegan包
library(ggcor)
library(ggplot2)
data(“varechem”)#加载vegan内置数据-理化数据
data(“varespec”)#加载vegan内置数据-物种数据
#数据准备
head(varechem)###查看数据
head(varespec)
write.csv(varechem,file=”varechem.csv”) ##保存数据
write.csv(varespec,file=”varespec.csv”)
mantel <- mantel_test(varespec, varechem,
spec.select = list(Spec01 = 1:7,#依次定义四种物种作为Mantel的分析对象
Spec02 = 8:18,
Spec03 = 19:37,
Spec04 = 38:44)) %>%
mutate(rd = cut(r, breaks = c(-Inf, 0.2, 0.4, Inf),
labels = c(“< 0.2”, “0.2 – 0.4”, “>= 0.4”)),#定义Mantel的R值范围标签,便于出图
pd = cut(p.value, breaks = c(-Inf, 0.01, 0.05, Inf),
labels = c(“< 0.01”, “0.01 – 0.05”, “>= 0.05”)))#定义Mantel检验的p值范围标签,便于出图
pdf(“corrplot.pdf”,10,10)
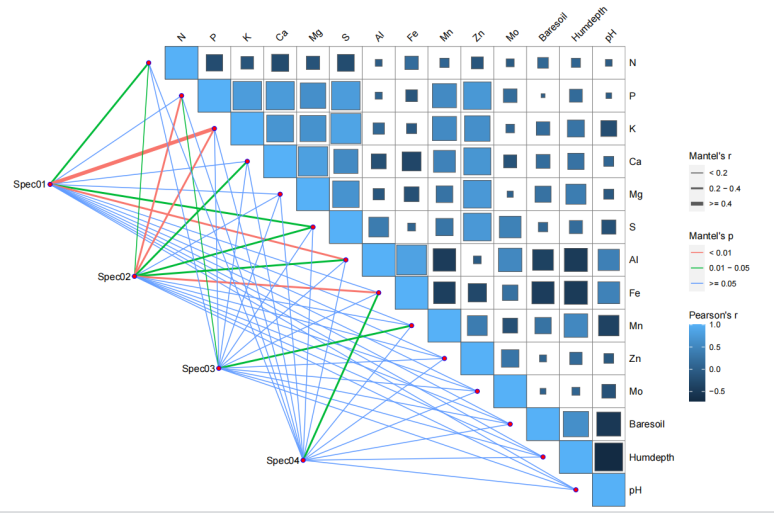
quickcor(varechem, type = “upper”) +#绘制理化数据热图
geom_square() +#定义成方块状
anno_link(aes(colour = pd, size = rd), data = mantel) +#定义连线
scale_size_manual(values = c(0.5, 1, 2))+
guides(size = guide_legend(title = “Mantel’s r”,#定义图例
order = 2),
colour = guide_legend(title = “Mantel’s p”,
order = 3),
fill = guide_colorbar(title = “Pearson’s r”, order = 4))
dev.off()

结果图片:
欢迎关注”生信果”、“云生信学生物信息学”、“尔云间meta分析”公众号,以及知乎、BiliBili等主要发表或收录生物信息学的教程,以及基于R的分析和可视化等内容,一起见证小白和大佬的成长。
