今天小果绘制一下基因位置的环形图,绘图代码如下:
- 安装所需要的包
BiocManager::install(“RCircos”)
install.packages(“magritter”)
install.packages(“tidyverse”)
2导入需要的R包
library(RCircos)
library(magritter)
library(tidyverse)
- 读取数据
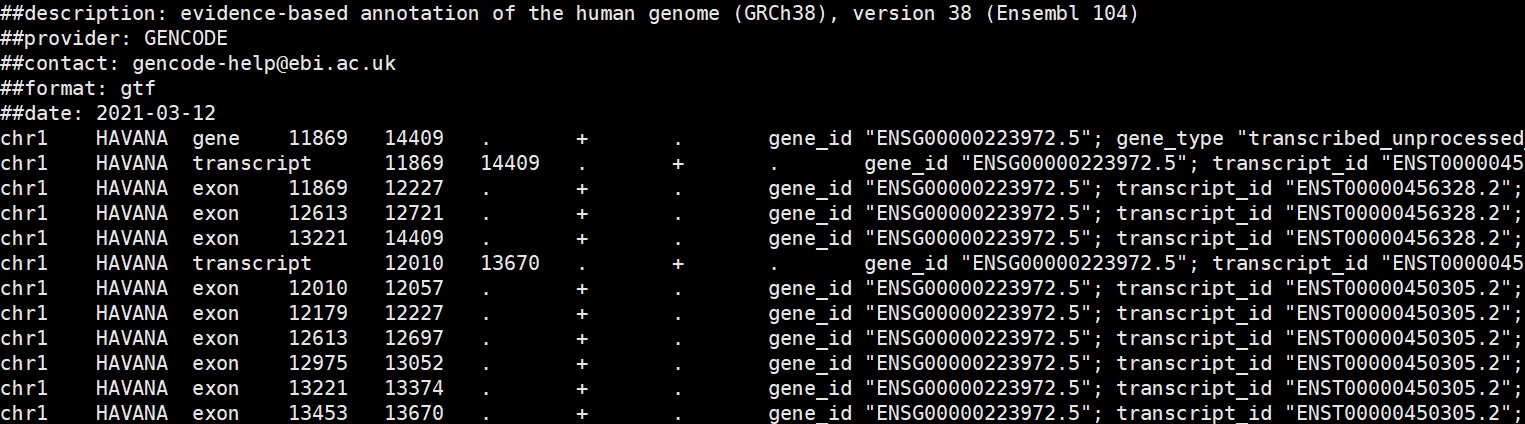
#图中展示目标基因文件
genes <- read.table(“gene.txt”, header = T)$SYMBOL

# 根据基因集提取出目的基因所在位置
gene_pos <- import(“hg38.gtf”) %>% # 载入gtf文件
as.data.frame %>%
# 仅选择基因,去除转录本等等
filter(source == “HAVANA”, type == “gene”) %>%
# 保留基因位置和名称
dplyr::select(seqnames, start, end, gene_name) %>%
# 挑选目的基因
filter(gene_name %in% genes)
# 原文没说内圈散点图代表什么特征,我们这里随机生成一列数值
gene_pos$gene_dot <- rnorm(nrow(gene_pos), 0, 2)
# 保存到文件,便于套用格式
write.csv(gene_pos,”gene_pos.csv”, row.names = F, quote = F)
#加载基因所在的位置和数值
gene_pos <- read.csv(“gene_pos.csv”, header = T)
# 加载染色体Ideogram
(data(“UCSC.HG38.Human.CytoBandIdeogram”))
pdf(file=”circGene.pdf”, height=5, width=5)
# 根据hg38构建染色体位置,只保留chr1-22,X,Y,在圈内部构建三圈轨道
RCircos.Set.Core.Components(UCSC.HG38.Human.CytoBandIdeogram,
chr.exclude = NULL,
tracks.inside = 3,
tracks.outside = 0)
RCircos.Set.Plot.Area()
# 绘制染色体
RCircos.Chromosome.Ideogram.Plot()
# 在第一圈用散点在基因所在的位置标注数值
# 调整配色
params <- RCircos.Get.Plot.Parameters()
params$track.background <- “grey” # 第三圈默认配色为wheat,模仿原文修改为灰色
RCircos.Reset.Plot.Parameters(params)
RCircos.Scatter.Plot(gene_pos,
data.col = 5, # 用第5列的数值作为点的纵坐标
by.fold = 1, # 点的颜色cutoff,大于等于1的基因显示为红色点,小于等于-1的显示为蓝色点,-1到1之间为黑点
track.num = 1,
side = “in”)
# 在第二圈绘制线段标注基因所在的位置
RCircos.Gene.Connector.Plot(genomic.data = gene_pos,
track.num = 2,
side = “in”)
# 在第三圈标注基因名
RCircos.Gene.Name.Plot(gene_pos,
name.col = 4,
track.num = 3,
side = “in”)
dev.off()
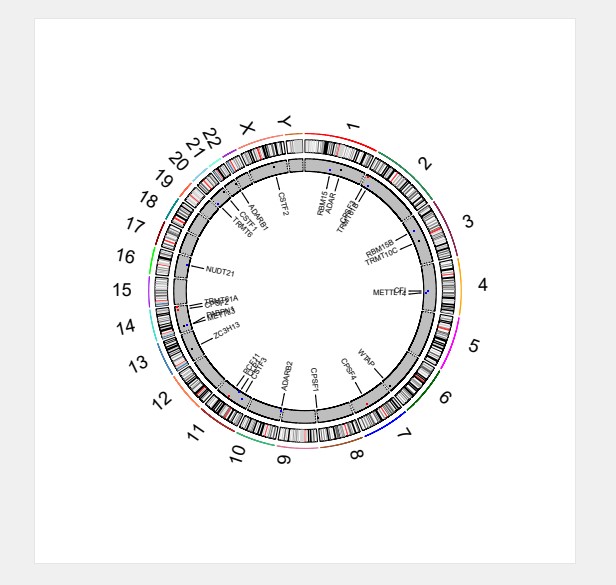
小果最终绘制出了目标基因位置环形图,有需要的可以借鉴学习。