今天在做ssGESA分析的时候发现,TCGA下载的基因ID为Ensembl ID,下载的基因集为Gene name,在做分析之前要做一下基因ID转化,代码如下:
- 安装需要的R包
install.packages(“tidyverse”)
- 导入需要的R包
library(tidyverse)
- 数据下载
在Xena数据库下载表达矩阵和ID对应表格
#表达矩阵下载
wget https://gdc-hub.s3.us-east-1.amazonaws.com/download/TCGA-KIRC.htseq_fpkm.tsv.gz
#基因ID转化列表
wget https://gdc-hub.s3.us-east-1.amazonaws.com/download/gencode.v22.annotation.gene.probeMap
4.代码展示
#表达矩阵
expr<-read.table(“TCGA-KIRC.tsv”,header=T,sep=”\t”,row.names=1)
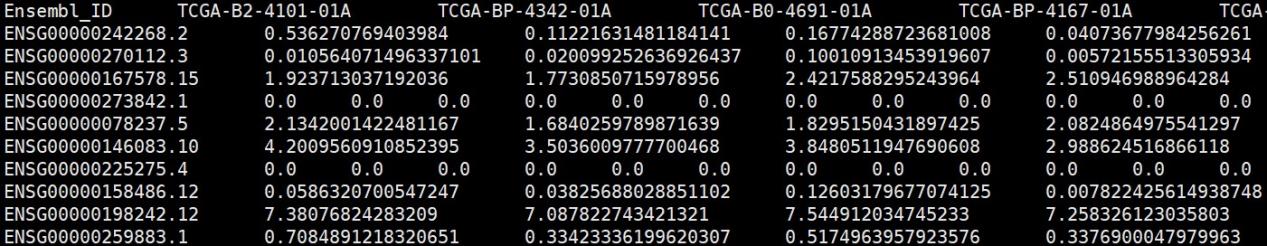
#ID和Gene symbol对应列表
geneann<-read.table(“gencode.v22.annotation.gene.probeMap”,header=T,sep=”\t”,row.names=1)
#二者ID进行匹配,并添加一列gsym
expr$gsym <- geneann[rownames(expr),]$gene
#去除重复的Gene name
expr<-distinct(expr,gsym,.keep_all=T)
#将行名改为Gene name
row.names(expr)<-expr$gsym
#将添加的gsym这一列删除
expr<-slect(expr,-gsym)
#将修改的结果文件保存
write.table(expr,”TCGA-KIRC-expr.txt”,col.names=T,row.names=T,sep=”\t”)
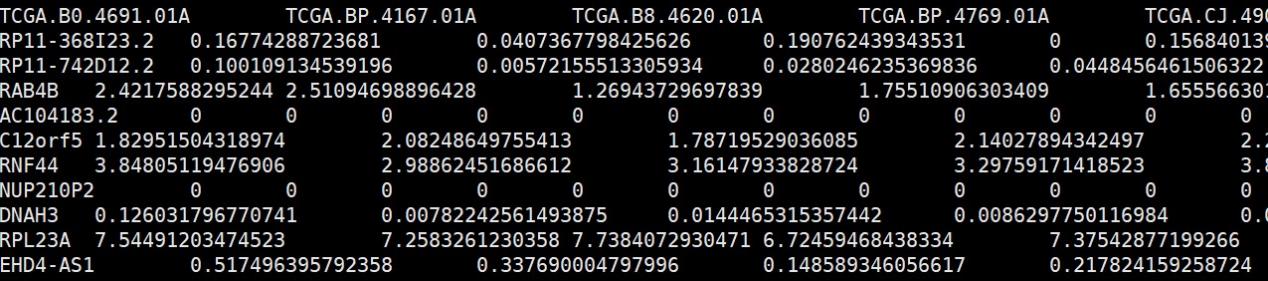
最终将Ensembl ID转化为gene name,有需要的可以借鉴学习,在这里需要注意的是多个Ensembl ID对应一个Gene name,小果今天的分享就到这里。