今天小果想用棒棒糖图来展示单细胞marker基因在不同细胞群里的表达量情况,展示出的效果非常不错,单细胞数据来自以前推文单细胞数据分析结果,有需要的可以系统学习一下单细胞数据分析流程,话不多说开始今天的分享,代码如下:
- 安装需要的R包
install.packages(“BiocManager”)
BiocManager::install(“Seurat”)
install.packages(“ggsci”)
install.packages(“reshape2”)
install.packages(“tidyverse”)
install.packages(“ggplot2”)
- 导入需要的R包
library(Seurat)
library(ggsci)
library(reshape2)
library(tidyverse)
library(ggplot2)
3.开始绘图
#需要展示的marker基因
gene <- c(“CD3E”,”MS4A1″,”GNLY”,”CD14″)
#计算平均表达量
Idents(pbmc) <- “seurat_clusters”
AveExpression <- AverageExpression(pbmc, assays = “RNA”, features = gene,verbose = TRUE) %>% .$RNA %>%as.data.frame
#将行名转换为列
AveExpression$Gene <- rownames(AveExpression)
#长宽数据转换
df <- melt(AveExpression,id.vars= “Gene”)
colnames(df) <- c(“Gene”, “Cluster”, “Expression”)
#对表达量进行设置
df$Expression[which(df$Expression>10)] <- 10 ####设置平均表达量大于10的值为10
#开始绘图
p1 <- ggplot(df,aes(x=Gene, y=Expression))+
geom_hline(yintercept = seq(0, 10, 2.5),linetype = 2, color = “lightgray”,size=1)+
geom_segment(aes(x=Gene,xend=Gene,y=0,yend=Expression),color=”lightgray”,size = 1.5)+
geom_point(size=3,aes(color=Gene))+
scale_color_npg() +
theme_bw()+
theme(panel.grid =element_blank()) +
labs(x=””,y=”Gene expression”)
#分面
p1 <- facet(p1, facet.by = “Cluster”,ncol = length(unique(df$Cluster)),panel.labs.font = list(size = 12),panel.labs.background = list(fill = “#a6cee3”))
#调整主题
p1 <- p1 + scale_y_continuous(position = “right”)+ ####用来将y轴移动位置
theme(axis.text.y = element_text(size=12, colour = “black”))+
theme(axis.text.x = element_blank())+ ## 删去X轴所有刻度标签
theme(axis.title.y = element_text(size=12, colour = “black”))+
theme(axis.title.x = element_blank())+
theme(legend.position = “right”,
panel.border = element_blank(),## 去掉最外层的正方形边框
axis.ticks.x = element_line(color = NA))
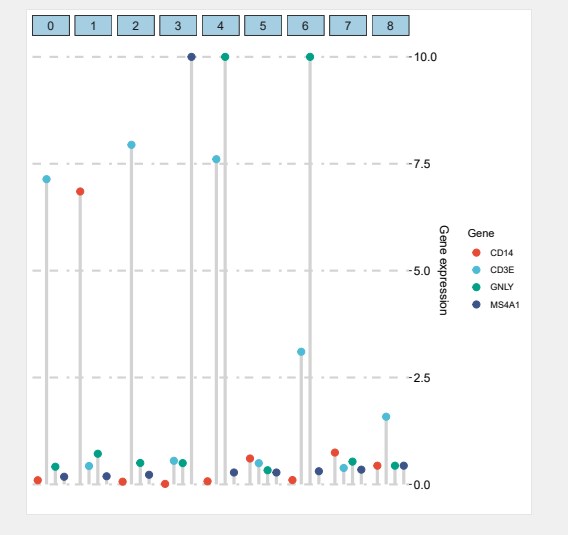
小果今天的分享就到这里,单细胞数据分析需要在性能好的服务器进行数据分析,小果安利本公司服务器,已安装所有单细胞分析软件和R包,有需要的可以联系小果,下期再见奥。