今天小果发现一款非常不错的R包EnhancedVolcano,该包画出来的火山图效果非常不错,火山图的颜值非常高,下面尝试一下,代码如下:
- 安装需要的R包
install.packages(“BiocManager”)
BiocManager::install(“EnhancedVolcano”)
install.packages(“tidyverse”)
- 导入需要的R包
library(BiocManager)
library(tidyverse)
library(EnhancedVolcano)
- 示例数据
#差异分析结果文件
df <- read.table(“limma.txt”, header = T)
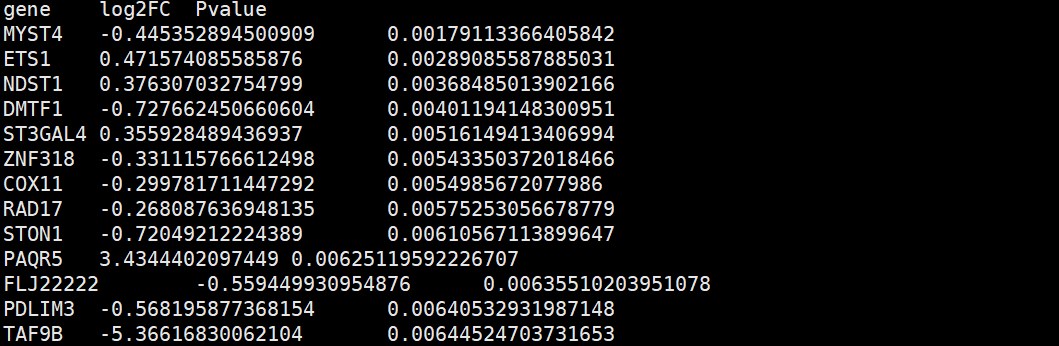
#目标基因文件
markergene<-read.table(“genelist”,header=T)$gene

- 代码展示
group<-ifelse(
df$log2FC<(-1.5)&df$Pvalue<0.05,’#4D4398′,
ifelse(df$log2FC>(1.5)&df$Pvalue<0.05,’#F18D00′,
‘#b5b5b5’))
group[is.na(group)]<-‘#b5b5b5′
names(group)[group==’#F18D00’]<-‘Up’
names(group)[group==’#b5b5b5′]<-‘Nosignif’
names(group)[group==’#4D4398′]<-‘Down’
#开始绘图
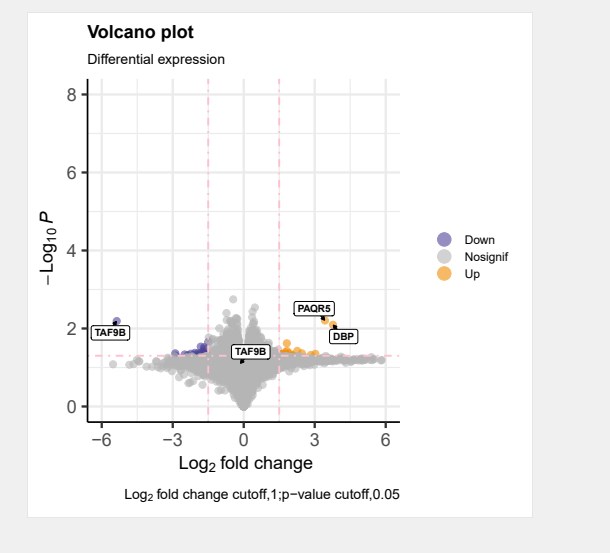
EnhancedVolcano(df,
x=”log2FC”,
y=”Pvalue”,
lab=df$gene,
pCutoff=10e-1/20,#y轴阈值线(水平)
FCcutoff=1.5,#x轴阈值线(垂直)
pointSize=3,#散点大小
labSize=3.5,#标签大小
xlim=c(-6, 6),#X轴范围
ylim=c(0,8),#Y轴范围
selectLab=c(markergene),#使用selectLab参数选定所关注的标签
xlab=bquote(~Log[2]~’fold change’),#将内容传递给xlab
labCol=’black’,#标签颜色
labFace=’bold’,#标签字体
boxedLabels=TRUE,#是否在框中绘制标签
drawConnectors=TRUE,#是否通过连线将标签连接到对应的点上
widthConnectors=0.8,#连线的宽度
endsConnectors=”last”,#连线绘制箭头的方向,可选first、both、last
colConnectors=’black’,#连线的颜色
colCustom=group,#用group覆盖默认配色方案
colAlpha=0.6,#调整透明度
cutoffLineType=’dotdash’,#阈值线类型
cutoffLineCol=’pink’,#阈值线颜色
cutoffLineWidth=0.88,#阈值线粗细
title=”Volcano plot”,#主标题
subtitle=”Differential expression”,#副标题
caption=bquote(~Log[2]~”fold change cutoff,1;p-value cutoff,0.05″),#注释说明
legendPosition=’right’,#图例位置
legendLabSize=12,#图例文字大小
legendIconSize=6)
今天小果的分享就到这里,有需要的可以借鉴学习,下期再见。