单细胞数据marker基因小提琴图绘制
今天小果想通过单细胞数据marker基因表达量数据来绘制分面小提琴图,通过小提琴图来展示marker基因在不同细胞群中的分布情况,并做了显著性分析,话不多说开始今天的分享,代码如下:
- 安装需要的R包
install.packages(“ggplot2”)
install.packages(“ggsci”)
install.packages(“ggpubr”)
install.packages(“rstatix”)
install.packafes(“tidyverse”)
- 载入需要的R包
library(ggplot2)
library(ggsci)
library(ggpubr)
library(rstatix)
library(tidyverse)
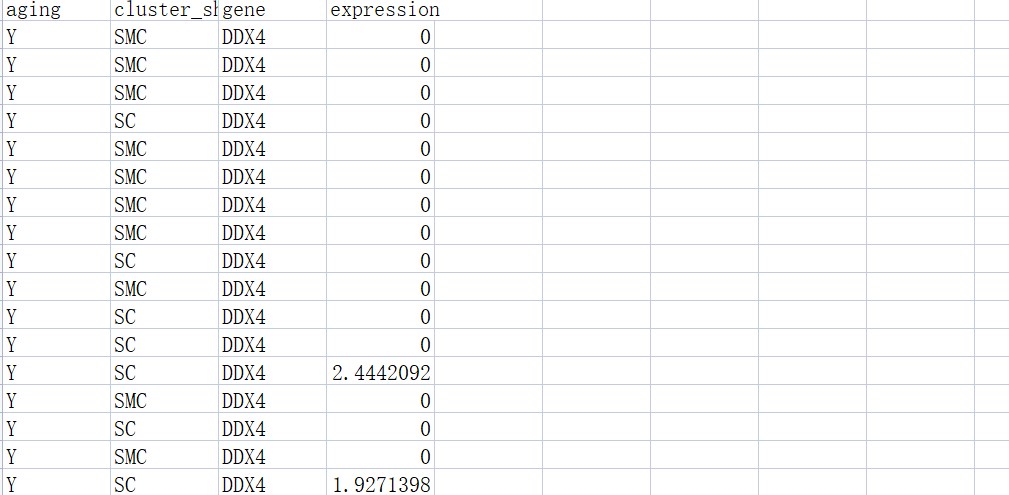
- 示例数据
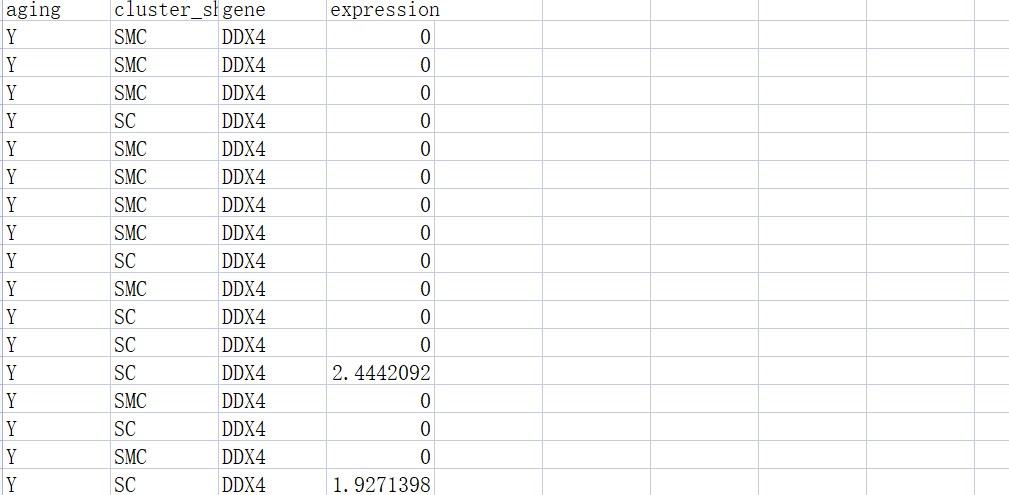
df <- read.csv(“scviolin.csv”, header = T)
# 按自己的要求排序,该顺序对应图中的顺序
df$aging <- factor(df$aging, levels = c(“Y”, “O”))
df$cluster_short <- factor(df$cluster_short, levels = c(“OO”,”GC”, “SC”, “SMC”, “EC”, “NKT”, “M”))
df$gene <- factor(df$gene, levels = c(“DDX4”, “AMH”, “COL1A2”, “ACTG2”, “CDH5”, “KLRB1”, “CD163”))
- 绘图代码
#显著性分析
stat.test <- df %>%
group_by(cluster_short, gene) %>%
t_test(expression ~ aging) %>%
adjust_pvalue(method = “bonferroni”) %>%
add_significance()
stat.test <- stat.test %>% add_xy_position(x = “aging”)
#开始绘图
p1 <- ggviolin(df, x = “aging”, y = “expression”,
fill = “aging”,
facet = c(“gene”,”cluster_short”)) + #分面
scale_fill_npg() +
xlab(“”) +
ylab(“Expression”) +
theme(panel.grid = element_blank(),
strip.text.y = element_text(angle = 0),
strip.background.x = element_rect(fill = “grey95”), # 顶部填充浅灰色
strip.background.y = element_blank())
p1 +
stat_pvalue_manual(stat.test,
bracket.nudge.y = 1,
hide.ns = TRUE, #不显示不显著的
label = “{p.adj}{p.adj.signif}”, # pvalue和星标
label.size = 3, # pvalue字的大小
vjust = -0.5) + # pvalue的上(负)下位置
scale_y_continuous(expand = expansion(mult = c(0, 0.1))) #给pvalue文字留足空间
ggsave(“scViolin.pdf”, width = 9, height = 9)
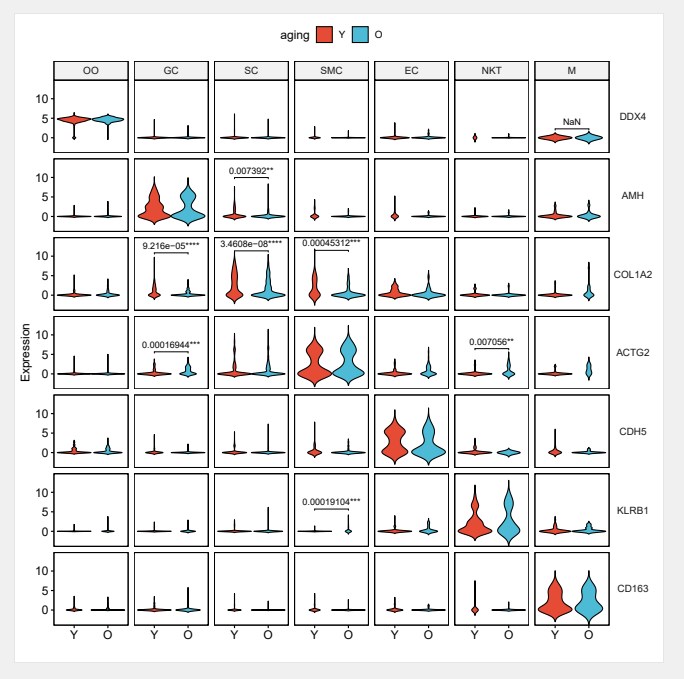
小果最终绘制出了这张漂亮的小提琴图,看起来效果还不错奥,在这里安利一下奥,本公司服务器已经安装好相关单细胞数据分析的R包和软件,有需要的可以联系小果,本期的分享就到这里下期在见奥。
单细胞数据marker基因小提琴图绘制
今天小果想通过单细胞数据marker基因表达量数据来绘制分面小提琴图,通过小提琴图来展示marker基因在不同细胞群中的分布情况,并做了显著性分析,话不多说开始今天的分享,代码如下:
- 安装需要的R包
install.packages(“ggplot2”)
install.packages(“ggsci”)
install.packages(“ggpubr”)
install.packages(“rstatix”)
install.packafes(“tidyverse”)
- 载入需要的R包
library(ggplot2)
library(ggsci)
library(ggpubr)
library(rstatix)
library(tidyverse)
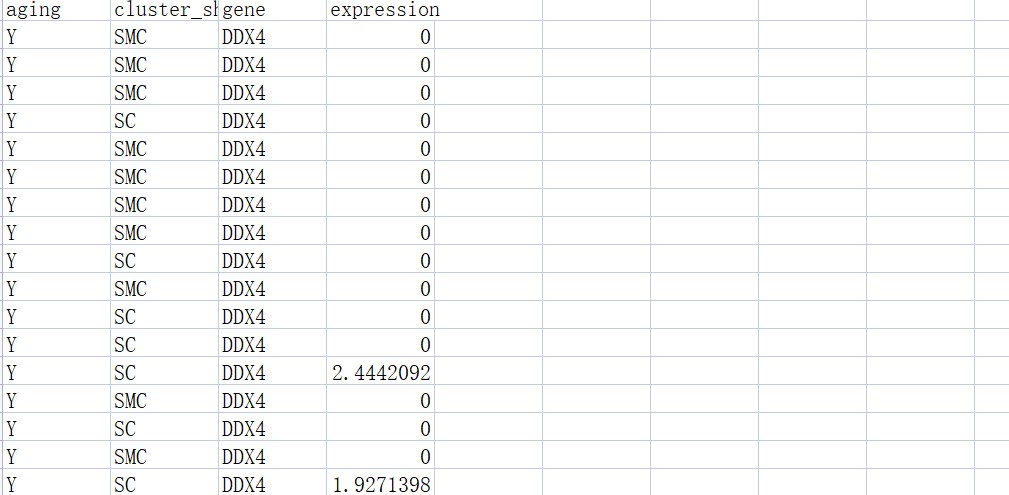
- 示例数据
df <- read.csv(“scviolin.csv”, header = T)
# 按自己的要求排序,该顺序对应图中的顺序
df$aging <- factor(df$aging, levels = c(“Y”, “O”))
df$cluster_short <- factor(df$cluster_short, levels = c(“OO”,”GC”, “SC”, “SMC”, “EC”, “NKT”, “M”))
df$gene <- factor(df$gene, levels = c(“DDX4”, “AMH”, “COL1A2”, “ACTG2”, “CDH5”, “KLRB1”, “CD163”))
- 绘图代码
#显著性分析
stat.test <- df %>%
group_by(cluster_short, gene) %>%
t_test(expression ~ aging) %>%
adjust_pvalue(method = “bonferroni”) %>%
add_significance()
stat.test <- stat.test %>% add_xy_position(x = “aging”)
#开始绘图
p1 <- ggviolin(df, x = “aging”, y = “expression”,
fill = “aging”,
facet = c(“gene”,”cluster_short”)) + #分面
scale_fill_npg() +
xlab(“”) +
ylab(“Expression”) +
theme(panel.grid = element_blank(),
strip.text.y = element_text(angle = 0),
strip.background.x = element_rect(fill = “grey95”), # 顶部填充浅灰色
strip.background.y = element_blank())
p1 +
stat_pvalue_manual(stat.test,
bracket.nudge.y = 1,
hide.ns = TRUE, #不显示不显著的
label = “{p.adj}{p.adj.signif}”, # pvalue和星标
label.size = 3, # pvalue字的大小
vjust = -0.5) + # pvalue的上(负)下位置
scale_y_continuous(expand = expansion(mult = c(0, 0.1))) #给pvalue文字留足空间
ggsave(“scViolin.pdf”, width = 9, height = 9)
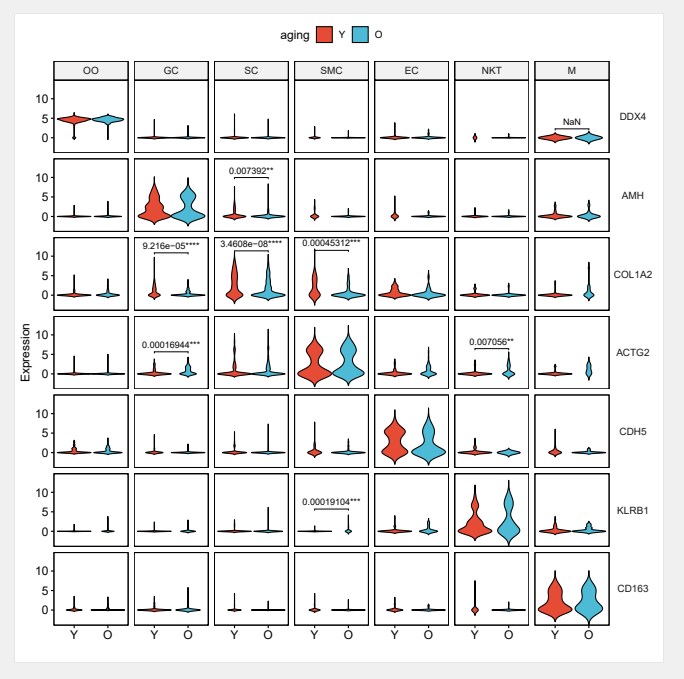
小果最终绘制出了这张漂亮的小提琴图,看起来效果还不错奥,在这里安利一下奥,本公司服务器已经安装好相关单细胞数据分析的R包和软件,有需要的可以联系小果,本期的分享就到这里下期在见奥。
单细胞数据marker基因小提琴图绘制
今天小果想通过单细胞数据marker基因表达量数据来绘制分面小提琴图,通过小提琴图来展示marker基因在不同细胞群中的分布情况,并做了显著性分析,话不多说开始今天的分享,代码如下:
- 安装需要的R包
install.packages(“ggplot2”)
install.packages(“ggsci”)
install.packages(“ggpubr”)
install.packages(“rstatix”)
install.packafes(“tidyverse”)
- 载入需要的R包
library(ggplot2)
library(ggsci)
library(ggpubr)
library(rstatix)
library(tidyverse)
- 示例数据
df <- read.csv(“scviolin.csv”, header = T)
# 按自己的要求排序,该顺序对应图中的顺序
df$aging <- factor(df$aging, levels = c(“Y”, “O”))
df$cluster_short <- factor(df$cluster_short, levels = c(“OO”,”GC”, “SC”, “SMC”, “EC”, “NKT”, “M”))
df$gene <- factor(df$gene, levels = c(“DDX4”, “AMH”, “COL1A2”, “ACTG2”, “CDH5”, “KLRB1”, “CD163”))
- 绘图代码
#显著性分析
stat.test <- df %>%
group_by(cluster_short, gene) %>%
t_test(expression ~ aging) %>%
adjust_pvalue(method = “bonferroni”) %>%
add_significance()
stat.test <- stat.test %>% add_xy_position(x = “aging”)
#开始绘图
p1 <- ggviolin(df, x = “aging”, y = “expression”,
fill = “aging”,
facet = c(“gene”,”cluster_short”)) + #分面
scale_fill_npg() +
xlab(“”) +
ylab(“Expression”) +
theme(panel.grid = element_blank(),
strip.text.y = element_text(angle = 0),
strip.background.x = element_rect(fill = “grey95”), # 顶部填充浅灰色
strip.background.y = element_blank())
p1 +
stat_pvalue_manual(stat.test,
bracket.nudge.y = 1,
hide.ns = TRUE, #不显示不显著的
label = “{p.adj}{p.adj.signif}”, # pvalue和星标
label.size = 3, # pvalue字的大小
vjust = -0.5) + # pvalue的上(负)下位置
scale_y_continuous(expand = expansion(mult = c(0, 0.1))) #给pvalue文字留足空间
ggsave(“scViolin.pdf”, width = 9, height = 9)
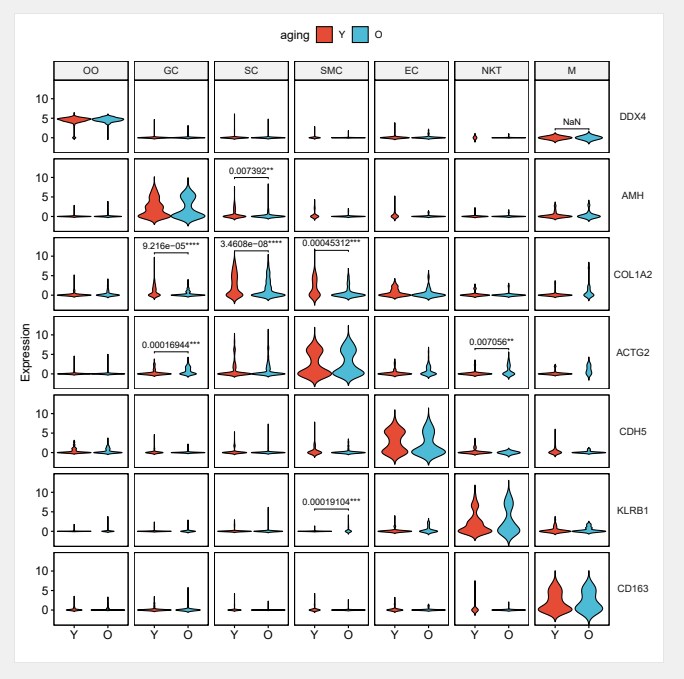
小果最终绘制出了这张漂亮的小提琴图,看起来效果还不错奥,在这里安利一下奥,本公司服务器已经安装好相关单细胞数据分析的R包和软件,有需要的可以联系小果,本期的分享就到这里下期在见奥。