单细胞不同类型测序文件读取
在进行单细胞数据分析时,第一步就是单细胞测序文件的读入,单细胞文件形式各种各样,
今天小果讲解一下几种不同类型文件的读入方法,方法如下:
1.10X的单细胞文件
#读取数据10X数据所在的文件夹
pbmc.data <- Read10X(data.dir = “filtered_gene_bc_matrices/hg19/”)
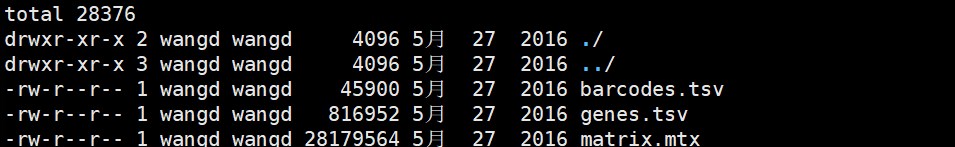
#创建Seurat文件
pbmc <- CreateSeuratObject(counts = pbmc.data, project = “pbmc3k”, min.cells = 3, min.features = 200)
- 输入数据是基于UMI的表达矩阵(其中数据集中的行表示基因,列表示细胞)
molecules <- read.delim(“molecules.txt”,header = TRUE,
row.names = 1)
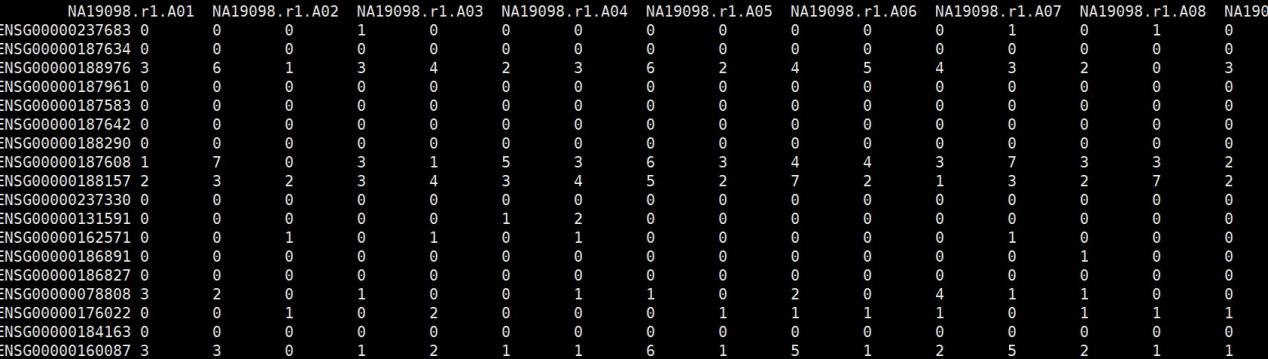
#创建Seurat文件
obj <- CreateSeuratObject(counts = molecules,
min.cells = 3, #筛选至少在3个细胞中表达的基因
min.features = 200) #筛选至少有200个基因表达的细胞
#增加Seurat的元信息(metadata)
ann <- read.delim(“annotation.txt”, header = TRUE)
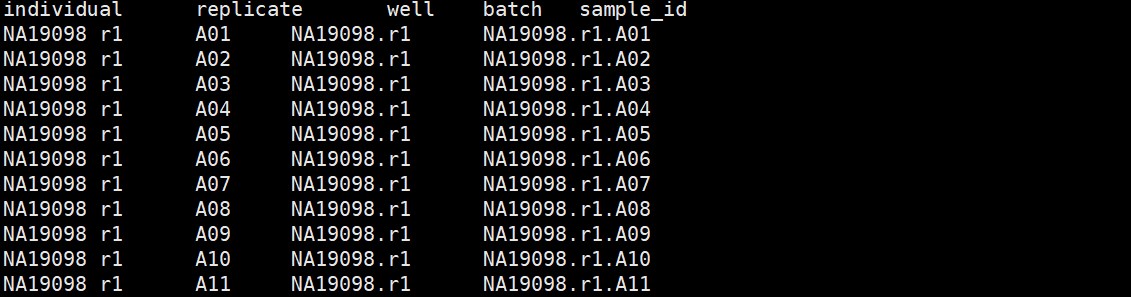
rownames(ann) <- ann$sample_id
obj <- AddMetaData(obj, ann)
- 输入数据是h5文件格式
devtools::install_github(repo = “hhoeflin/hdf5r”)
library(hdf5r)
obj<-Read10X_h5(“GSE154989_mmLungPlate_fQC_dSp_rawCount.h5”)
obj <- CreateSeuratObject(counts = obi, project=”obj”,
min.cells = 3, #筛选至少在3个细胞中表达的基因
min.features = 200) #筛选至少有200个基因表达的细胞
- 利用SeuratData包导入内置数据
devtools::install_github(‘satijalab/seurat-data’,force = TRUE)
library(SeuratData)
InstallData(“ifnb”)
LoadData(“ifnb”)
- Seurat对象
如果文件是已经构建好的seurat对象,后期可以直接进行质控和单细胞分析流程;如果发现发现上传seurat版本较低,需要更新转化。
obj<-UpdataSeuratObject(object=obj)
单细胞测序文件类型大概就这几种,在进行数据读入时可以按照数据类型选择不同的方法进行读取,今天小果的分享就到这里,有需要的可以点赞收藏加个关注奥,下期再见奥!!!