生信学习之scater包的安装与简单应用
什么是scater?
小果今天学习的R包是scater,那么首先来简单了解一下scater包的基本概念吧!
scater是一个用来对单细胞RNA-seq数据进行预处理、质量控制、规范化和可视化的R工具包。首先,该R工具包可以对输入的原始测序数据处理为高质量的表达数据集,也提供了不同种类的绘图模块以实现单细胞测序数据不同维度方面的可视化工作。那接下来大家就跟着小果一起来看一下如何安装scater包吧!
scater的安装
因为scater的安装是基于github来安装的,所以没有安装devtools软件包的同学要先安装好devtools工具包~
if(!require(devtools))
install.packages(“devtools”)
if(!require(scater))
devtools::install_github(“davismcc/scater”, build_vignettes = TRUE)
- scater软件包安装时可能会出现的错误
下载的时候你也许回收到报错信息,这是怎么回事呢?在下载的过程中小果也遇到了同样的问题!让我们来一起看一下吧!
哈哈哈!原来是是由于scater工具包的安装还依赖了‘BiocStyle’软件包,如果没有提前安装,则会出现以下错误信息:
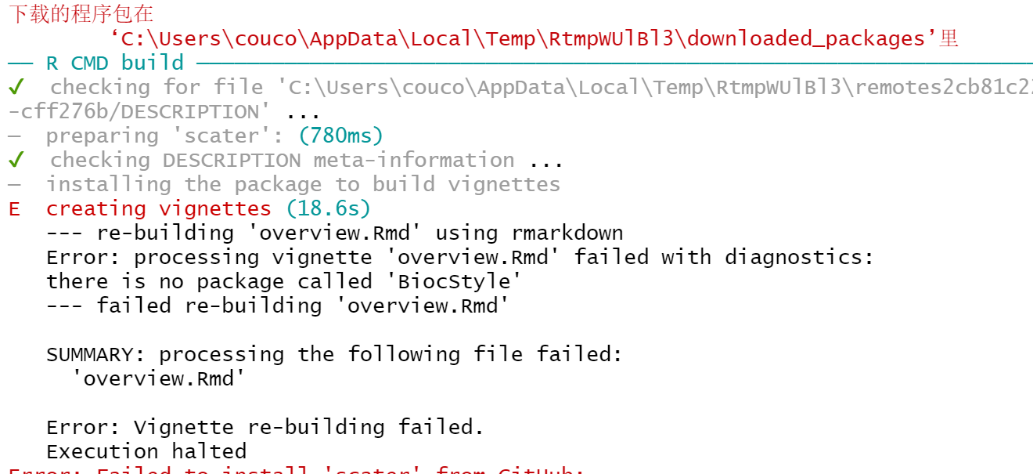
为了解决这一问题,我们先安装 BiocStyle软件包,我们借助Bioconductor来安装:
BiocManager::install(c(“BiocStyle”))
好了,下面我们就可以继续安装scater软件包:
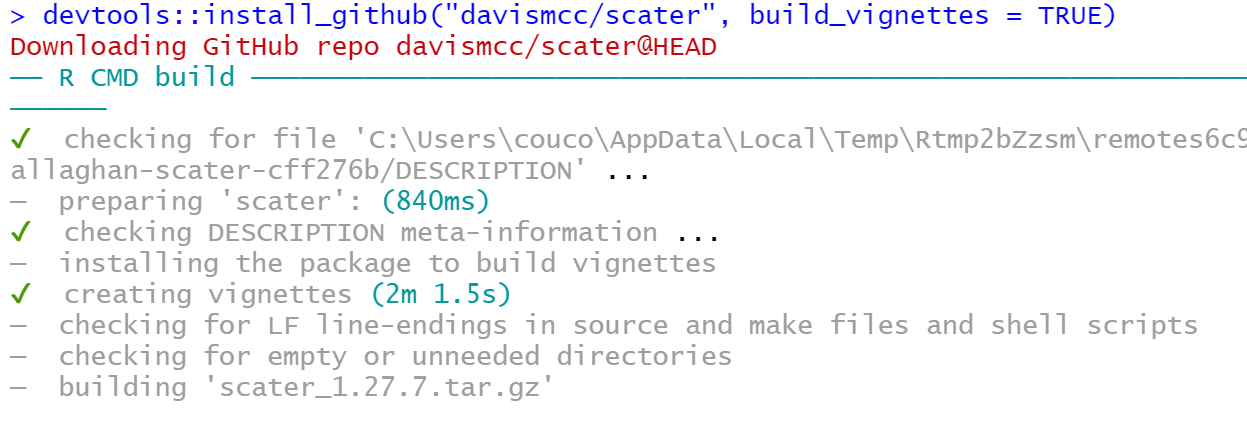
scater包的使用
- 准备数据
哼哼,小果提醒:我们的数据基于BiocStyle和scRNAseq这两个包来准备哦!
library(BiocStyle)
library(scRNAseq)
set.seed(10918)
data <- ZeiselBrainData()
data
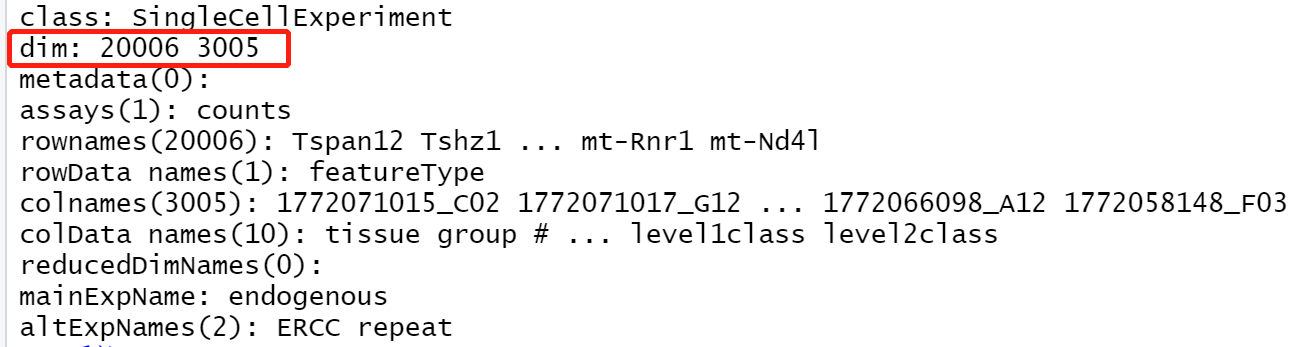
从输出信息中可以看出数据的维度是20006行和3005列。
- 数据质控
好啦!数据准备好后我们就要进行我们经常会进行的数据质控工作啦~
现在小果要基于准备好的数据进行质控,去掉其中的线粒体基因,也就是以“mt”开头的基因哦。
library(scater)
data <- addPerCellQC(data,
subsets=list(Mito=grep(“mt-“, rownames(data))))
- 细胞信息可视化
将我们上面质控后的数据提取tissue和class:
tp = as.data.frame(data@colData)
table(tp$tissue)

table(tp$level1class)

table(tp$sex)
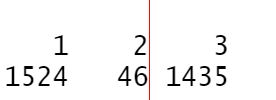
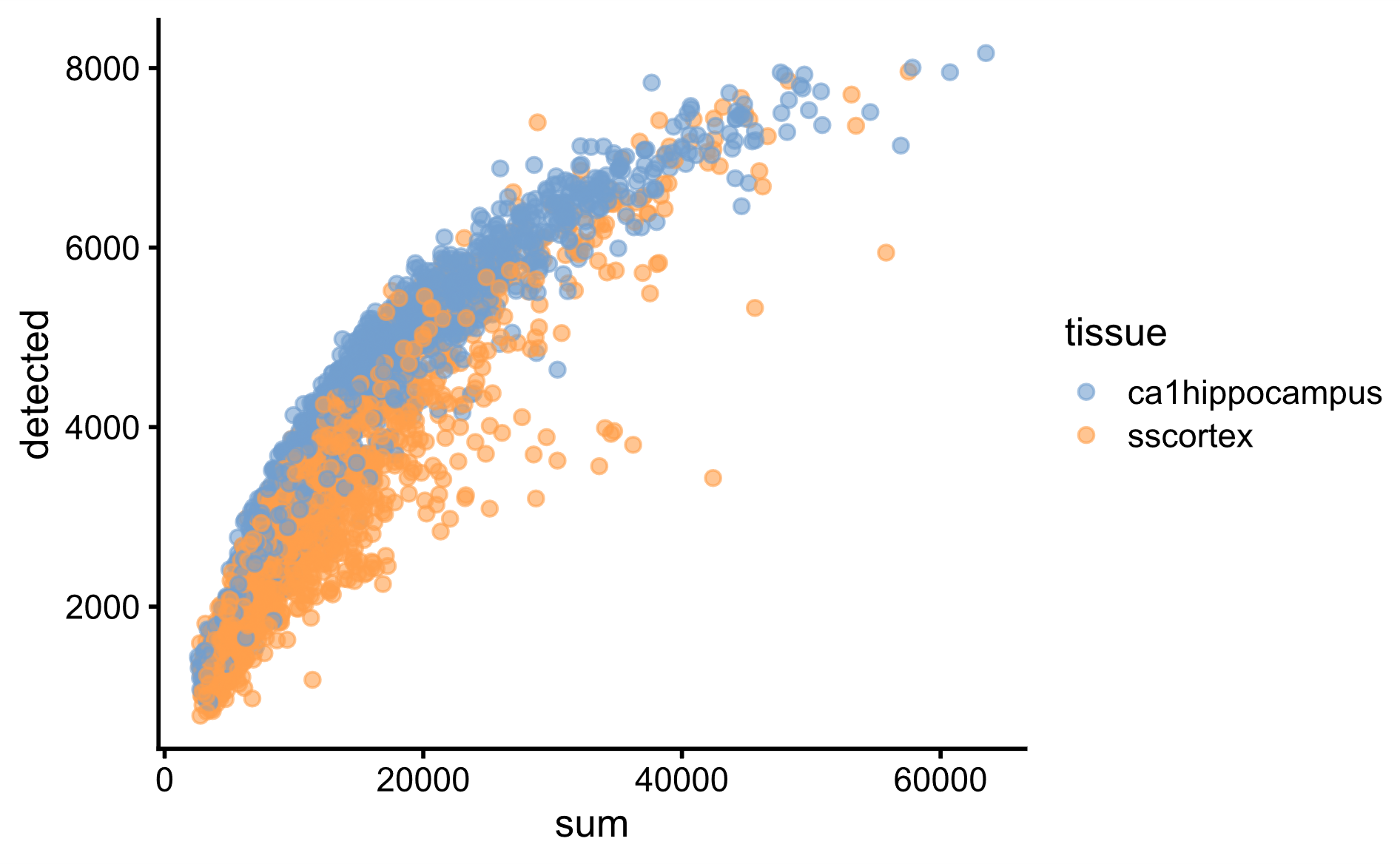
接下来,小果以上面的信息列为基础,接下来用scater的可视化功能展示count数和检测到基因数的关系,蓝色和橙色分别代表tissue下的ca1hippocampus和sscortex信息列。
plotColData(data, x = “sum”, y=”detected”, colour_by=”tissue”)
D=====( ̄▽ ̄*)b今天的学习课程就到这里哦~~
以上就是小果对scater包的简单介绍,你学会它的基本操作了嘛?scater下还有很多中可视化的领域等着我们探索,让我们继续研究下去吧!今天的小知识对你的学习是否有用捏?如果有用记得继续关注小果奥~~