用MEGA构建进化树,不会还有人不会吧!(二)
上次小果带大家在数据库下载了fasta序列并将其导入MEGA中进行序列比对,结果输出后保存了mas文件,本期我们接上期继续:
3.筛选模型。
双击保存的mas文件,点击Data➡Phylogenetic Analysi
回到主界面➡点击MODELS➡Find Best DNA/Protein Models(ML)…
这一步是为了寻找用来构建进化树最合适的模型,默认参数即可。
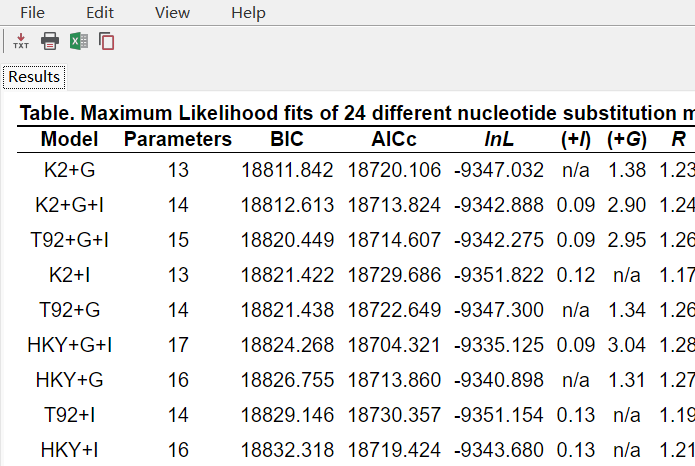
结束后会弹出一个界面,在BIC这一列中给出了模型得分,越靠前的越好,第一个是K2+G,但是软件不支持组合模型,所以选择排名最靠前的单个模型即可,这里小果选择了K2.
4.构建进化树
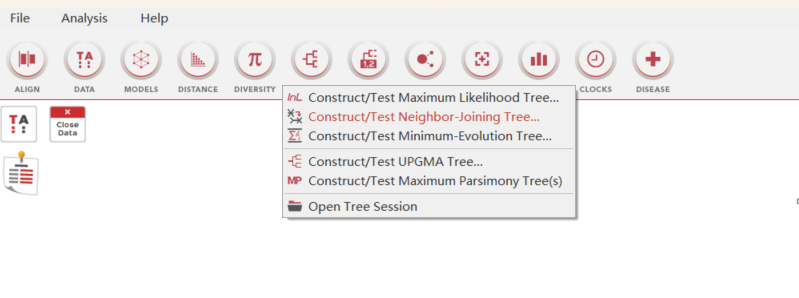
在主界面点击PHYLOGENY(树状图标)➡选第二个NJ邻接法建树
*最大似然法 (Maximum Likelihood) 和邻接法 (Neighbor-Joining) 是两种常见的建树方法两种方法之间的主要区别在于,最大似然法需要计算所有可能的树形状,因此对于大型数据集来说计算量很大,并且可能会受到计算资源的限制。而邻接法则是一种启发式方法,能够处理更大的数据集,但它没有考虑所有可能的树形状,可能会得到不同的树形状,取决于其启发式算法的选择和特定数据集的属性。总的来说,最大似然法对于小型和中型数据集是一个更准确的方法,而邻接法则则适用于更大的数据集。当然,你可以根据自己的偏好和分析目的选择建树方法。 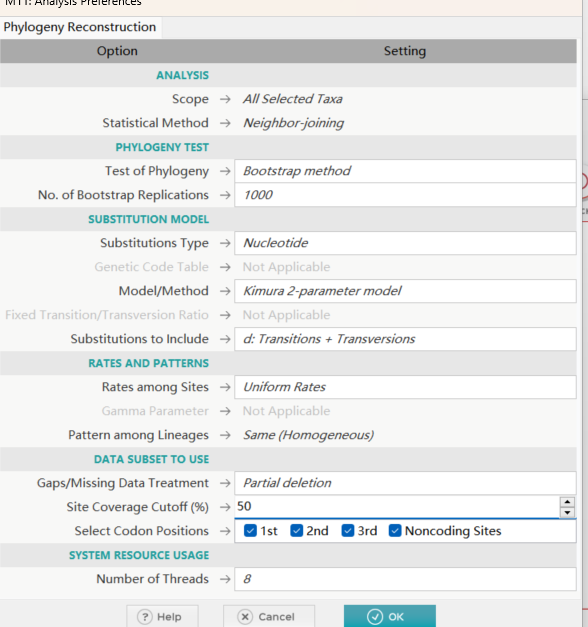
在Test of Phylogeny中选择bootstrap(bootstrap是一种常用的重复抽样方法,用于评估构建的进化树的可靠性和统计显著性)我们在步长检验次数选择1000次(默认500)。
Model选择筛选出的模型即可,这里用到刚刚选择的K2
在Gaps/MissingData Treatment处选择Partial deletion,Site CoverageCutoff选择50。
*在分子序列中存在一些缺失的数据点或空缺的位置。这种缺失数据可能会对系统发育分析结果产生影响,因此需要对缺失数据进行处理。Partial deletion是其中一种方法,它将含有缺失数据的序列删除,但保留在其它序列中完整的数据点。Site Coverage Cutoff是指在Partial deletion方法中,允许保留的数据点的最小比例或阈值。例如,选择50表示只有在某个数据点上的数据在至少50%的样本中都有观测值时,才会将该数据点纳入分析。这个阈值的选择会影响结果的可靠性和准确性,较低的阈值可能会导致较多的噪音和偏差,较高的阈值可能会削减掉太多的数据点,可能会影响结果的精度和有效性。
点击OK,稍等片刻树就画好了。
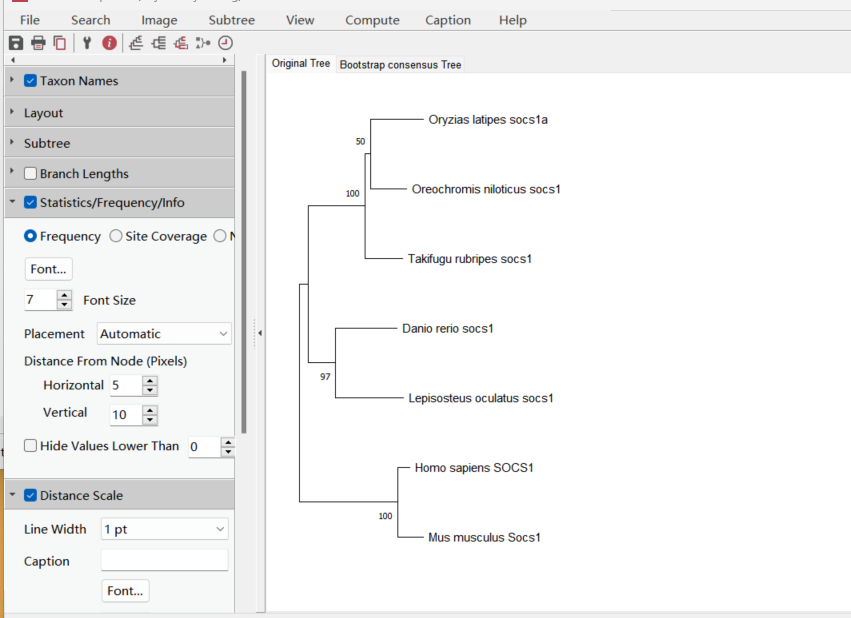
MEGA可以对进化树进行可视化和编辑。可以添加标签、改变颜色和样式等。最终的进化树可以导出为多种格式,如PDF、PNG、SVG等。
以上就是基于MEGA软件构建进化树的详细步骤,怎么样,是不是很简单呢?
后记:
如果有绘制进化树的需求,小果强烈安利本公司的云生信平台,打开链接,在云生信·迎新春一栏找到“进化树”,动动手指直接上传自己的数据就出图啦!下图是用平台示例数据做的图哟~
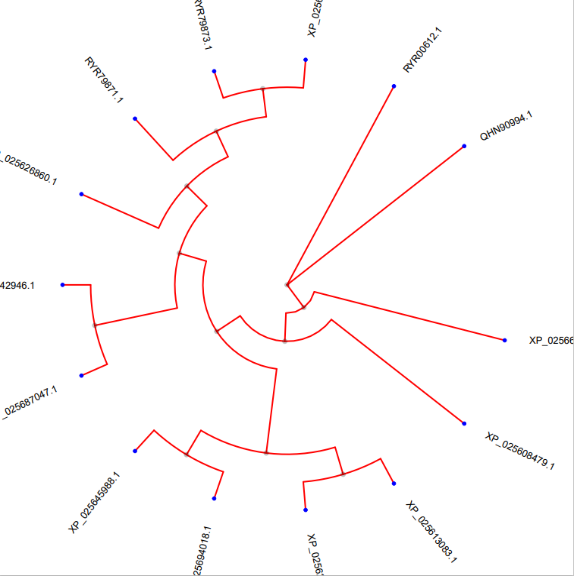
怎么样是不是很简单呢!快来注册账号吧~