揭开肿瘤免疫分析的神秘面纱:使用CIBERSORT分析你的免疫细胞!
今天,小果将给大家介绍一个基于反卷积分析的算法及其对应的R包。
在本教程中,我们将使用 R 语言中的 CIBERSORT 库对基因表达数据进行免疫细胞类型的分析,并使用 ggplot2 库对分析结果进行可视化。那就和小果一起来看一下吧!
初识CiberSort
在学习怎么使用之前,小果来给大家简单的介绍一下什么是CIBERSORT。
CIBERSORT是一种广泛应用于肿瘤免疫学研究的计算方法,它能够利用基因表达数据对肿瘤组织中不同免疫细胞亚群的比例进行定量分析。CIBERSORT基于一个线性回归模型,该模型通过对已知免疫细胞特征基因表达谱的训练来估计混杂细胞样本中各个免疫细胞亚群的相对丰度。
好了,接下来我们一起来看一下怎么使用它吧!
步骤 1: 准备工作
在开始分析之前,我们需要准备所需的工具和数据。具体来说,我们需要以下内容:
- 安装以下 R 库:ggplot2, ggpubr, reshape2, dplyr, CIBERSORT
- 下载 LM22.txt数据集 和 TCGA deg_fpkm.tsv 数据文件
install.packages(c(“ggplot2”, “ggpubr”, “reshape2”, “dplyr”, “CIBERSORT”))
步骤 2: 运行 CIBERSORT 进行免疫细胞类型分析
首先,我们将使用 CIBERSORT 库对基因表达数据进行免疫细胞类型的分析。具体来说,我们将从 LM22.txt 和 TCGA deg_fpkm.tsv 文件中读取基因表达数据,并使用 CIBERSORT 函数计算出各种免疫细胞类型的比例。请按照以下步骤进行操作:
- 将 LM22.txt 和 TCGA deg_fpkm.tsv 文件放在工作目录中。
- 输入以下代码,导入所需的库:
library(CIBERSORT)
- 输入以下代码,读取基因表达数据并运行 CIBERSORT 函数:
lm22.file <- “LM22.txt”
rawdata_file.file <- “TCGA deg_fpkm.tsv”
cibersort_file <- “TCGA cibersort.txt”
TCGA_TME.results <- CIBERSORT(lm22.file, rawdata_file.file, perm = 50, QN = F)
其中,lm22.file 和 rawdata_file.file 分别为 LM22.txt 和 TCGA deg_fpkm.tsv 文件的文件名,cibersort_file 为输出文件名,perm 表示重复运行的次数,QN 表示是否进行量化归一化。小果提醒:运行时间可能较长,具体取决于数据集的大小和计算机的性能哦。
- 输入以下代码,将 CIBERSORT 计算结果输出到文件中:
write.csv(TCGA_TME.results, “TCGA_CIBERSORT_Results_fromRcode.csv”)
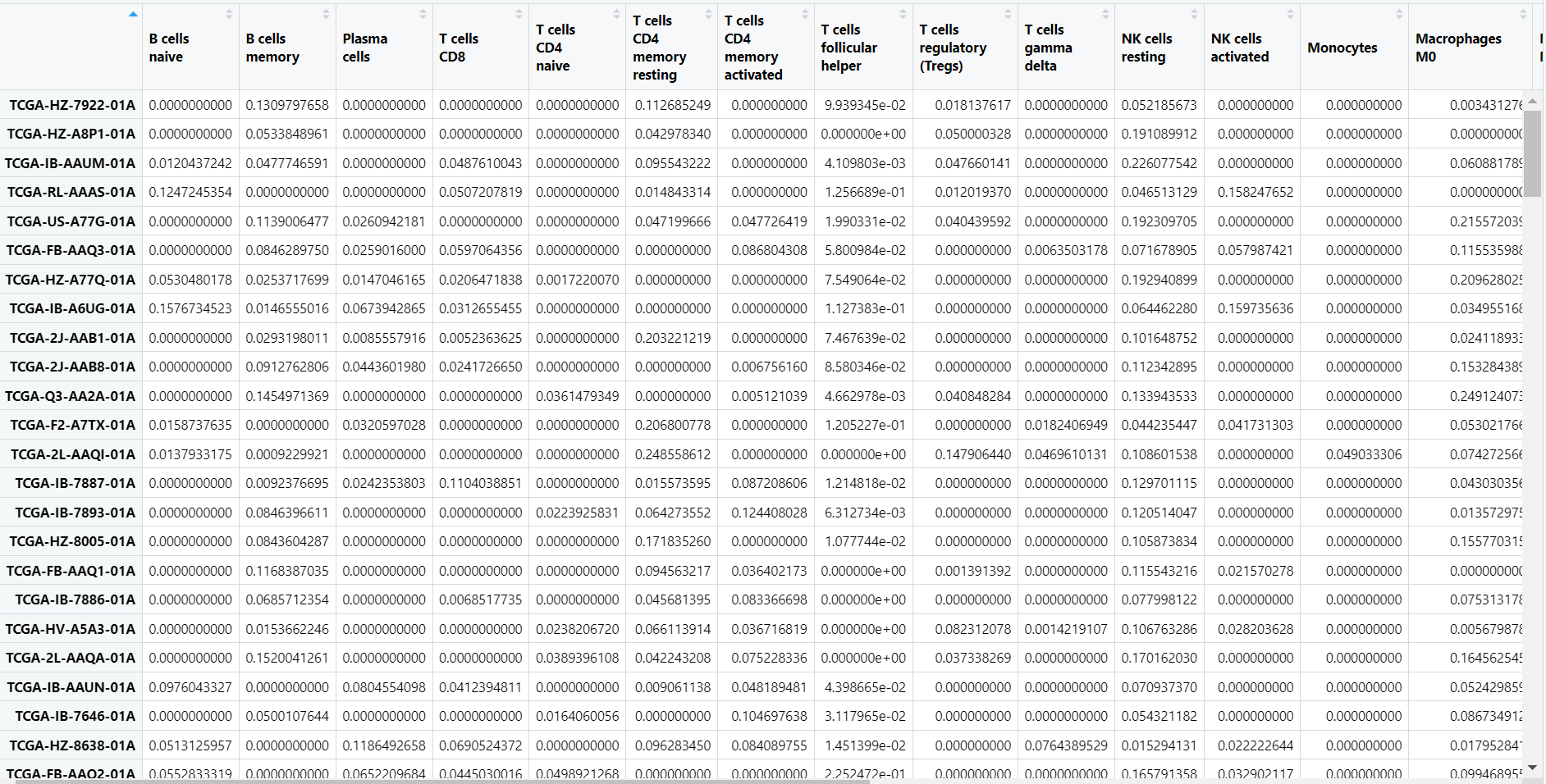
接下来,小果带大家一起看一下最后生成的结果数据是什么样的吧!
现在,你已经成功完成了免疫细胞类型分析哦!怎么样,是不是很简单呢!
怎么样,今天的教程你学废了嘛?小果专属云生信平台:云生信 – 学生物信息学 (biocloudservice.com)
云生信平台也有免疫专版的学习模块哦,快来找到你想学习的专属模块吧!