PCA:你的单细胞数据主成分分析大师
在处理单细胞测序数据的过程中,如何对数据矩阵进行降维是我们“必经”的步骤之一,那么你知道最常用的数据降维方法有哪些嘛?接下来小果将教大家最常用的数据降维方法,一起来和小果看看吧!
- 数据集准备
library(scRNAseq)
data(fluidigm)
fluidigm<-ReprocessedFluidigmData()
ct <- floor(assays(fluidigm)$rsem_counts) #生成数据矩阵
ct[1:4,1:4]
sample_ann <- as.data.frame(colData(fluidigm))
DT::datatable(sample_ann)
View(ct)
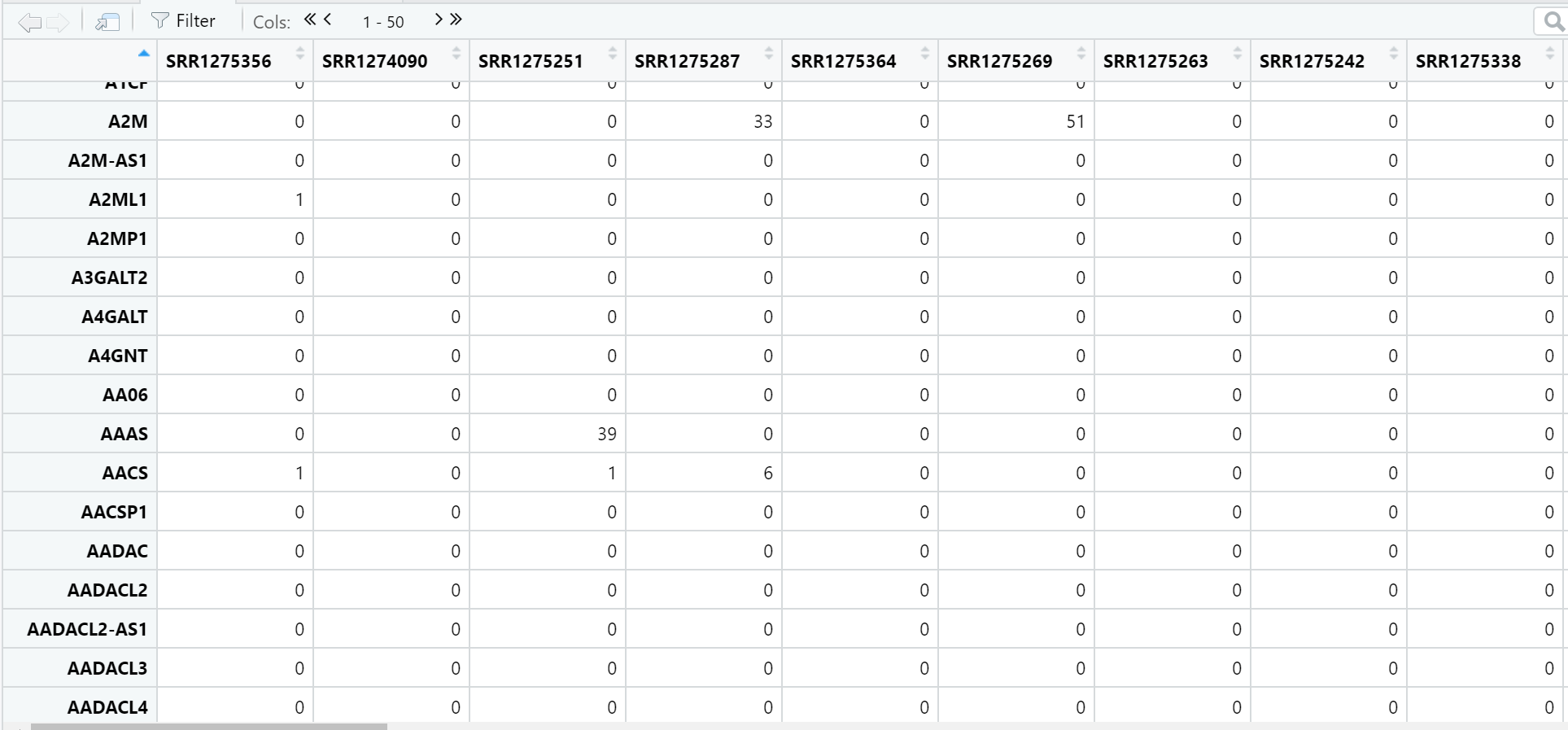 还是老规矩,和小果简单看一下我们生成的数据矩阵吧!
还是老规矩,和小果简单看一下我们生成的数据矩阵吧!
- 数据处理
因为本篇教程主要针对数据降维的方法,小果在这里就不细细阐述中间对数据处理的过程啦!小果把数据处理的代码贴在下面供同鞋们参考,包括数据过滤、数据矫正。
#数据过滤
chose_gene= apply(counts,1,function(x) sum(x>0) )>0
table(chose_gene)
counts <- counts[chose_gene,]
#数据矫正
data <- log2(edgeR::cpm(counts) +1)
- 数据降维处理
- 数据降维有哪些方法?
在进行具体操作之前,小果先向大家科普一些常用的数据降维方法吧!
1.PCA(主成分分析法)
2.MDS(多维缩放)
3.t-SNE
4.LLE(局部线嵌入)
-
- 什么是PCA主成分分析?
PCA(Principal Components Analysis)。其实就是找出处理数据中一个最主要的特征,然后进行深入分析。小果来给大家举个简单的例子理解一下:现在要考察公司中一个员工的整体能力水平,我们目前的参考因素有他的业绩、出勤时间、高考成绩、工资水平等等因素,那么我们为了更快速地做出判断,我们只要提取他地业绩来分析即可。
-
- R语言实现PCA主成分分析
install.packages(c(“FactoMineR”, “factoextra”)) #下载PCA()函数所需导入的R包
#PCA降维
dat <- data_back #导入上面处理后的矩阵
dat <- t(dat)
dat <- as.data.frame(dat) #生成表格
plate <- sample_ann$Biological_Condition # 定义分组信息
dat <- cbind(dat,plate) #合并列
dat[1:4,1:4]
table(dat$plate) #分组信息展示
 现在展示的是按照Biological_Condition对数据矩阵分组后的统计信息哦!
现在展示的是按照Biological_Condition对数据矩阵分组后的统计信息哦!
library(FactoMineR)
library(factoextra)
dat.pca <- PCA(dat[,-ncol(dat)],graph = FALSE) #PCA降维处理
head(dat.pca$ind$coord) #每个细胞的前5个主成分取值
 PCA函数降维后,大家和小果先一起来看下每个细胞中前5个主成分的分值吧!
PCA函数降维后,大家和小果先一起来看下每个细胞中前5个主成分的分值吧!
得到了以上的分析结果,我们如何将PCA分析后的结果可视化展示呢,和小果一起来学一下吧!
 #PCA降维可视化
#PCA降维可视化
fviz_pca_ind(
dat.pca,
geom.ind = “point”,
col.ind = dat$plate,
addEllipses = TRUE,
legend.title = “Groups”
)
好啦,还有更多的数据降维方法等着大家去学习和使用,同学们可以根据自己的情况自行选择去了解,在这里大家也可以移步我们的云生信平台的画图专版去深入学习哦!
小果云生信平台:云生信 – 学生物信息学 (biocloudservice.com)
小果云生信平台画图专版:
云生信pca图绘制小工具:www.biocloudservice.com/443/443.php
今天小果教你的降维方法学会了吗!更多生信小技巧请继续关注小果吧!~